なんの記事?
Entity Linking チュートリアル 前編 ざっくりとした歴史編 に続く中編です。実際に手を動かしてエンティティ・リンキングのデータセットのパース・モデルの訓練と予測を行っていきたいと思います。
今回作るもの
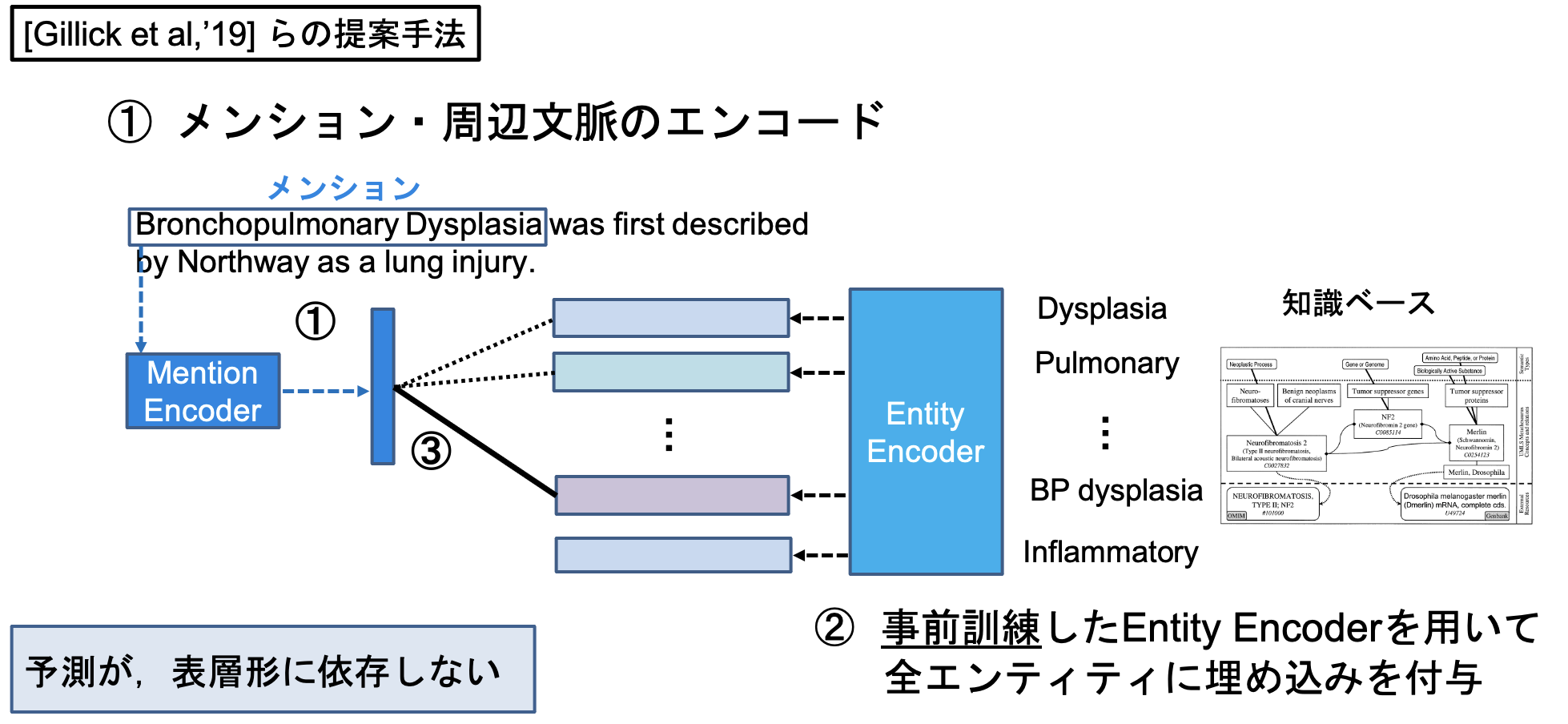
Bi-encoder ベースのエンティティ・リンキングを実装していきます。 Bi-encoderベースのエンティティリンキングシステムは [Gillick et al., '19] が初出と考えられ、現在はそれのTransformerベースが主流になっています([Wu et al., '20])。
Bi-encoderベースのエンティティ・リンキングでは、メンション側とエンティティ側でそれぞれ独立にベクトルをエンコーディングし、ベクトル同士の近さ(L2距離、コサイン類似度、内積など)を用いてメンションと知識ベース内エンティティを比較し、メンションに紐づくであろうエンティティを予測します。
今回このシステムの前処理部分を作っていきたいと思います。
また、今回のシステムでは事前にある程度候補の絞り込みを行った上で、エンティティ・リンギングを行っていきます。
Gillickらの手法では近似近傍検索を用いて、メンションに紐づくであろうエンティティを予測していますが、今回の実装では予めメンションに対して生成された候補のみを対象として、Bi-encoderモデルによる予測を行っていきます。
ですので、今回実装するモデルは以下になります。
使用するフレームワーク・ライブラリなど
適宜最終的にはDockerfile内に記述するので、ここでは最小限に留めます。
深層学習のフレームワークとしてはPyTorchを使用し、前処理や実験についてはAllenNLPを使用します。また、今回扱う BC5CDRデータセットの前処理に、自作の前処理ツールを使用します。
データセット
エンティティ・リンキングは文書内に存在する、エンティティと呼ばれる文字列スパンに対して、知識ベース内のエンティティを対応させるタスクです。(前回の絵を再掲します。)

メンションを含む文書群としては、今回はBC5CDR を用います。元の論文に記されているように、紐付け先の知識ベースには生命科学用語集(Medical Subject Headings: MeSH) を利用します。
BC5CDR データセット
BC5CDRは、BioCreative Vの化学物質と疾患の言及認識タスクのために作成されたデータセットです。1500件の論文から構成されており、15935件の化学物質と12852件の疾患に関する言及が含まれています。
ほぼすべてのメンションに対してMeSH内のゴールドエンティティがアノテーションされています。
MeSH

(画像はホームページより)
今回紐付ける知識ベースです。約3万語のエンティティについて、主に生命医学ドメインのものを階層構造と共に格納しています。MeSH自体は毎年更新されています。
前処理
では実際に前処理を行っていきます。
データセットの前処理
大変手間が掛かるので前処理ツールを用意しました。
BC5CDR データセット
$ git clone https://github.com/izuna385/PubTator-Multiprocess-Parser.git
$ cd PubTator-Multiprocess-Parser
$ docker build -t multiprocess_pubtator .
$ docker run -it multiprocess_pubtator /bin/bash
コンテナ内に移った後 sh ./scripts/quick_start_BC5CDR_full.sh により前処理が走ります。
fullを実行する場合スペックによっては相当の時間が掛かるので、まず動かすだけ!という方はsh ./scripts/quick_start_BC5CDR_sample.shを実行してください。
手元のMac(8コア)だと、約5分程度で終わりました。
./preprocessed_doc_dir 下に、前処理の終了した論文(タイトル+アブスト+アノテーション)が ***.json
として保存されています。これらのファイルと、./dataset/ディレクトリを後で使用するので、コンテナの外に持ち出します。
# コンテナの外に出る
$ exit
# コンテナ外
$ docker cp flamboyant_tesla:/projects/dataset .
$ docker cp flamboyant_tesla:/projects/preprocessed_doc_dir .
この時点で、docker を出た状態でディレクトリ構成は以下のようになっています。

datasetディレクトリとpreprocessed_doc_dirも確認しておきましょう。


MeSH
ここまでで、実際にEntity Linkingタスクの学習と評価に使用する、アノテーションされたデータセット側の前処理が終わりました。
ここでは、BC5CDRデータセットの紐付け先の知識ベース、MeSHを用いて、各メンションに対する表層ベースでの候補を生成します。
from tqdm import tqdm
from scispacy.candidate_generation import CandidateGenerator
MeshCandidateGenrator = CandidateGenerator(name='mesh')
KB=MeshCandidateGenrator.kb
K=100
Resolve_abbreviations = True
Threshold = 0.3
No_definition_threshold = 0.95
Filter_for_definitions = True
Max_entities_per_mention = 30
def candidate_dui_generator(mention_strings):
batch_candidates = MeshCandidateGenrator(mention_strings, K)
batched_sorted_candidates = list()
for candidates in batch_candidates:
predicted = []
for cand in candidates:
score = max(cand.similarities)
if (
Filter_for_definitions
and KB.cui_to_entity[cand.concept_id].definition is None
and score < No_definition_threshold
):
continue
if score > Threshold:
predicted.append((cand.concept_id, score))
sorted_predicted = sorted(predicted, reverse=True, key=lambda x: x[1])
sorted_predicted = sorted_predicted[: Max_entities_per_mention]
batched_sorted_candidates.append(sorted_predicted)
return batched_sorted_candidates
def batcher(iterable, n=1):
l = len(iterable)
for ndx in range(0, l, n):
yield iterable[ndx:min(ndx + n, l)]
ScispaCy内のソースを一部用いた候補生成をここでは行っています。このスクリプトは sh ./scripts/quick_start_BC5CDR_full.sh 内に含まれています。
candidates.pkl が各メンションに対する候補のエンティティIDを格納するファイルになります。こちらもコンテナの外に出しておきましょう。
AllenNLP を用いた実験準備
ここまでで必要なデータセットの前処理を完了することが出来ました。しかし、ただ単純にPyTorchのみを用いて実験を行う場合、パディングやBERTの埋め込み付与・fine-tuning等には膨大な手間が掛かります。
そこで、今回のチュートリアルではAllenNLPを用いてこれまでに用意したデータセットを、実験用に変換したいと思います。
Local Model と Global Model
詳しくはこちらに書かれており、本記事では詳細な説明は省略します。AllenNLPでは各データはInstanceオブジェクトとして管理され、本記事で扱うコードでもそれに倣っています。
また、Instance オブジェクトとして今回は各メンションを扱います。各メンションとそのメンションが含まれる1文章、メンションに対するゴールドエンティティと、ゴールドエンティティが持つ情報を一つのInstanceとして格納します。
後述になってしまいましたが、本記事ではエンティティ・リンキングの Local model を扱います。エンティティ・リンキングにおける Local model ならびに Global model についての説明は [Cao et al., '18] などが詳しいです。
下に簡単な概念図を載せます。
Local Modelでは、各メンションごとに、そのメンションに紐付けるべき最適なエンティティを探索します。言い換えれば、1つのメンションのみに注目して、知識ベース内のエンティティ内において最も近しいエンティティを探索します。(図ではその近さをψとしています。)
Global Modelでは、各メンションのスコアψに加え、メンションが含まれるドキュメント全体での一貫性を定義する関数Φの最適化まで考えます。ざっくり言えば、文書全体が何らかのトピックや話題について言及しており、そのトピックや話題がドキュメント全体で保たれている、という前提を置きます。
BERTの登場以前まではGlobal Modelも少なからずありましたので、表にまとめます。ただし現在では、Bi-encoderの特性上、Local Modelが主流となっています。
本チュートリアル用のソースコード
本セクションからは、以下のチュートリアル用リポジトリを使用します。
DatasetReaderの実装
今回のBC5CDRデータセットは、Local Modelで実装します。つまり、各メンションをそれぞれ独立に扱います。(なので、メンションが含まれていたアブストの情報は、メンションを含む1文のみに限定されることになり、それ以外は無視されることになります。)
read メソッドのオーバーライド
Datasetクラスを継承し、_read メソッドをオーバーライドすることにより、本タスク用にカスタムしたDatasetReaderの作成が可能になります。
import tempfile
from typing import Dict, Iterable
from overrides import overrides
from commons import CANONICAL_AND_DEF_CONNECTTOKEN, MENTION_START_TOKEN, MENTION_END_TOKEN
import torch
from allennlp.data import Instance
from allennlp.data.dataset_readers import DatasetReader
from allennlp.data.fields import SpanField, ListField, TextField, MetadataField, ArrayField, SequenceLabelField, LabelField
from allennlp.data.fields import LabelField, TextField
from allennlp.data.tokenizers import Token, Tokenizer, WhitespaceTokenizer
from parameteres import Biencoder_params
import glob
import os
import random
import pdb
from tqdm import tqdm
import json
from tokenizer import CustomTokenizer
import numpy as np
class BC5CDRReader(DatasetReader):
def __init__(
self,
config,
max_tokens: int = None,
**kwargs
):
super().__init__(**kwargs)
self.custom_tokenizer_class = CustomTokenizer(config=config)
self.token_indexers = self.custom_tokenizer_class.token_indexer_returner()
self.max_tokens = max_tokens
self.config = config
self.train_pmids, self.dev_pmids, self.test_pmids = self._train_dev_test_pmid_returner()
self.id2mention, self.train_mention_ids, self.dev_mention_ids, self.test_mention_ids = \
self._mention_id_returner(self.train_pmids, self.dev_pmids, self.test_pmids)
# kb loading
self.dui2idx, self.idx2dui, self.dui2canonical, self.dui2definition = self._kb_loader()
@overrides
def _read(self, train_dev_test_flag: str) -> List:
'''
:param train_dev_test_flag: 'train', 'dev', 'test'
:return:
'''
mention_ids, instances = list(), list()
if train_dev_test_flag == 'train':
mention_ids += self.train_mention_ids
# Because Iterator(shuffle=True) has bug, we forcefully shuffle train dataset here.
random.shuffle(mention_ids)
elif train_dev_test_flag == 'dev':
mention_ids += self.dev_mention_ids
elif train_dev_test_flag == 'test':
mention_ids += self.test_mention_ids
elif train_dev_test_flag == 'train_and_dev':
mention_ids += self.train_mention_ids
mention_ids += self.dev_mention_ids
if self.config.debug:
mention_ids = mention_ids[:50]
for idx, mention_uniq_id in tqdm(enumerate(mention_ids)):
try:
data = self._one_line_parser(mention_uniq_id=mention_uniq_id)
instances.append(self.text_to_instance(data=data))
# yield self.text_to_instance(data=data)
except:
print(mention_uniq_id, self.id2mention[mention_uniq_id])
print('Warning. This CUI is not included in MeSH.')
return instances
今回はBERTあるいはBioBERT([Lee et al., '20])を用いるため、それ用の CustomTokenizer も作製しています。このCustomTokenizerで定義したトークナイザを用いて、各メンションを_one_line_parser にてトークナイズし、_text_to_instance によって Instance オブジェクトに変換しています。
細かい前処理については こちらのコード を見て下さい。
トークナイズと Instance オブジェクトへの格納
トークナイズについては、transformers 内 BertTokenizer を使用します。今回のデータセットがメンションの目印(<target>,</target>)を含むので、そのトークンのみ恣意的にトークナイズしないようにしてあります。
# tokenizer.py
def bert_tokenizer_returner(self):
if self.config.bert_name == 'bert-base-uncased':
vocab_file = './vocab_file/bert-base-uncased-vocab.txt'
do_lower_case = True
return transformers.BertTokenizer(vocab_file=vocab_file,
do_lower_case=do_lower_case,
do_basic_tokenize=True,
never_split=['<target>', '</target>'])
elif self.config.bert_name == 'biobert':
vocab_file = './vocab_file/biobert_v1.1_pubmed_vocab.txt'
do_lower_case = False
return transformers.BertTokenizer(vocab_file=vocab_file,
do_lower_case=do_lower_case,
do_basic_tokenize=True,
never_split=['<target>', '</target>'])
else:
print('currently not supported:', self.config.bert_name)
raise NotImplementedError
このトークナイザを用いてメンションやメンションを含む文、また知識ベース内エンティティの実体名(canonical name)とその説明文(definition)をトークナイズし、インスタンスに情報を格納します。
データセットの呼び出し
_read メソッドにより、実際にデータセットを呼び出します。
# Loading Datasets
train, dev, test, train_and_dev = reader._read('train'), reader._read('dev'), reader._read('test'), \
reader._read('train_and_dev')
vocab = build_vocab(train_and_dev)
train_loader, dev_loader = build_data_loaders(params, train, dev)
train_loader.index_with(vocab)
dev_loader.index_with(vocab)
MeSHの前処理とファイル配置
MeSH内のエンティティへのインデキシングを行うために、preprocess_mesh.py を実行しましょう。
また、最初の前処理で得られたcandidates.pkl 及び preprocessed_doc_dir/ を以下のように配置しておきましょう。
最終的なファイル配置は以下のようになります。
まとめ
今回はEntity Linkingタスク用のデータセット、BC5CDRおよびその紐付け先知識ベースのMeSHを用いた候補生成の前処理を行いました。
次回は、Bi-encoderベースのエンティティリンキングモデルを実装し、本データセットで実験と評価を行います。
ソースコードは以下になります。こちらは次回でも使用予定です。
次の記事