はじめに
前回の記事では、コレラマップを作成しました。
https://qiita.com/iwasaki_kenichi/items/d7b59d63efe62a3881bd
今回は、COVID-19のフリープロジェクトに参加します。
https://learn.datacamp.com/projects/870
備忘録も兼ねて、投稿したいと思います。
また、翻訳はDeepLを使います。
概要
数ヶ月のうちに、COVID-19は流行からパンデミックへと変化しました。2019年12月に最初に確認された症例から、このウイルスはどのようにしてこれほど急速に、そして広範囲に広がったのでしょうか?この無料のRプロジェクトでは、コロナウイルスの発生初期の数ヶ月間のデータを可視化して、このウイルスがどのようにして世界的なパンデミックに成長したのかを確認します。
使ったデータの可視化の中級コースで学ぶことができます。この
このプロジェクトでは、dplyrを使ってデータフレームを操作し、ggplot2を使ってプロットを作成できることを前提としています。これらのスキルはコースで学ぶことができます。dplyrを使ったデータ操作とggplot2をプロジェクトのデータセットはこちらで見ることができます。
1. 伝染病からパンデミックへ
2019年12月、中国の武漢地域でCOVID-19コロナウイルスが初めて確認されました。2020年3月11日までに、世界保健機関(WHO)はCOVID-19のアウトブレイクをパンデミックに分類しました。イラン、韓国、イタリアでの大規模なアウトブレイクとの間に、多くのことが起こりました。
COVID-19は、咳、くしゃみ、会話などの呼吸器の飛沫を介して広がることがわかっています。しかし、どのくらいの速さでウイルスは世界中に広がったのでしょうか?また、シャットダウンや検疫のような国を挙げての政策は効果があるのでしょうか?
幸いなことに、世界中の組織がデータを収集しているので、政府はこのパンデミックを監視し、そこから学ぶことができます。特に注目すべきは、ジョンズ・ホプキンス大学システム科学・工学センターが、WHO、疾病対策センター(CDC)、保健省などの複数の国からのデータを統合するために、一般公開されているデータリポジトリを作成したことです。
このノートでは、このウイルスがどの時点で世界的なパンデミックになったのかを確認するために、発生から数週間後のCOVID-19のデータを可視化します。
COVID-19に関する情報やデータは頻繁に更新されていることに注意してください。本プロジェクトで使用したデータは、2020年3月17日に引っ張り出されたものであり、利用可能な最新のデータであると考えるべきではありません。
# Load the readr, ggplot2, and dplyr packages
library(readr)
library(ggplot2)
library(dplyr)
# Read datasets/confirmed_cases_worldwide.csv into confirmed_cases_worldwide
confirmed_cases_worldwide <- read_csv("datasets/confirmed_cases_worldwide.csv")
# See the result
confirmed_cases_worldwide
結果は下記の通り
date cum_cases
<date> <dbl>
2020-01-22 555
2020-01-23 653
2020-01-24 941
2020-01-25 1434
2020-01-26 2118
2020-01-27 2927
2020-01-28 5578
2020-01-29 6166
2020-01-30 8234
2020-01-31 9927
2020-02-01 12038
2020-02-02 16787
2020-02-03 19881
2020-02-04 23892
2020-02-05 27635
2020-02-06 30817
2020-02-07 34391
2020-02-08 37120
2020-02-09 40150
2020-02-10 42762
2020-02-11 44802
2020-02-12 45221
2020-02-13 60368
2020-02-14 66885
2020-02-15 69030
2020-02-16 71224
2020-02-17 73258
2020-02-18 75136
2020-02-19 75639
2020-02-20 76197
2020-02-21 76823
2020-02-22 78579
2020-02-23 78965
2020-02-24 79568
2020-02-25 80413
2020-02-26 81395
2020-02-27 82754
2020-02-28 84120
2020-02-29 86011
2020-03-01 88369
2020-03-02 90306
2020-03-03 92840
2020-03-04 95120
2020-03-05 97882
2020-03-06 101784
2020-03-07 105821
2020-03-08 109795
2020-03-09 113561
2020-03-10 118592
2020-03-11 125865
2020-03-12 128343
2020-03-13 145193
2020-03-14 156097
2020-03-15 167449
2020-03-16 181531
2020-03-17 197146
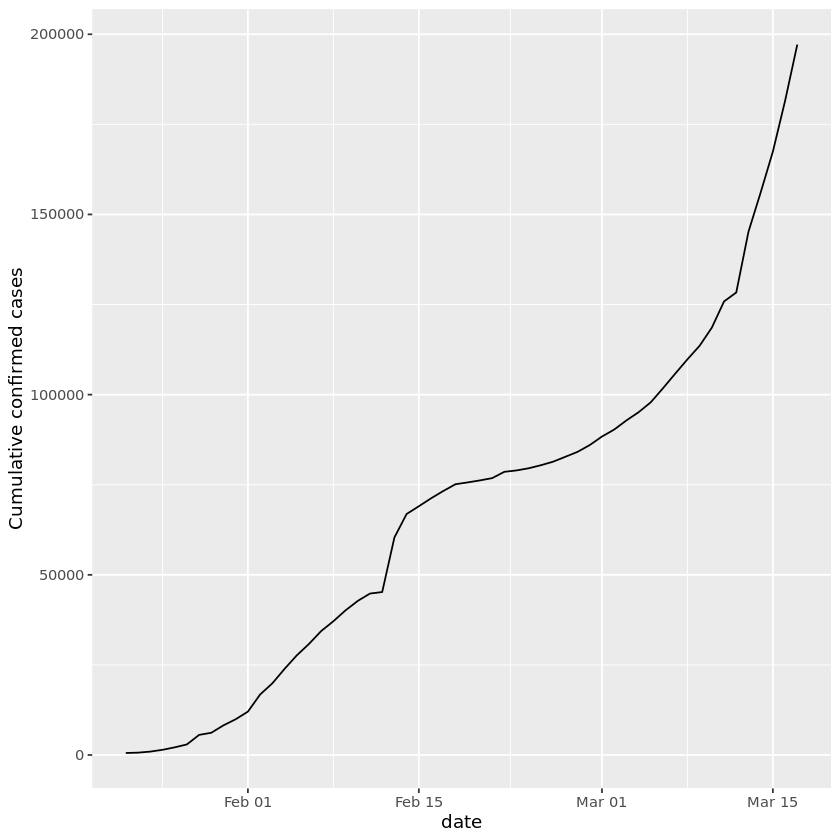
2. 世界中で確認されている症例
上の表は、全世界で確認されたCOVID-19の累積症例数を日付別に示したものです。表の中の数字を読むだけでは、発生の規模や規模の拡大を実感することができません。世界の確定症例を可視化するために、折れ線グラフを描いてみましょう。
# Draw a line plot of cumulative cases vs. date
# Label the y-axis
ggplot(confirmed_cases_worldwide,aes(x = date,y = cum_cases)) +
geom_line()+
ylab("Cumulative confirmed cases")
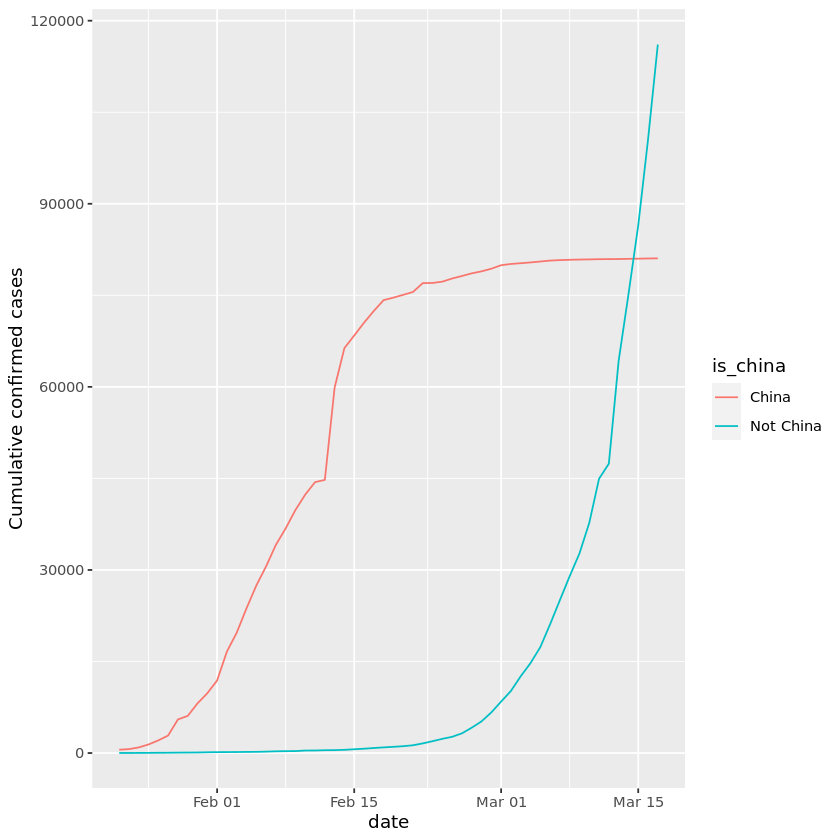
3. 世界と比較した中国
このプロットのY軸はかなり恐ろしいもので、世界中で確認された症例の総数は20万人に近づいています。それ以外にも、奇妙な事が起きています。2月中旬に奇妙なジャンプがあり、その後、しばらくの間、新規症例の発生率が低下し、3月には再びスピードアップします。何が起きているのか、もっと詳しく調べる必要があります。
発生初期、COVID-19の症例は主に中国に集中していました。中国とその他の地域で確認された COVID-19の症例を 別々にプロットしてみましょう 洞察力があるかどうか見てみましょう
今後の課題でこのプロットを構築していきます 次のタスクで重要になることの一つは、グローバルな美学を作るのではなく、GGPLOTの線のジオメトリの中に美学を追加することです。
# Read in datasets/confirmed_cases_china_vs_world.csv
confirmed_cases_china_vs_world <- read_csv("datasets/confirmed_cases_china_vs_world.csv")
# See the result
confirmed_cases_china_vs_world
# Draw a line plot of cumulative cases vs. date, grouped and colored by is_china
# Define aesthetics within the line geom
plt_cum_confirmed_cases_china_vs_world <- ggplot(confirmed_cases_china_vs_world) +
geom_line(aes(x = date,y = cum_cases,group = is_china,color = is_china)) +
ylab("Cumulative confirmed cases")
# See the plot
plt_cum_confirmed_cases_china_vs_world
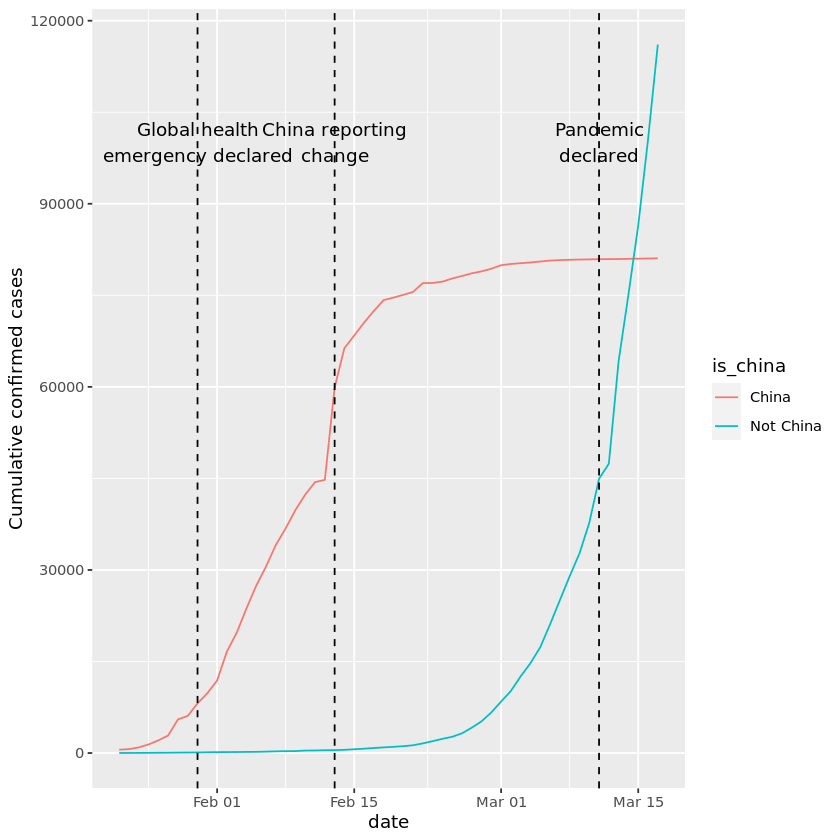
4. 注釈をつけよう!
うわぁー!!!(笑)。2本の線の形が全然違いますね。2月は中国での発生が多かったのですが、3月になって本格的に世界的な流行になりました。それが3月になって、世界的な流行になったのです。3月14日頃、中国国外の患者数が中国国内の患者数を上回ったのです。これはWHOがパンデミックを宣言した数日後のことでした。
この大流行の間には、他にもいくつかの画期的な出来事がありました。例えば、2020年2月13日の中国線の大ジャンプは、アウトブレイクに関して単に悪い日だっただけではありませんでした。
このような出来事に注釈をつけることで、プロットの変化をよりよく解釈することができます。
who_events <- tribble(
~ date, ~ event,
"2020-01-30", "Global health\nemergency declared",
"2020-03-11", "Pandemic\ndeclared",
"2020-02-13", "China reporting\nchange"
) %>%
mutate(date = as.Date(date))
# Using who_events, add vertical dashed lines with an xintercept at date
# and text at date, labeled by event, and at 100000 on the y-axis
plt_cum_confirmed_cases_china_vs_world +
geom_vline(aes(xintercept = date),data = who_events , linetype = "dashed") +
geom_text(aes(x = date, label =event), data =who_events, y = 1e5)
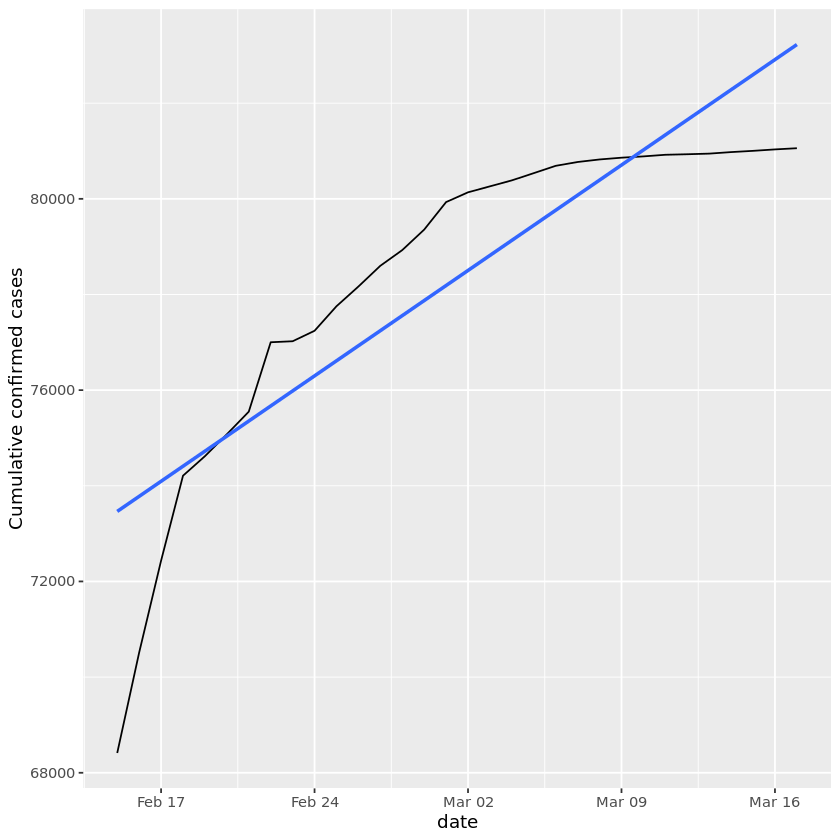
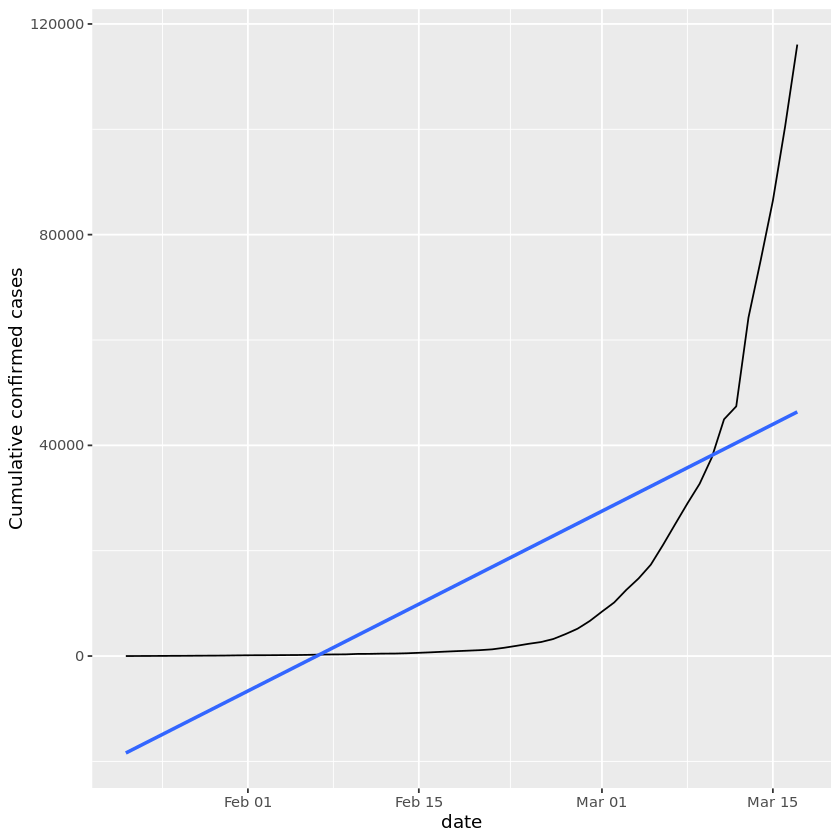
5. 中国にトレンドラインを追加
将来の問題がどれだけ大きなものになるかを評価しようとするときには、ケースの数がどれくらいの速さで増えているかを測る必要があります。良い出発点は、症例数が直線的に増加しているのではなく、速く増加しているのか、遅くなっているのかを見ることです。
2020年2月13日の中国での報告変更前後には、明らかに症例数が急増しています。しかし、その数日後には中国での症例の伸びが鈍化します。2020年2月15日以降の中国でのCOVID-19の成長をどのように表現すればよいのでしょうか。
# Filter for China, from Feb 15
china_after_feb15 <- confirmed_cases_china_vs_world %>%
filter(is_china == "China" , date >= "2020-02-15")
# Using china_after_feb15, draw a line plot cum_cases vs. date
# Add a smooth trend line using linear regression, no error bars
ggplot(china_after_feb15,aes(x = date, y = cum_cases)) +
geom_line() +
geom_smooth(method = "lm",se = FALSE) +
ylab("Cumulative confirmed cases")
6. 世界の他の地域は?
上のプロットを見る限り、中国の成長率は線形よりも遅くなっています。これは、中国が2月下旬と3月上旬に少なくともある程度ウイルスを封じ込めたことを示しているので、素晴らしいニュースです。
世界の他の地域は、直線的な成長と比べてどうでしょうか?
# Filter confirmed_cases_china_vs_world for not China
not_china <- confirmed_cases_china_vs_world %>%
filter(is_china!="China")
# Using not_china, draw a line plot cum_cases vs. date
# Add a smooth trend line using linear regression, no error bars
plt_not_china_trend_lin <- ggplot(not_china,aes(x = date,y = cum_cases)) +
geom_line() +
geom_smooth(method = "lm",se = FALSE) +
ylab("Cumulative confirmed cases")
# See the result
plt_not_china_trend_lin
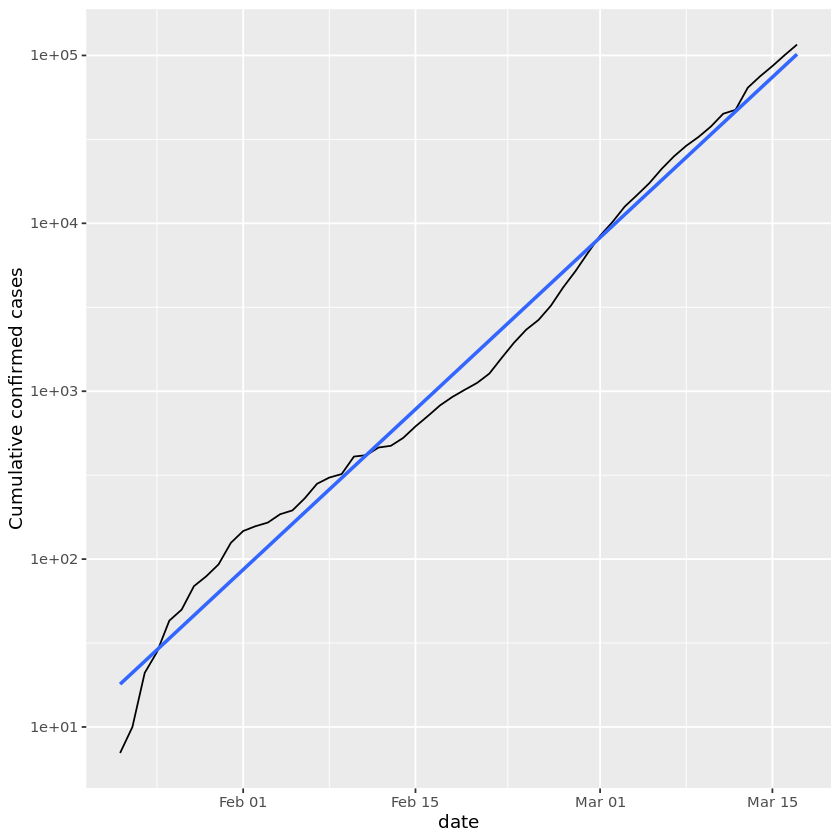
7. 対数スケールの追加
上のプロットを見ると、直線では全くフィットしないことがわかりますし、それ以外の部分は直線よりもずっと早く成長していることがわかります。Y軸に対数スケールを追加したらどうでしょうか?
# Modify the plot to use a logarithmic scale on the y-axis
plt_not_china_trend_lin +
scale_y_log10()
8. 中国以外のどの国が最も打撃を受けているか?
対数スケールを使用することで、データにはるかに近いフィット感を得ることができます。データ科学の観点からは、良いフィットは素晴らしいニュースです。残念ながら、公衆衛生の観点から見ると、世界の他の地域でCOVID-19の感染者が指数関数的な速度で増加していることを意味しますが、これは恐ろしいニュースです。
すべての国が同じようにCOVID-19の影響を受けているわけではなく、世界のどこで問題が最も大きいのかを知ることは有用であろう。我々のデータセットの中で最も症例が確認されている中国以外の国を見つけよう。
# Run this to get the data for each country
confirmed_cases_by_country <- read_csv("datasets/confirmed_cases_by_country.csv")
glimpse(confirmed_cases_by_country)
# Group by country, summarize to calculate total cases, find the top 7
top_countries_by_total_cases <- confirmed_cases_by_country %>%
group_by(country) %>%
summarize(total_cases = max(cum_cases)) %>%
top_n(7)
# See the result
top_countries_by_total_cases
country total_cases
<chr> <dbl>
France 7699
Germany 9257
Iran 16169
Italy 31506
Korea, South 8320
Spain 11748
US 6421
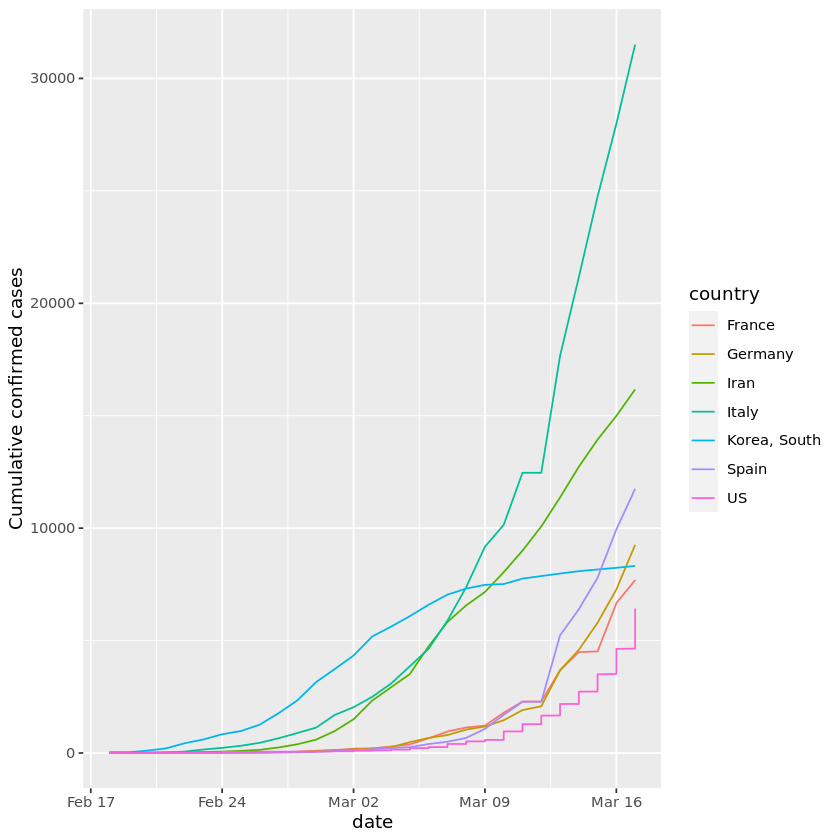
9. 2020年3月中旬時点でのハードヒット国のプロット
最初に中国で発生が確認されたにもかかわらず、上の表の東アジアの国は1カ国(韓国)のみである。このうち4カ国(フランス、ドイツ、イタリア、スペイン)はヨーロッパにあり、国境を共有しています。これらの国で確認された症例を時間経過とともにプロットすることで、より詳細な状況を把握することができます。
最後に、最後のステップに到達したことを祝福します。ビジュアライゼーションを続けたい方、または今日の時点で最もヒットした国を見つけたい方は、こちらの最新のデータを使ってご自身の分析を行うことができます。
# Run this to get the data for the top 7 countries
confirmed_cases_top7_outside_china <- read_csv("datasets/confirmed_cases_top7_outside_china.csv")
glimpse(confirmed_cases_top7_outside_china)
# Using confirmed_cases_top7_outside_china, draw a line plot of
# cum_cases vs. date, grouped and colored by country
ggplot(confirmed_cases_top7_outside_china,aes(x = date,y = cum_cases, group = country,color = country))+
geom_line()+
ylab("Cumulative confirmed cases")
おわりに
使ったことのない関数とかがあったので、少し時間がかかってしまいました。
収束したころに、もう一度このプログラムを流してみたいですね。