Kerasでハイパーパラメータの自動調整いろいろ
ディープラーニングを使う際の大きな課題の一つがハイパーパラメータのチューニングです。
ニューラルネットワークのニューロン数やドロップ率、ラーニング率といったパラメータを調整し、より良いモデルを作る必要があります。
どのパラメータ値が良いのかは先例や経験から決めることができますが、始めてのモデルを使う場合は、やはりパラメータのチューニングをしなければなりません。
ハイパーパラメータ・チューニングの方法は手動調整と自動調整があります。
手動でパラメータを試していくよりも、やはり自動調整でパラメータを探索していくほうが便利です。
パラメータ自動調整の方法
ニューラルネットワークに限りませんが、機械学習のパラメータ調整を行う方法は多種多様にあります。以下に例を挙げます。
- ランダム・サーチ RandomizedSearchCV: ランダムにパラメータを試していく。scikit-learnを使って実装。
- グリッド・サーチ GridSearchCV: パラメータの候補値を指定して、その組み合わせを全て試す。scikit-learnを使って実装。
- ラテン超方格サンプリング法 LHS: 層別サンプリング法のひとつで、各変数をn個に分割して、ランダムに値を取り出して試す。pyDOEを使って実装。参考
- ベイズ最適化 Bayesian Optimization: パラメータに対する評価関数の分布がガウス過程に従うと仮定、パラメータ値を試していくことで実際の分布を探索することで、より良いパラメータ値を得る。GpyOptで実装。参考
- 遺伝アルゴリズム Genetic Algorithm: N個のランダムなパラメータの組み合わせ(遺伝個体)を作り、その評価関数を得て、2つの遺伝個体間で交叉と突然変異を行うことでより良いパラメータを得る。Deapで実装。参考
- SMBO TPE: パラメータに対する評価関数を予測する関数を使い、パラメータの変化でモデルがどのくらい改善されるかを予測して探索する。hyperoptを使う。参考
探せば他にもあると思います。
今回は上記のうち、ベイズ最適化、遺伝アルゴリズム、SMBO TPEをKerasと組み合わせてみたので、そのコードを紹介します。
(参考)グリッド・サーチとランダム・サーチ
Kerasを使ったグリッド・サーチとランダム・サーチは以下をご参照ください。
Keras with GridSearchCVでパラメータ最適化自動化
ニューラルネットワークのハイパーパラメータをランダムに探索してみる
KerasとLHSの組み合わせは見たことないですが、気が向いたら試すかもしれません。
検証のネタ
MNISTを使います。
MNISTを選ぶ理由は速い・楽・簡単だからです。
本当はCifar10で所要時間と評価関数を比較したかったのですが、時間がかかるので止めました。
Kerasとベイズ最適化
Kerasとベイズ最適化は以前試しています。
ベイズ最適化のKeras DNNモデルへの適用
今回も同じプログラムを使いますので、細かい説明は省きます。
プログラム全文はこちらのとおりです。
プログラムのうち、遺伝アルゴリズムやSMBO TPEでも使う共通部分を以下に掲載します。
MNISTのモデル(class MNIST())を定義し、学習を実行する関数(def run_mnist())を用意しています。
import numpy as np
import pandas as pds
import random
from keras.layers import Activation, Dropout, BatchNormalization, Dense
from keras.models import Sequential
from keras.datasets import mnist
from keras.metrics import categorical_crossentropy
from keras.utils import np_utils
from keras.optimizers import Adam
from keras.callbacks import EarlyStopping
# MNIST class
class MNIST():
def __init__(self,
l1_out=512,
l2_out=512,
l1_drop=0.2,
l2_drop=0.2,
bn1=0,
bn2=0,
batch_size=100,
epochs=10,
validation_split=0.1):
self.l1_out = l1_out
self.l2_out = l2_out
self.l1_drop = l1_drop
self.l2_drop = l2_drop
self.bn1 = bn1
self.bn2 = bn2
self.batch_size = batch_size
self.epochs = epochs
self.validation_split = validation_split
self.__x_train, self.__x_test, self.__y_train, self.__y_test = self.mnist_data()
self.__model = self.mnist_model()
params = """
validation_split:\t{0}
l1_drop:\t{1}
l2_drop:\t{2}
l1_out:\t{3}
l2_out:\t{4}
bn1:\t{5}
bn2:\t{6}
batch_size:\t{7}
epochs:\t{8}
""".format(self.validation_split,
self.l1_drop, self.l2_drop,
self.l1_out, self.l2_out,
self.bn1, self.bn2,
self.batch_size, self.epochs)
print(params)
# load mnist data from keras dataset
def mnist_data(self):
(X_train, y_train), (X_test, y_test) = mnist.load_data()
X_train = X_train.reshape(60000, 784)
X_test = X_test.reshape(10000, 784)
X_train = X_train.astype('float32')
X_test = X_test.astype('float32')
X_train /= 255
X_test /= 255
Y_train = np_utils.to_categorical(y_train, 10)
Y_test = np_utils.to_categorical(y_test, 10)
return X_train, X_test, Y_train, Y_test
# mnist model
def mnist_model(self):
model = Sequential()
model.add(Dense(self.l1_out, input_shape=(784,)))
if self.bn1 == 0:
model.add(BatchNormalization())
model.add(Activation('relu'))
model.add(Dropout(self.l1_drop))
model.add(Dense(self.l2_out))
if self.bn2 == 0:
model.add(BatchNormalization())
model.add(Activation('relu'))
model.add(Dropout(self.l2_drop))
model.add(Dense(10))
model.add(Activation('softmax'))
model.compile(loss='categorical_crossentropy',
optimizer=Adam(),
metrics=['accuracy'])
return model
# fit mnist model
def mnist_fit(self):
early_stopping = EarlyStopping(patience=0, verbose=1)
self.__model.fit(self.__x_train, self.__y_train,
batch_size=self.batch_size,
epochs=self.epochs,
verbose=0,
validation_split=self.validation_split,
callbacks=[early_stopping])
# evaluate mnist model
def mnist_evaluate(self):
self.mnist_fit()
evaluation = self.__model.evaluate(self.__x_test, self.__y_test, batch_size=self.batch_size, verbose=0)
return evaluation
# function to run mnist class
def run_mnist(l1_out=512, l2_out=512,
l1_drop=0.2, l2_drop=0.2,
bn1=0, bn2=0,
batch_size=100, epochs=10, validation_split=0.1):
_mnist = MNIST()
mnist_evaluation = _mnist.mnist_evaluate()
return mnist_evaluation
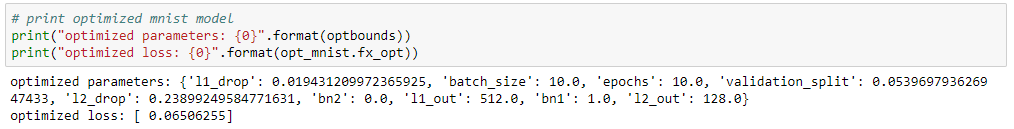
ベイズ最適化によるパラメータの結果は以下になります。
Kerasと遺伝アルゴリズム
Pythonで遺伝アルゴリズムを実装する際はDeapを使います。
プログラム全文はこちらです。
まずは必要なライブラリをインポートします。
from deap import base, creator, tools, algorithms
遺伝アルゴリズムでは個体のとる性質をパラメータで表します。
複数の性質をもつ個体を作る場合、各パラメータのとりうる値を定義して、'individual'に登録します。
# creator
creator.create('FitnessMax', base.Fitness, weights = (-1.0,))
creator.create('Individual', list , fitness = creator.FitnessMax)
# defining attributes for individual
toolbox = base.Toolbox()
# layer outputs
toolbox.register("l1_out", random.choice, (64, 128, 256, 512, 1024))
toolbox.register("l2_out", random.choice, (64, 128, 256, 512, 1024))
# dropout late
toolbox.register("l1_drop", random.uniform, 0.0, 0.3)
toolbox.register("l2_drop", random.uniform, 0.0, 0.3)
# batchnormalization
toolbox.register("bn1", random.randint, 0, 1)
toolbox.register("bn2", random.randint, 0, 1)
# training
toolbox.register("batch_size", random.choice, (10, 100, 500))
toolbox.register("epochs", random.choice, (5, 10, 20))
toolbox.register("validation_split", random.uniform, 0.0, 0.3)
# register attributes to individual
toolbox.register('individual', tools.initCycle, creator.Individual,
(toolbox.l1_out, toolbox.l2_out,
toolbox.l1_drop, toolbox.l2_drop,
toolbox.bn1, toolbox.bn2,
toolbox.batch_size, toolbox.epochs, toolbox.validation_split),
n = 1)
# individual to population
toolbox.register('population', tools.initRepeat, list, toolbox.individual)
# evolution
toolbox.register('mate', tools.cxTwoPoint)
toolbox.register('mutate', tools.mutFlipBit, indpb = 0.05)
toolbox.register('select', tools.selTournament, tournsize=3)
toolbox.register('evaluate', run_mnist)
各パラメータからランダムに値をとった性質をもつ個体を作り、その交叉と突然変異を繰り返すことで進化していきます。
def genAlg(population=5, CXPB=0.5, MUTPB=0.2, NGEN=5):
random.seed(64)
pop = toolbox.population(n=population)
print("Start of evolution")
fitnesses = list(map(toolbox.evaluate, pop))
for ind, fit in zip(pop, fitnesses):
ind.fitness.values = fit
print(" Evaluated %i individuals" % len(pop))
for g in range(NGEN):
print("-- Generation %i --" % g)
offspring = toolbox.select(pop, len(pop))
offspring = list(map(toolbox.clone, offspring))
for child1, child2 in zip(offspring[::2], offspring[1::2]):
if random.random() < CXPB:
print("mate")
toolbox.mate(child1, child2)
del child1.fitness.values
del child2.fitness.values
for mutant in offspring:
if random.random() < MUTPB:
print("mutate")
toolbox.mutate(mutant)
del mutant.fitness.values
try:
invalid_ind = [ind for ind in offspring if not ind.fitness.valid]
fitnesses = map(toolbox.evaluate, invalid_ind)
for ind, fit in zip(invalid_ind, fitnesses):
ind.fitness.values = fit
except AssertionError:
pass
print(" Evaluated %i individuals" % len(invalid_ind))
pop[:] = offspring
try:
fits = [ind.fitness.values[0] for ind in pop]
length = len(pop)
mean = sum(fits) / length
sum2 = sum(x*x for x in fits)
std = abs(sum2 / length - mean**2)**0.5
print(" Min %s" % min(fits))
print(" Max %s" % max(fits))
print(" Avg %s" % mean)
print(" Std %s" % std)
except IndexError:
pass
print("-- End of (successful) evolution --")
best_ind = tools.selBest(pop, 1)[0]
print("Best individual is %s, %s" % (best_ind, best_ind.fitness.values))
return best_ind
最適なパラメータはbest_indに格納しています。
KerasとSMBO TPE
Hyperoptの使い方はこちらが参照になります。
プログラム全文はこちらです。
まずは必要なライブラリをインポートします。
from hyperopt import hp, tpe, Trials, fmin
import numpy as np
import pandas as pds
import random
from keras.layers import Activation, Dropout, BatchNormalization, Dense
from keras.models import Sequential
from keras.datasets import mnist
from keras.metrics import categorical_crossentropy
from keras.utils import np_utils
from keras.optimizers import Adam
from keras.callbacks import EarlyStopping
MNISTの学習実行関数は引数にargsを取ります。
# function to run mnist class
def run_mnist(args):
_mnist = MNIST(**args)
mnist_evaluation = _mnist.mnist_evaluate()
print("loss:{0} \t\t accuracy:{1}".format(mnist_evaluation[0], mnist_evaluation[1]))
return mnist_evaluation[0]
ベイズ最適化や遺伝アルゴリズム同様、TPEでもパラメータの範囲や候補値をdictで指定します。
hyperopt_parameters = {
'validation_split': hp.uniform('validation_split', 0.0, 0.3),
'l1_drop': hp.uniform('l1_drop', 0.0, 0.3),
'l2_drop': hp.uniform('l2_drop', 0.0, 0.3),
'l1_out': hp.choice('l1_out', [64, 128, 256, 512, 1024]),
'l2_out': hp.choice('l2_out', [64, 128, 256, 512, 1024]),
'bn1': hp.choice('bn1', [0, 1]),
'bn2': hp.choice('bn2', [0, 1]),
'batch_size': hp.choice('batch_size', [10, 100, 500]),
'epochs': hp.choice('epochs', [5, 10, 20]),
}
探索します。
# number of evaluation
max_evals = 20
# trials instance
trials = Trials()
best = fmin(
# function to minimize
run_mnist,
# list of hyperparameters
hyperopt_parameters,
# optimization logic
algo=tpe.suggest,
max_evals=max_evals,
trials=trials,
# output evaluations
verbose=1
)
上記で変数bestに最も良いパラメータを入れていますので、標準出力します。
まとめ
ハイパーパラメータチューニングの方法をいろいろ試してみました。
いずれも共通しているのは、
- パラメータの範囲は自分で決めねばならない
- 時間がかかる
ことです。
プログラムの書き方はいずれも大差ない(ニューラルネットワークモデルを定義する → パラメータの範囲を決める → 探索する)ですが、ベイズ最適化とSMBO TPEが所要時間が短く(=探索回数が少ない)済みました。
遺伝アルゴリズムは探索前の事前作業として個体を作って評価する(トレーニングする)ため、所要時間が長いようです。
MNISTの判定モデルでは、もはや精度に大差が出ないので、時間があったらもう少し差異が出るネタを使ってみます。