連続で失礼します。少しづつパソコンに慣れてきているところです、、
今回は統合したデータについて、簡単な検索を行ってみようと思います!!
- 今回タグにSQLを入れていますが、これはあくまでもSQLライクのクエリ言語です!!
- PGQLというタグも勝手に入れていますが、これがPGXで用いている専用言語です!!
---
今回入っているデータを整理すると
- 薬剤-タンパク質のつながりのデータ(有向性アリ)
- タンパク質-タンパク質のつながりのデータ(有向性ナシ)
なので、このイメージでやっていきましょう!
①ある薬剤が結合するタンパク質は何?
pgx > G.queryPgql(" \
SELECT a.name, x.name \
WHERE (x)-->(a), x.name='DB00099' \
").print(10)
「薬剤番号99番の薬剤はどのタンパク質に結合する?」という質問
<結果>
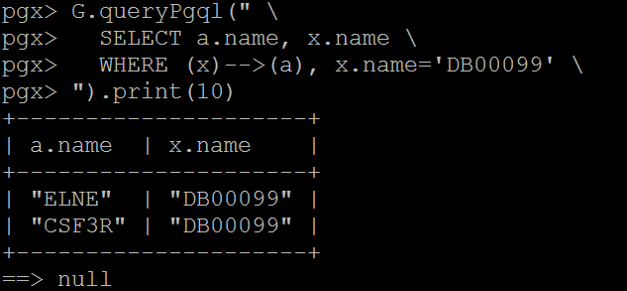
2つのタンパク質が表示されました!このように、今回はどちらが薬剤でどちらがタンパク質かが分かるのでノードの識別の必要はないです。
②その続きのつながりは?
pgx > G.queryPgql(" \
SELECT x.name, a.name, b.name, r.combined_score \
WHERE (x)-->(a)-[r]-(b), x.name='DB00099' \
").print(10)
薬剤番号99番の薬剤が結合するタンパク質は他にどのタンパク質と繋がっている?という質問
<結果>
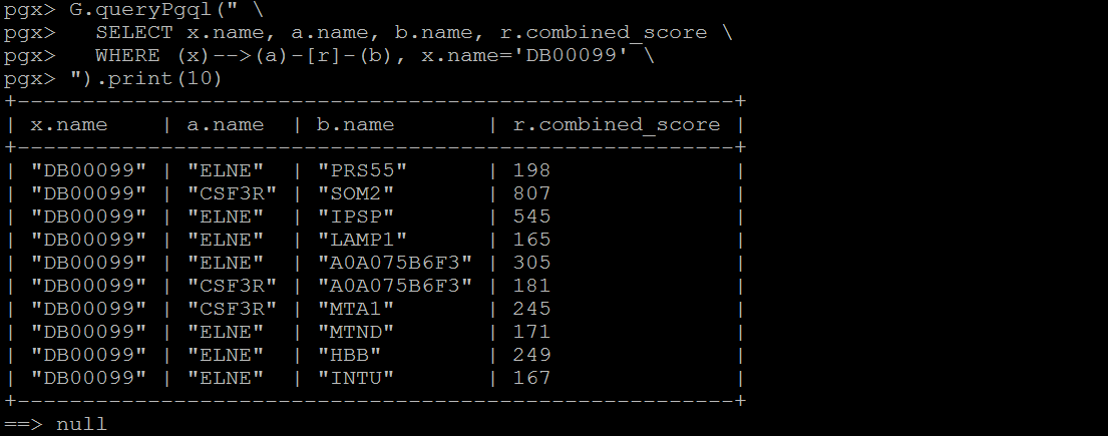
色々出てきました!ネットワークはこの時点で複雑になりそうな予感がしますね、、、
なお、タンパク質同士の結合力も r.combined_scoreという形でクエリ内で指定して見ています!
このような感じで「()-[r]-()」ノード()をつなぐエッジ--の間に参照したい値を入れれば簡単に検索することが出来るのもPGXのいいところです!
③ある薬剤Aと薬剤Bが結合しているタンパク質同士はつながりを持つ?
pgx > G.queryPgql(" \
SELECT x.name, a.name, b.name, y.name, r.combined_score \
WHERE (x)-->(a)-[r]-(b)<--(y), x.name='DB00099', y.name='DB00244' \
ORDER BY r.combined_score DESC \
").print(10)
<結果>
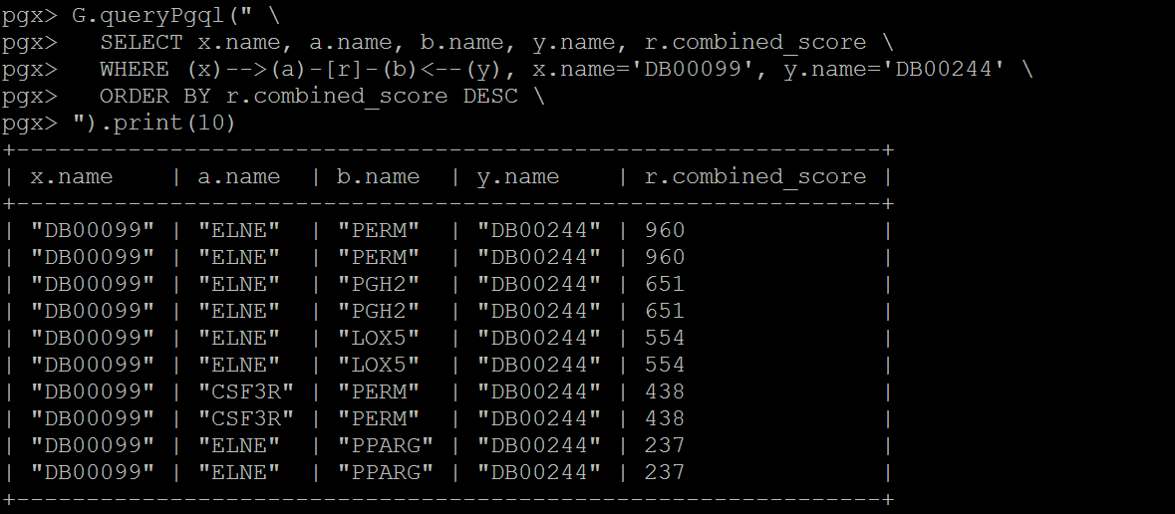
重複していますが出てきました!もしかしたら、この画像の”ELNE”と”PERM”というタンパク質は薬剤番号99番と244番の影響をどちらも受けるのかもしれませんね。繋がっているから!
---
簡単なクエリ検索を行ってみましたが、このようにグラフ構造をとっていれば2つ以上のDBを統合しても検索しやすいですね!
今後は可視化を視野に入れていきたいと思っています!
それでは~