この記事の1番の目的
絶対にもっといい方法があるはずなのでそれを有識者に教えてもらうこと
現状の結論:for loopを安直に使えばだいたいよさそう。
やりたいこと(タイトル参照)
あるリスト(a)内で,ある値(value_search)に最も近い値のインデックスが欲しいとき,例えば
a = np.arange(0,10,1)
value_search = 5
idx = np.argmin(np.abs(a - value_search))
みたいに探すと思う.
参考
問題なのは,この探したい値が単一の値ではなく,リストのような場合に
a = np.arange(0,10,1)
value_list_search = np.array([1,5,7])
idx = np.argmin(np.abs(a - value_list_search))#できない
とやりたいが,それはできない.次のようなエラーがでる.
ValueError: operands could not be broadcast together with shapes (10,) (3,)
長さのちがうリスト同士の差なので差を取れない.それはそう.ついbroadcastを信じてしまう.(後述の方法はこれを修正する)
解決策として,for文で value_list_search の要素をひとつひとつ取り出してidxをそれぞれで求める,という方法が考えられる.多分よくやられる方法.
a = np.arange(0,10,1)
value_list_search = np.array([1,5,7])
idx_list = []
for value_search in value_list_search:
idx = np.argmin(np.abs(a - value_search))
idx_list.append(idx)
idx_list
ただこのやり方はパイソニックでないと思う.この処理が少ない場合は気にならないだろうが,大規模かつ何度もやるようになると時間がかかる.(と思った)
pythonではfor文は避けたい.numpyを大規模化にして処理した方がよいのでは?
以上がモチベーション.
解決方法:numpyのbroadcastを有効活用.
横ベクトル同士の差を横ベクトルと縦ベクトルの差にする→答えとして行列が返される.
reshapeつかって片方を縦ベクトルにする.
a = np.arange(0,10,1)
value_list_search = np.array([1,5,7])
value_list_search = value_list_search.reshape(3, 1)
idx_list = np.argmin(np.abs(a - value_list_search), axis = 1)
これができるのがnumpyのいいところ.
速度比較
ということでそれぞれの方法の速度を比較をしてみた.
def make_list(length, length_search):
a = np.arange(0, length, 1)
value_list_search = np.random.randint(0, length, (length_search))
return a, value_list_search
def search_with_for(a, value_list_search):
idx_list = []
for value_search in value_list_search:
# idx = np.argmin(np.abs(a - value_search))
idx = np.abs(a - value_search).argmin() #np.argmin()より若干速い
idx_list.append(idx)
return idx_list
def search_with_reshape(a, value_list_search):
value_list_search = value_list_search.reshape(np.size(value_list_search),1)
idx_list = np.argmin(np.abs(a - value_list_search), axis = 1)
return idx_list
def test_time(search_method, length, length_search):
t1 = time.time()
for i in range(10):
a, value_list_search = make_list(length, length_search)
search_method(a, value_list_search)
t2 = time.time()
elapsed_time = t2 - t1
print(f"length = {length}, length_search = {length_search}, 経過時間:{elapsed_time}")
return elapsed_time
参考:
結果
パラメータを振って調べた結果がこれ.
l = length(探索対象のリストの大きさ),ls = length_search(探索したい値のリストの大きさ).
値は対数で,log_10(かかった時間)を表す.値が小さい方が高速で良い.
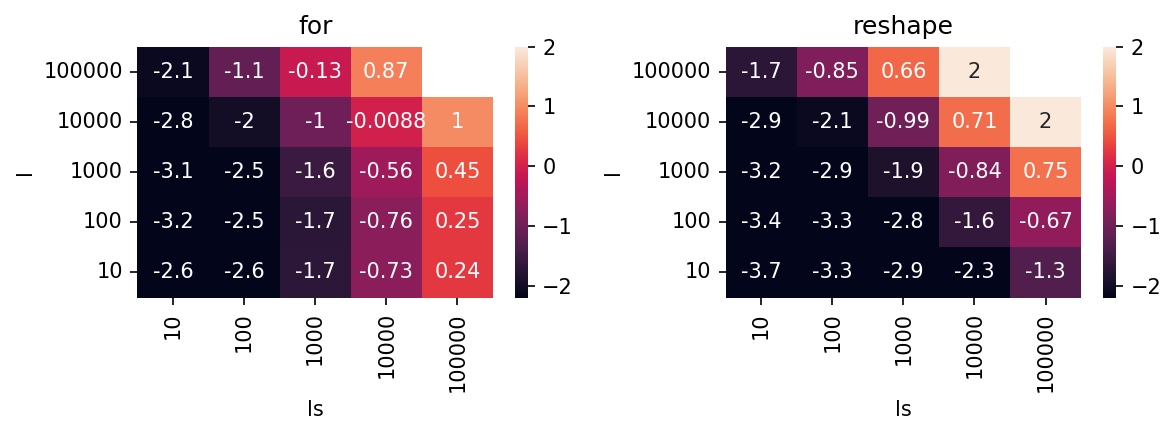
考察
for loopの回数が大きくなるlsが大きい領域でforの方法が遅くなる.ただlの増加には比較的鈍感.lsが大きい領域でforの方法が不利になり,reshapeの方が有利.
lが大きくなってもfor loopの回数が増えるわけではないので,この振る舞いは納得(の割には増えてるな).
小規模の範囲ではreshapeの方法が早いが、大規模化するとfor の方が早いという想定とは逆の結果になった。
今回の問題の場合、想定される状況としてはl >> lsが多いと思うので,結局のところfor loopを使う方法が有利な状況になる.
まとめ
for loopでええわ.解散.
小規模なリストの場合 or 探す値が多い場合はreshapeも考える価値あり.場合によっては一桁早い.
