以前にもマハラノビスとirisの記事を書いたけど、
前の記事見直すと分かりにくいし、いい記事を書いてくださった方がいたので。
コードを少し改修しています。
早速
train_index <- c(1:10)
train <- iris[train_index,]#setosaであれば受け入れる
test <- iris[-train_index,]#setosaも含むがはじきたいものも含んでいる
マハラノビス距離を求めるための分散共分散行列と列平均
## Training (setosa)
center <- apply(train[,1:4], 2, mean)
covar <- cov(train[,1:4])
# Calculate distance
test[1,1:4]
# Sepal.Length Sepal.Width Petal.Length Petal.Width
# 11 5.4 3.7 1.5 0.2
test[41,1:4]
# Sepal.Length Sepal.Width Petal.Length Petal.Width
# 51 7 3.2 4.7 1.4
test[91,1:4]
# Sepal.Length Sepal.Width Petal.Length Petal.Width
# 101 6.3 3.3 6 2.5
人間がこの数値だけ見ても何がどの種に該当するかわからない。
成長によるばらつきだってあるだろうし。
mahalanobis( test[1,1:4], center, covar ) #setosa
# 11
# 4.85708
mahalanobis( test[41,1:4], center, covar ) #versicolor
# 51
# 1424.809
mahalanobis( test[91,1:4], center, covar ) #virginica
# 101
# 3176.181
setosaだけはマハラノビス距離が近いものであると判定されている。
判別できている
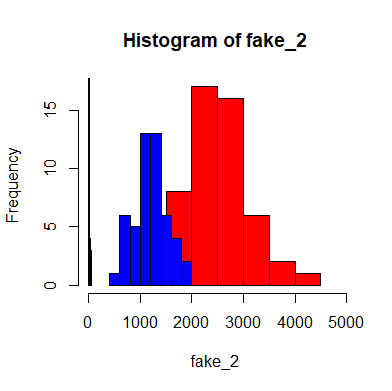
元記事ではヒストがあったがコードはなかったので以下。
colect<-mahalanobis( test[1:40,1:4], center, covar ) #setosa
fake_1<-mahalanobis( test[41:90,1:4], center, covar ) #versicolor
fake_2<-mahalanobis( test[91:140,1:4], center, covar ) #virginica
hist(fake_2,col="red",xlim=c(0,4900))
hist(fake_1,col="blue",add=T)
hist(colect,add=T)
setosaはほとんどすべてが0付近に集まっている。
他の種は離れている。
同じような特徴を持っている変数があっても、他の変数で差があれば検知できる。