Seurat、楽しいですよね。チュートリアルも充実しているし、公式からのフィードバックも超丁寧で...
でも、覚えることが多すぎて、ムリゲーと感じている人も多いのではないでしょうか。特に理論的な部分については、初見では難しいのではないかと思います。
今回は図の見方と、ちょっとした理論に焦点を当て、そういった方の理解を助けられたらなと思います。
大まかなポイントは以下の2つです。
① 正規化は、細胞間比較(クラスター間比較)のために必須
※ 遺伝子間比較は、scRNA-seq では現状不可能
② 標準化(Z-Score 変換)は、遺伝子間の scale を合わせるために必要
順を追って説明します。
まず、scRNA-seq データは、遺伝子数×細胞数の、行列データになっています。主な目的は細胞間の発現比較です。
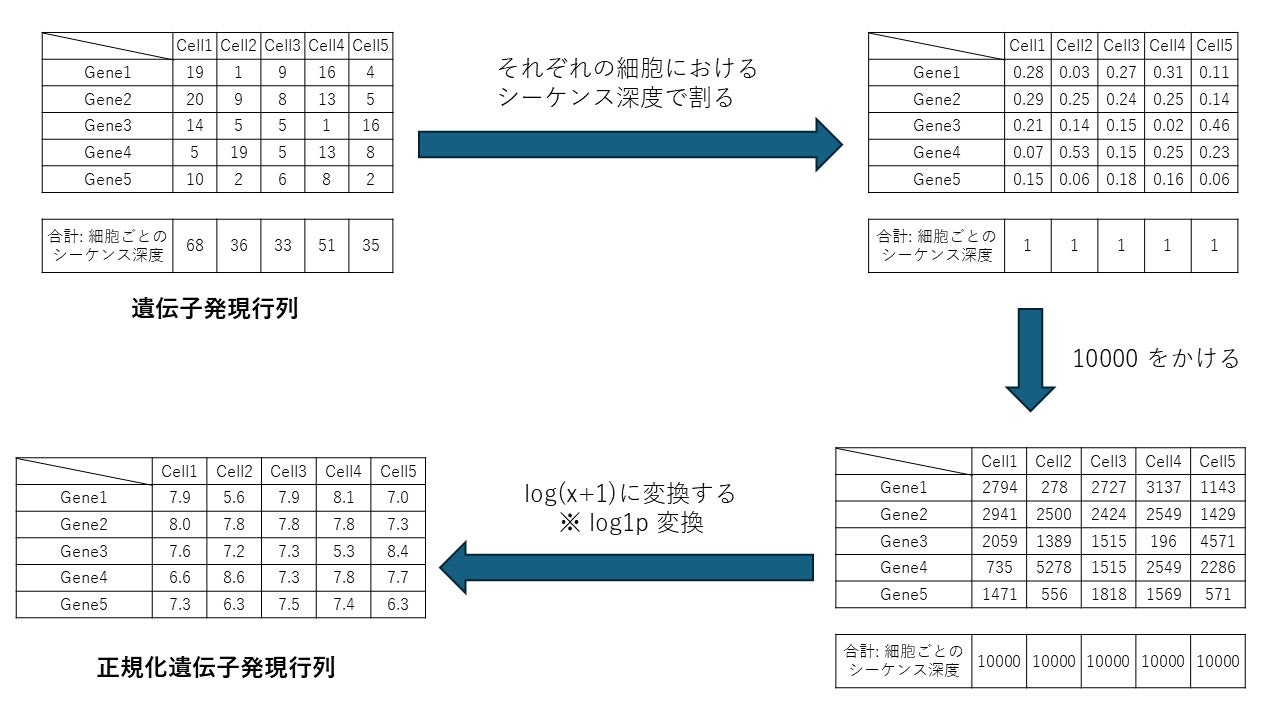
細胞間発現比較の際には準備として、細胞ごとの mRNA 読み込み深度(シーケンス深度)を修正する必要があります。この操作を正規化と言います。正規化には二種類ありますが、ここでは簡単な LogNormalize を紹介します。
画像の通りですが、式にすると以下のようになります。
$$
x_{norm,ij} = \log\left(\frac{x_{i,j}}{\sum_{i=1}^{n_i} x_{i,j}}\times10000 + 1\right)
$$
xi,j: 細胞 j における遺伝子 i のカウント値
ni: 遺伝子数
10000倍しているのは、小さすぎる値に密集することを防ぐためで、対数変換は大きすぎる値を丸めるためです。また、対数変換の際には +1 をすることで、対数変換時に log0 が発生することを防いでいます。
bulk の RNA-seq に慣れている人にとっては以下が分かりやすいでしょうか。
$$
x_{norm,ij} = \log\left(\frac{CPM_{i,j}}{100} + 1\right)
$$
ちなみに、正規化の定義として良く聞くのが「最小値 0, 最大値 1 にする変換」ですが、今回も Σ の式で割ることにより、実質的に同じ操作を含んでいます。
ではこれを踏まえた上で、VlnPlot を見てみましょう。データの参考文献は最後に記載します。
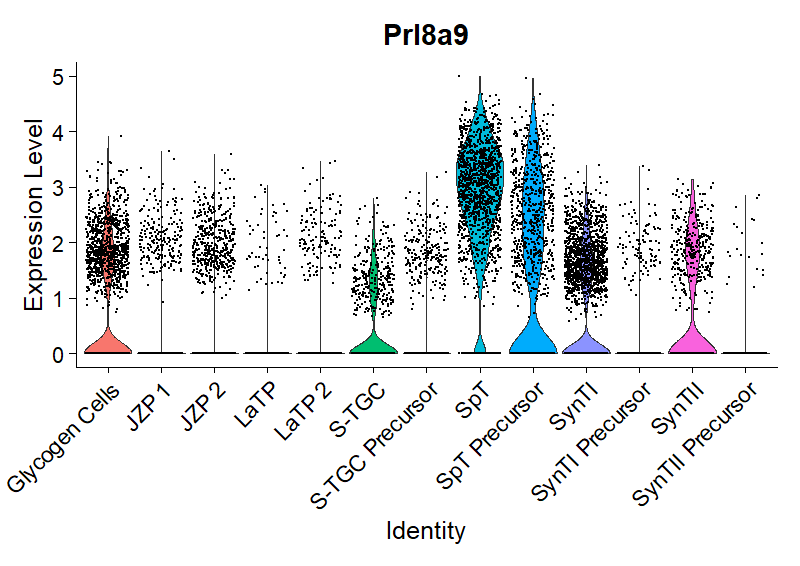
図は、マウス胎盤栄養膜細胞における Prl8a9 遺伝子の VlnPlot です。縦軸は先ほど計算した正規化後の値、点は細胞(核)を表します。バイオリンは細胞の分布を示す密度で、0 付近に見える峰は、0 未満の密度がカットされているために生じているものです。本来は、マイナス方向の密度がある、二峰性の密度分布をしています。検出が無い細胞も多いということですね。ちなみに、あまりにも 0 が多い場合には、 Violin 自体が描画されないようです。
これらを踏まえ、VlnPlot の解釈としては、Prl8a9 は SpT に多く、SynTI, SynTII, S-TGC, Glycogen Cells にも少しは発現がありそうだ、となります。バイオリン の描画が無い部分は判別不可です。
では次に、DotPlot を見てみましょう。
ここでも大事なのは、細胞間 (クラスター間)比較 であるということです。まずは Average Expression から、順を追って説明します。先ほど、正規化の計算について説明しました。AverageExpression では、正規化後の値を真数変換した後、クラスターごとに算術平均を取ります。
$$
Average Expression_{i,Cluster_k} = Average_{j ∊k}\left(\frac{x_{i,j}}{\sum_{i=1}^{n_i} x_{i,j}}\times10000\right)
$$
$$
= Average_{j ∊ k}\left(\frac{CPM_{i,j}}{100}\right)
$$
k: クラスター k
大小関係を見るだけならこのままでも良いのですが、遺伝子を並列する場合にはスケールを合わせる必要があります。身長の大小関係と、体重の大小関係が分かっても、値としてそのまま表示することが出来ないのと同じです。そこで、クラスターごとの平均発現に対して標準化(Z-Score 変換)を行います。
$$
Z_{i, Clusterk} = \frac{Average Expression_{i, Clusterk} - μ_{i , clusters}}{σ_{i, clusters}}
$$ μi,clusters: (AverageExpression i, Clusterk) の平均
σi,clusters: (AverageExpression i, Clusterk) の標準偏差
これでようやく、Average Expression として遺伝子の並列表示が出来ます。ただし、スケールを変えたと言っても、表示上の問題を解決するためであって、遺伝子間比較はできないという事実は変わりません。
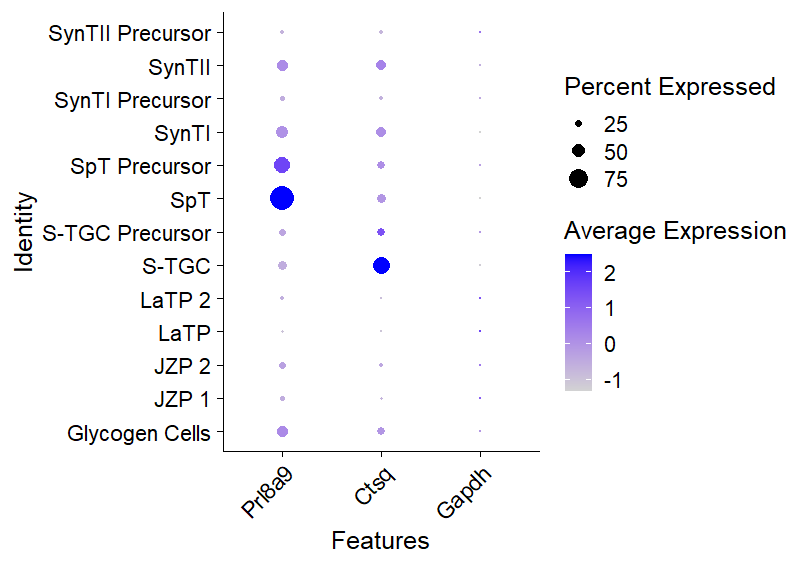
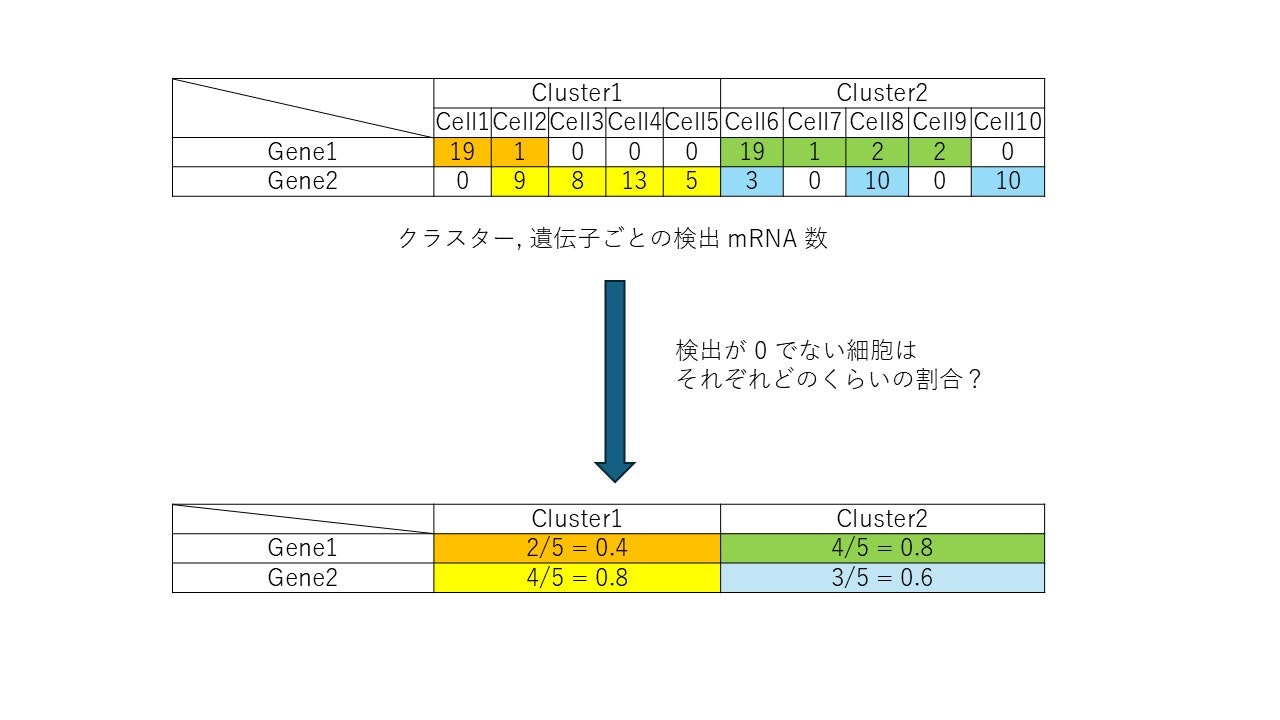
では、次に、Percent Expressed について説明します。これは図に示したほうが早いかもしれません。
この、"検出が 0 でない細胞の割合" が Percent Expressed です。
色の濃淡だけでは反映できない、遺伝子ごとの検出の程度を表す指標となっています。この値も、遺伝子間比較はできないので注意してください。遺伝子ごとの信頼性のようなものです。
これらを踏まえ、DotPlot の解釈としては、Prl8a9 は SpT に多く、Ctsq は S-TGC に多いだろう、となります。Gapdh については検出があまり見られていないので、判断がつきません(細胞核のデータなので、当たり前と言えば当たり前)。
いかがだったでしょうか。今回は DotPlot, VlnPlot を見ていきました。scRNA-seq の基本となる図なので、見方の把握は必須だと思います。Z スコア変換はヒートマップにも使用されているので、DotPlot との違いを考えてみるのも大事だと思います。これからもシングルセル、楽しんでいきましょう。
次回は Seurat Object の話をするか、SCTransform の話をするか、悩みどころです。
参考文献
Marsh B, Blelloch R. Elife. 9: e60266. (2020)