概要
Ruibo Wu が報告した天然物の生合成の"逆合成解析"ツール BioNavi のインストールログ。
BioNavi は、天然物の生合成経路を予測するための深層学習に基づく新しいツール。Transformer ニューラルネットワークと AND-OR 木に基づいた計画アルゴリズムを組み合わせている。これにより、既知の生合成反応と有機化学反応のデータを活用して、天然物から単純な出発原料への複数の経路を効率的に検索することができる。このツールは、テストセットにおいて既存の手法よりも高い精度と効率を示し、新しい経路の発見や既知の経路の再構築に役立つことが示されている。
BioNavi の元論文は以下。
2022 年 Nature Communication
2024 年 JACS Au
検証環境
CPU: Ryzen 9 7950x
GPU: Nvidia A6000 (vRAM:48GB)
RAM: 96 GB
Fedora40
インストール手順
# BioNavi のダウンロード
sudo git clone https://github.com/zengtsysu/BioNavi
# miniconda の設定
sudo wget https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh -O miniconda.sh
bash miniconda.sh
source ~/miniconda3/etc/profile.d/conda.sh
conda init bash
conda create -n bionavi python==3.7
conda activate bionavi
# BioNavi のインストール
cd BioNavi
sh install.sh # /opt で実行すると権限エラーで止まる。
# 学習済みモデルの配置
cd transformer/checkpoints
wget https://figshare.com/ndownloader/files/44330843
unzip 44330843
実行
BioNavi/config/bionavi_conf.yaml を編集する。
1 行目の target_mol の部分に探索対象の化合物の smiles を記入する。
buliding_block_path のデータセットの csv ファイルを切り替える。
生合成の逆合成解析の場合は、bio_building_block.csv を使用する。
結果を保存するために、viz: Fales を viz: True に変更する。
# 以下のコマンドで計算を実行する
cd BioNavi
# export PYTHONPATH=$PYTHONPATH:$(pwd)/onmt
python run_bionavi.py
5 - 10 分ほどで計算は完了する。
探索結果は、BioNavi/viz に保存される。
search_tree は terminal、svg ファイルは web ブラウザで開く。
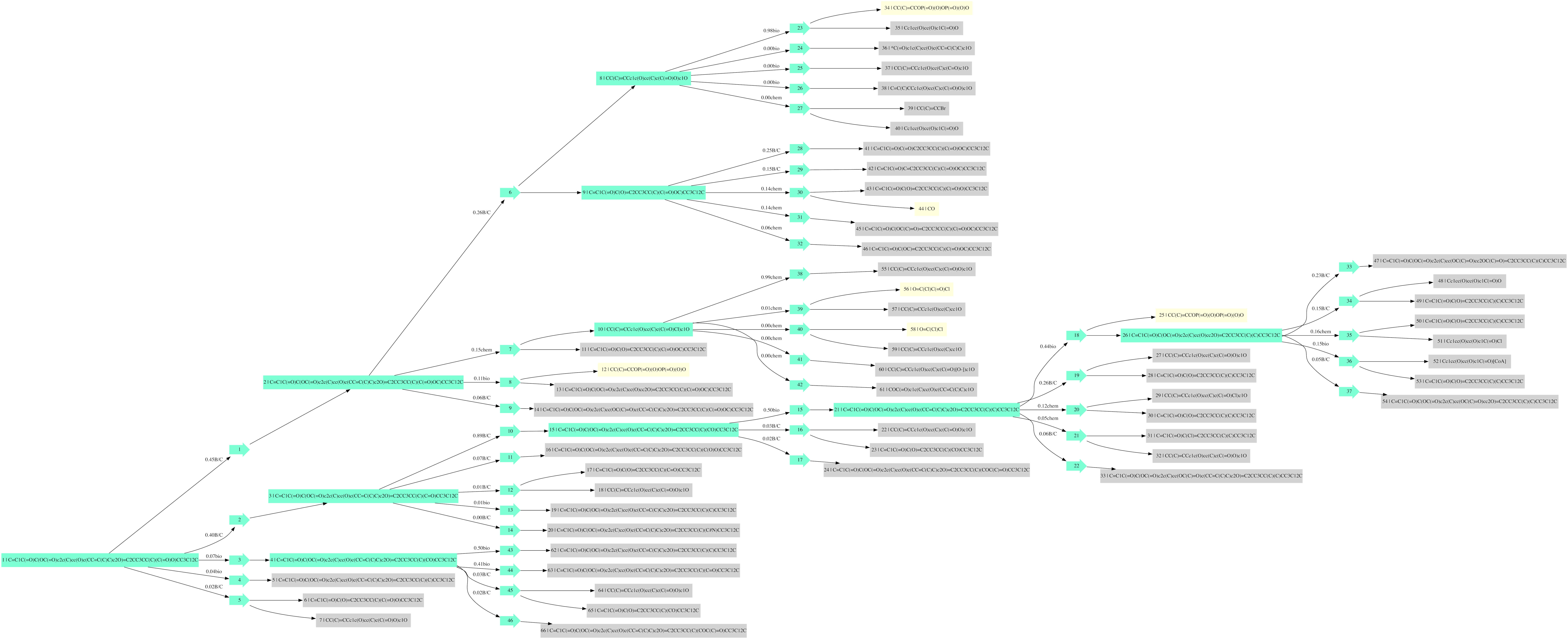
以下のように search_tree というファイルに smiles の一覧が書かれている。
digraph G {
rankdir=LR
node [shape=box]
"1 | C=C1C(=O)C(OC(=O)c2c(C)cc(O)c(CC=C(C)C)c2O)=C2CC3CC(C)(C(=O)O)CC3C12C" [color=aquamarine shape=box style=filled]
1 [color=aquamarine shape=rarrow style=filled]
"1 | C=C1C(=O)C(OC(=O)c2c(C)cc(O)c(CC=C(C)C)c2O)=C2CC3CC(C)(C(=O)O)CC3C12C" -> 1 [label="0.45B/C"]
2 [color=aquamarine shape=rarrow style=filled]
"1 | C=C1C(=O)C(OC(=O)c2c(C)cc(O)c(CC=C(C)C)c2O)=C2CC3CC(C)(C(=O)O)CC3C12C" -> 2 [label="0.40B/C"]
3 [color=aquamarine shape=rarrow style=filled]
"1 | C=C1C(=O)C(OC(=O)c2c(C)cc(O)c(CC=C(C)C)c2O)=C2CC3CC(C)(C(=O)O)CC3C12C" -> 3 [label="0.07bio"]
4 [color=aquamarine shape=rarrow style=filled]
"1 | C=C1C(=O)C(OC(=O)c2c(C)cc(O)c(CC=C(C)C)c2O)=C2CC3CC(C)(C(=O)O)CC3C12C" -> 4 [label="0.04bio"]
5 [color=aquamarine shape=rarrow style=filled]
"1 | C=C1C(=O)C(OC(=O)c2c(C)cc(O)c(CC=C(C)C)c2O)=C2CC3CC(C)(C(=O)O)CC3C12C" -> 5 [label="0.02B/C"]
...
search_tree.svg に下図のように代謝経路が示される。
smiles 表記は、以下のサイトを利用すると簡単に構造式に変換できる。
https://www.hpc.co.jp/tools/index_smi_to_2d.html