はじめに
こんにちは 2023iGEMGifu代表の今川です。初出場で初代表は苦労も多かったですがiGEMに取り組む前と後では見違えるほど知識もついたし技術もついたと思います。

目次
今回感じたこと
AIを使ったプロジェクト空中戦じゃね?
空中戦: AIを使うほうもジャッジ側も理解も不十分でありモデルの複雑さ、学習データの量で評価している事
iGEMerでありKagllerである私(iGEN Kaggle部作りたい)はモデルと適切に評価することが一番難しいと感じています。
特に過学習(評価データや事前に知りえないデータを学習に使用すること(学校のテストでいうとカンニングや過去問の入手?))を起こさないように十分気を付ける必要があります。
この記事の目的と結論
そこで入賞チームの研究を読み
機械学習モデルの評価方法、
ベンチマークとの比較方法
を通じ機械学習モデルを過大評価(もしくは過少評価)していないか調べることでiGEM2023ではiGEM)はAIを使用したプロジェクトを適切に評価されているかわかるのではないかと考えました。
結論
-入賞チームの研究の見せ方、研究デザインには改良の余地がまだある。
-ジャッジにも機械学習の素養を持った人がもっと加わると良い。
(上から目線ですいません![]() )
)
2nd Runner Up 🥉を受賞したNUS-Singaporeの研究
このiGEMプロジェクトでは、二つの主要なモデリング目標が設定されました。
そのうちの2つ目に機械学習を使用してました。
wiki: https://2023.igem.wiki/nus-singapore/
研究概要(機械学習に関わるとこのみ)
表1 機械学習モデル別に見た研究の概要
研究内容に対応するようコメントを書いてみました。

下に行くほど複雑なモデルになっておりが分かると思います。
->線形回帰モデル,MLP,トランスフォーマーで学習するにつれて精度が良くなった印象を受けるが実際は3つのモデルで予測しているものは違うので一概に精度を比較することはできません。
今回評価指標に使われていた最小二乗誤差(MES)などの大きさはタスク(予測するターゲット)によりスケールが違うので小さいから良いモデルといえることはなく相対的な指標です。
->各モデル一試行分しか記載がなく工夫点が実際にどれほど予測に寄与しているか分かりません。
次に3つ目のモデルについて詳しく見ていきます。
- ディープラーニングモデル(RNAスイッチ):
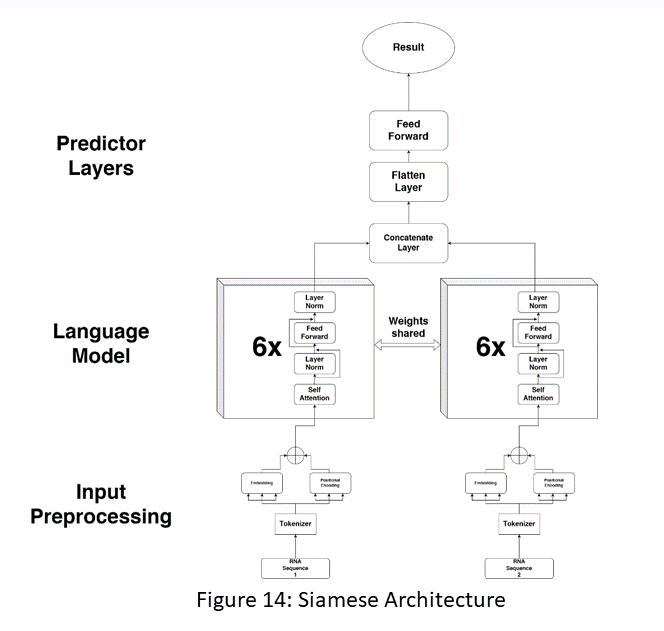
図1 予測モデル(Siamese Large Languageモデル)の構造
wikiに記載されたもの
we trained it from scratch ourselves.と記載があった
->複雑なモデルの学習には膨大なデータが必要であり、訓練するには相当な苦労があったと思う。
評価方法について
生成モデルがデザインしたスイッチを用いて実験したわけではないのでこのワークフローをそのまま検証することはできず実際には先行研究(Angenent-Mari et al., 2020)のデータセットで評価している。
->個人的にここが一番のトラップ(生成モデルも含めて評価したと誤解を与える)
->学習と推論データをどんな比率で分割したのか書いてあると良いと思いました。
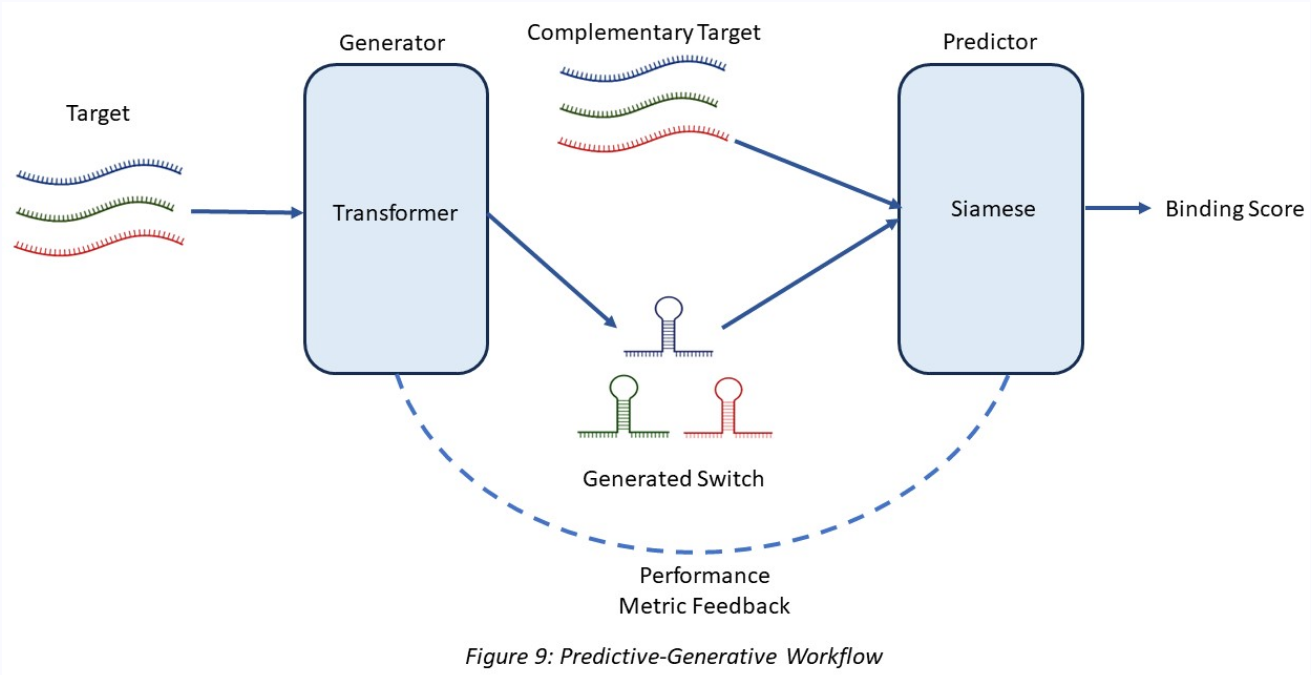
図2 3つ目のモデルのワークフロー
wikiに記載されたもの
まとめ
研究の見せ方、研究デザインには改良の余地がまだあると思いました
3つ目のトランスフォーマーモデルについて生成モデルからの出力を特徴量にしているが実際に評価するときには生成モデルは使用していない点に気づかないとこの研究を過大評価しかねないのでしっかりその旨を明記する必要があると感じました。
(私がジャッジならこの点を質問していたと思います。)
入賞研究を批判する暗い感じの記事になってしまいましたが最後まで読んでいただき嬉しいです。NUS-Singaporeの2023の研究は気になる点は多々見受けられるが機械学習以外のパートも含めて素晴らしい内容となっていてAIを使用したiGEMの先駆けとしては良い例だと思います。
今後この分野(分子生物学×AI)の発展が加速していく事は間違いないと思います。
学生が分子生物学×AIに実践的に触れられる場はiGEMだけでなく以下のような大会でも可能です。良かったらどうぞ―
Stanford Ribonanza RNA Folding(kaggle)
https://www.kaggle.com/competitions/stanford-ribonanza-rna-folding/overview