Chandra 衛星のデータ解析の方法
Chandra 衛星の初学者に向けて、データ解析についての基本を簡単にまとめた。
- ver1: (2019.1.25, ciao ver4.10), updated 2019.3.29 for ciao version 4.11)
- ver2: updated 2020.1.3. ciao 4.11
- ver3: updated 2021.4.6. ciao 4.13
Chandra衛星の基礎
詳しい方向けには、下記の資料は CIAO で何ができるかよくまとまっています。
- Did you know Chandra & CIAO do this?
基礎スペックの簡単なまとめ
- 角度分解能
- Chandra衛星は0.5秒角というX線帯域で最高の角度分解能がウリ。XMM-Newton衛星は10秒角度、すざく衛星は100秒程度と、1桁から2桁の角度分解能の違いがある。
CIAOを使ったイメージ作成とデコンボリューションについては下記の酒井さんの記事を参考にしてください。
- 焦点面検出器
- CCD(ACIS) : ACISはエネルギー分解能が~100eVと高いが、時間分解能はCCDの読み出し時間で律速するため天体だとパイルアップしやすい。
- MCP(HRC)HRC : 2つのMCPと周りをプラシンチのantiで囲ったもので構成。エネルギー分解能が悪い(ほとんどない)が、単一光子の検出時間できまるので、時間分解能が良いのがメリット。HRC-Iは100nmのAl, HRC-Sは80nmのAlのフィルタのため、有効面積はHRC-Iの方が若干高い(cf. HRC focal plane layout)。
- HRC-I
- 10cmx10cmのMCPで主にイメージャーとして用いる。
- HRC-S
- 10cmx3cmのMCPで、主にLETGと組み合わせて、分光+sub-arcsecのイメージングの両方に持ちる。アンチコとのタイミングエラーがあるようで、バックグランドがHRC-Iよりも高め(cf. Event Screening for HRC)。
- HRC-I
- 回折格子
- 焦点面検出器とは独立に、ミラーと焦点面検出器の間の光路に回折格子を入れることが可能。これにより、光子を分散し、焦点面検出器上に、回折格子で分散された0字光から高次光が撮像され、CCDよりも高エネルギー分解能が可能になる。ただし、回折光をいれることで光が分散されるため有効面積は下がる。
- ゼロ次光(エネルギー分解能は焦点面検出器に依存)
- 高次光(エネルギー分解能は結像場所(角度)に依存)
- 焦点面検出器とは独立に、ミラーと焦点面検出器の間の光路に回折格子を入れることが可能。これにより、光子を分散し、焦点面検出器上に、回折格子で分散された0字光から高次光が撮像され、CCDよりも高エネルギー分解能が可能になる。ただし、回折光をいれることで光が分散されるため有効面積は下がる。
Chandra/HETGについては下記の酒井さんの記事を参考にしてください。
基礎情報
-
基礎資料
-
有効面積
-
最新の有効面積
- オーダー
- ACIS-I, ACIS-S (NONE=gratingなし) 400cm^2 at 4keV
- HRC-I 50cm^2 at 4keV
- ACIS-S HETG の 0 次光 120cm^2 at 4keV
- HRC-S LETG の 0 次光 5cm^2 at 4keV
- Suzaku ~200cm^2 x 望遠鏡の台数 at 4keV
- オーダー
-
最新の有効面積
-
アーカイブデータの検索
-
Chandraのマニュアル
Chandra衛星のデータ解析のための準備
必要なソフトウェアのリスト
-
ciao
- ciao-install を実行すると、CALDBのダウンロードから、ciaoのインストールまでしてくれる。
- CALDB(~3GB)のダウンロードが遅い場合は、別途ダウンロードして、ciao-instalの作業場所に置いおくと、再インストールはされずに早く終わる。
-
ds9
- X線の画像解析ソフト。
-
heasoft
- Chandraの場合はciaoだけで色々とできるが、xspecやftoolsが使えたほうが何かと便利なので入れておく。
- [python] (https://www.python.org)
- version 4.10 より古い場合は、ciao-install --python2、として python2系を使うことを陽に指定できた。4.11以降は変更になり、python3系がmustになった。
- anacondaでpythonを入れておくと、astropyを用いたfitsの読み書きから、WCS、時刻変換、などが簡単に行える。
動作確認
起動チェック
/ciao-4.10/bin/ciao.bash を実行して、
> ciao.bash
IAO configuration is complete...
CIAO 4.10 Thursday, April 12, 2018
bindir : ..../software/ciao/ciao0410/install/ciao-4.10/bin
CALDB : 4.8.1
とエラーが出ずに表示されることを確認する。
これは、2021年以降ではobsoleteになっており、ファイルのダウンロードも 4.10では動かない。
ciao 4.3 以降は、
conda activate ciao-4.13
で activate して、
(ciao-4.13) [syamada] $ download_chandra_obsid 659 -d [~/work/tmp/test/ciao]
download_chandra_obsid: 05 March 2021
Using CDA HTTPS site
Setting up for ObsId 659
Looking for directory: https://cxc.cfa.harvard.edu/cdaftp/byobsid/9/659
Finding all files available at: https://cxc.cfa.harvard.edu/cdaftp/byobsid/9/659
Recursing into https://cxc.cfa.harvard.edu/cdaftp/byobsid/9/659/secondary/
Recursing into https://cxc.cfa.harvard.edu/cdaftp/byobsid/9/659/secondary/ephem/
Recursing into https://cxc.cfa.harvard.edu/cdaftp/byobsid/9/659/secondary/aspect/
...
とすればダウンロードが可能になる。もし、ダウンロードがうまくできなければ、ciaoのバージョンを上げてみよう。
観測データの検索ができることを確認
> find_chandra_obsid 3C123
# obsid sepn inst grat time obsdate piname target
829 0.0 ACIS-S NONE 47.0 2000-03-21 Hardcastle "3C 123"
として、OBSID情報が取得できることを確認する。
これらはpythonスクリプトで、その中身は、
less `which find_chandra_obsid`
で確認できる。#!/usr/bin/env python で python を呼び出しているため、pythonにPATHが通っていれば実行されるはず。
観測ログをquickに表示したい場合は下記の酒井さんの記事を参考にしてください。
Chandra衛星の解析の基礎
quick start guide に従えば、大体のイメージはつかめるので、最初はこのあたりの例題から試してみることをお勧め。
リプロセス
chandra_reproが万能のリプロセスのスクリプトである。ACIS, HRC, grating の有無を自動で判断し、適切に処理してくれる。
データのダウンロードから、リプロセスを行い、repro1 というディレクトリに生成物を保存するbashスクリプトを書くと次のようになる。
#!/bin/sh
for obsid in 659 # OBISD which you want
do
echo $obsid
download_chandra_obsid $obsid # download data if needed
mkdir -p $obsid/repro1
chandra_repro $obsid outdir=$obsid/repro1 # do reprocessing
done
obsidを自分の欲しいデータに変更すれば、ダウンロードからリプロセスまでやってくれる。
領域ファイルの生成
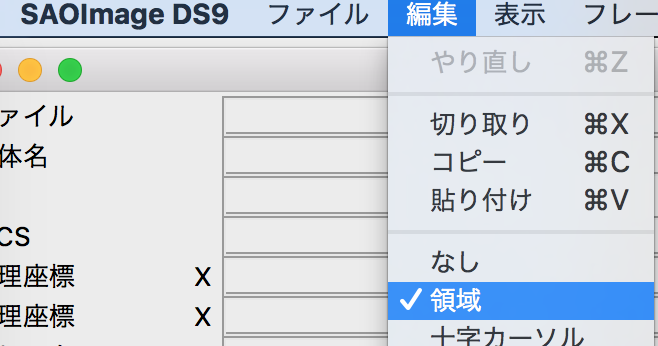
領域ファイルを生成するには、ds9の「編集」==>「領域」にして、領域の編集モードにする。
イメージの作り方
Chandraのイメージを作るには、fluximage というコマンドを用いる。
特に画像の容量が大きくて、ある場所だけのイメージが欲しい場合は、xygridというオプションをつかって画像を切り出す。簡単な例を示す。
- ACIS
fluximage evt2ファイルのあるディレクトリ ファイルに付けたい名前 bin=1 xygrid=3970.5:4100.5:1,4013.5:4143.5:1 clobber=yes
xygrid は自分がイメージを作りたい座標を入れる。下限値:上限値:ビンサイズの順番となる。最後を":1"とすると、1pixelのイメージを生成してくれる。
- HRC
fluximage evt2ファイルのあるディレクトリ ファイルに付けたい名前 bands=::1.1 bin=1 xygrid=16360.5:16560.5:1,16225.5:16425.5:1
この例では、bands=::1.1 として、1.1keVをreferenceとなるように指定している。
[注意] xselectで領域をカットしてイメージを作ることも可能であるが、set xybinsize 1 としてイメージを保存するとなぜか、4GBくらいのフルイメージになって、領域で指定してない場所まで refill されてしまう(バグ?)がある。(as of 2019.3.29)
スペクトルの生成
specextractを用いる。
src.reg(physical座標で保存されたregionファイル)という領域ファイルからスペクトルを生成したい場合は、
specextract "acisf00659_repro_evt2.fits[sky=region(src.reg)]" src659
これで自動的にレスポンスからgroupingしたスペクトルまでを生成してくれる。
ただし、これが使えるのはイメージの0次光の場合のみ。gratingの場合は下記を参照。
Grating を使った観測の場合は、chandra_reproを実行すると、tg/ というディレクトリ以下にスペクトル、rmf, arf が生成されるので、それを使うだけで良い。Grating の 0 次光のデータを使いたい場合は、円で領域をカットするなど自分でカスタマイズする必要がある。
スペクトルの足し込み方法
異なるOBSIDのイベント足す時には、
各々のOBSIDから specextract を用いてスペクトルを作成してから、combine_spectra を用いて、スペクトルを足しこむ。
イベントファイルを足しこむことは、https://cxc.cfa.harvard.edu/ciao/caveats/merged_events.html を読むとciaoではツールレベルでも推奨されていない。一般論では、同じ観測条件でない限り、イベントファイルを足しこむことは推奨されない。(観測条件がほぼ同じ時に、ftools の xselect などでイベントファイルから足しこむことに慣れてる人は少し注意しましょう。)
ライトカーブの生成
dmextractでライトカーブを抽出する。
dmextract "acisf01711_repro_evt2.fits[sky=region(src.reg)][bin time=::100]" flare.lc op=ltc1 clob+
生成されたライトカーブは lcurve でプロットする。
>lcurve
lcurve 1.0 (xronos5.22)
Number of time series for this task[1] 1
Ser. 1 filename +options (or @file of filenames +options)[flare.lc] flare.lc
Name of the window file ('-' for default window)[-] # セレクション条件(windowファイルと呼ばれる)の設定。 "-" でデフォルトを使用。
Newbin Time or negative rebinning[1000] 1000 # 時間ビン
Number of Newbins/Interval[12] INDEF # 一インターバルあたりのビン数。デフォで良い場合はINDEFとする。
Do you want to plot your results?[Y] #結果を表示したい場合
Enter PGPLOT device[/xw] # /xwで xwindowで表示される。
lcurve の interval というパラメータは、一つのライトカーブをマルッと全部表示したい(解析したい)場合もあるだろうし、長いライトカーブが入力ファイルにあって、短い時間ごとに細かく見たい場合があるときにもあるので、それを調節するためのパラメータである。例えば、time binsize を 10秒にしたとする。全ライトカーブの時間は100ksとする。Number of Newbins/Interval を 1000 にすると、10秒x1000 = 10ks で 1 interval になるので、全ライトカーブを 10個のインターバルに分割することになる。
物理座標の確認 (skipしてもよい)
ds9 でデフォルトで表示されるのはX,Yという座標で、これはSKY(空)にマップした座標系になっている。実際に検出器の座標でどういうイメージができているのかを確認するのは装置の癖をしる上では重要となる。
chandra_reproが終わった後に生成される evt2 というファイルをds9で開き、「ビンまとめ」==>「ビンまとめの設定」==> 「ビンまとめする列」でdetx, detyを選択して、適用をおすと、dext, dety という検出器座標のイメージに変更される。

のようにdetx, detyに変更すると、

このような四角のイメージに変わる。
これは、dither という技術をChandraが採用しているためである。Chandraは角度分解能がよいため、1pixelの良し悪しが観測全体に影響を及ぼしやすいために検出器を動かすことでその効果を緩和するためでもあり、パイルアップの影響を下げるためでもある。詳しく知りたい人は、chandraのditherを参考にされたい。
少し高級な解析方法
凝ったビンニング
CIAOから下記の領域ビンニングの方法については詳しく紹介されてます。
- Bin Event File to Image; Specify Bin Size
- Bin Event File to Image; Specify Number of Bins
- Bin Event File to Image With Filtering
- Bin Event File to Image With FOV Filtering
- Weighted Binning
- Adaptive Binning
- Adaptive Binning (Polar Coordinates)
- Contour Map Based Binning
- Map Based Binning: Hardness Ratio Map
- Binning Arbitrary Axes
- Alternative binning: hexagonal grid
- Alternative binning: Voronoi tessellation
- Alternative binning: centroid map
- Alternative binning: steepest ascent with pathfinder
- Alternative binning: stack of regions with mkregmap
Data Model Tools (core tools)の基礎
Chandraのciaoをインストールすると、dmから始まる Data Model Tools がインストールされる。これをcoreツールとも言う。これはおそらく今のftoolsが完成する前に設計されたため、ftoolsとの重複もある。同じことが出来るのであれば、ftoolsでもよいが、chandraの解析ツール全体がftoolsやxselectを前提として設計されてないため、後段にファイルを渡すような解析をする場合は、自力でftoolsやxselectを使うのではなくて、ciaoに準じた方が良い。
癖はそれほどなく、fitsファイルに対して、filename[block][filter][binning][option][rename] のように[]でfitsのHDU(=block)を指定し、filterをかけ、binnningでビンまとめなど、簡単にできる仕組みである。細かいことだが、unixやmacでインタラクティブに実行する場合は""がなくてよいが、一行で命令を与える場合は、"filename[...]"のように""が必要となる。
dmcopy, dmextract
Data Model tools の中でも、dmcopy, dmextract は覚えておいた方がよい。大雑把な理解としては、
- dmcopy はイベント or イメージを生成
- dmextract はライトカーブ or スペクトルを生成
である。
xselectユーザーは、xselect で filter をかけて extract event or extract image をするのが dmcopy に対応し、extract spec or extract curve に相当するのが dmextract である。下記でも登場するが、一般的な注意点として、dm ツールは、block という fits の HDU の単位でアクセスする設計のため、dmcopyで event ファイルを書き出した場合に全てのHDU(regionファイルなど)が書き出されるわけではなく、全てを書き出したい場合は option=all をつける必要がある。
HETG/ACIS-S Gratingの解析方法 (簡単な場合)
単純に平均スペクトルが欲しいだけであれば、chandra_reproを実行した後に、tg/ というディレクトリ以下にスペクトル、rmf, arf が生成されるので、それを使うだけで良い。
http://cxc.harvard.edu/ciao/threads/spectra_hetgacis/
ライトカーブは、chandra_reproで生成される acisf${obsid}_repro_evt2.fits を使えば良い。領域ファイルは、少し特殊で、
fitsの REGION コラムがdmappendによって中に保存されている。複雑なものではなく、長方形と円だけである。ds9 で evt2 ファイルを叩くと自動で領域も読み込まれる仕様である。この領域を使っても良いし、自分で生成して、ライトカーブを生成する。
HETG/ACIS-S Gratingの解析方法 (時間のカットをかけたい場合)
出発点はchandra_reproを一度走らせたあとに生成されるacisf${obsid}_repro_evt2.fits を使えば良い。mkdir で作業スペースを生成したのち、例えば、OBSID=20131の場合は、下記のスクリプトでtime1とtime2の間のスペクトルを生成することができる。(chandra_reproで生成されるディレクトリの下(tg/と等位)にいることを想定。)
#!/bin/sh
obsid=20131
time1=650561110 # need to be integer
time2=650609080 # need to be integer
tag=${time1}_${time2}
echo "\n..... start process " $obsid from $time1 to $time2
ln -fs ../acisf${obsid}_repro_evt2.fits .
echo "\n..... do timing filter. option=all is needed to keep REGION in the output fits file. "
#dmcopy "acisf${obsid}_repro_evt2.fits[time=${time1}:${time2}]" acisf${obsid}_repro_evt2_${tag}.fits option=all clobber=yes
dmcopy "acisf${obsid}_repro_evt2.fits[time=${time1}:${time2}]" acisf${obsid}_repro_evt2_${tag}.fits clobber=yes
dmappend "acisf${obsid}_repro_evt2.fits[REGION][subspace -time]" acisf${obsid}_repro_evt2_${tag}.fits
echo "\n.... created light curve for check"
dmextract "acisf${obsid}_repro_evt2_${tag}.fits[energy=500:7000][bin time=::2000]" lc_${obsid}_2ks_${tag}.lc op=ltc1 clob+
echo "\n.... create a collection of pha file. type ENTER several times for a standard analysis"
punlearn tgextract
pset tgextract infile=acisf${obsid}_repro_evt2_${tag}.fits
pset tgextract outfile=acisf${obsid}_repro_pha2_${tag}.fits
tgextract clobber=yes
ln -fs ../pcadf*_asol1.fits .
ln -fs ../acisf${obsid}_repro_bpix1.fits .
ln -fs ../acisf${obsid}_000N001_msk1.fits .
echo "\n..... create each pha files, rmf, and arf files"
mktgresp acisf${obsid}_repro_pha2_${tag}.fits acisf${obsid}_repro_evt2_${tag}.fits acis_repro_${tag} clobber=yes
- dmcopy で時間カットをかける。この時、option=all を忘れると、fitsファイルの中に保存されている領域ファイルが引き継がれず、後段でコケる。しかし、バージョンによっては、 https://cxc.harvard.edu/ciao/bugs/dmcopy.html#bug-12841 のバグがあるので、上記のようにdmcopyで一発でやるのではなくて、dmappendをすることにした。
- dmextract でライトカーブを生成し、lcurveで正しくカットされていることを確認する。
-
tgextract
でスペクトルが一式詰まったfitsファイルを生成する。 - mktgresp で回折次数ごとのスペクトル、レスポンス、arf を生成する
これで、Type2 PHA ファイルと arfとrmf は生成される。pha の source と background もそれぞれ出す方法もありそうだが、イマイチわからず。
この Type2 PHA ファイルをどうやってxspecで使うのが分からないので、最後に、
Type1 PHAの生成を行う。これは tgextractを再度実行し、
# ファイル名の区別用にtype1_を入れて、
If typeII, enter full output file name or '.'; if typeI, enter output rootname (acisf20131_repro_pha2_650561110_650609080.fits): type1_
...
# pha_typeI を選択する
Output file type: typeI (single spectrum) or typeII (multiple spectra) (pha_typeI|pha_typeII) (pha_typeII): pha_typeI
pha_typeI を選択すると、回折次数ごとのfitsのスペクトルファイルが生成され、それぞれの回折次数(m1,p1,m2,...)ごとのphaファイル、rmf, arf をxspecで読み込ませるとフィットできる。source と background の領域は fits に書かれて
いるので、何か方法はありそうだが。
一応、別の方法として、https://cxc.harvard.edu/ciao/threads/examinepha2/ によると、dmtype2split を使って、type2 pha ファイルから、type1 pha ファイルに戻して分割している。PHA2ファイルについてはhttps://cxc.harvard.edu/ciao/threads/examinepha2/index.html#what に書かれているが、PHAファイルの塊として定義しただけに思える。
足しこむ場合
combine_grating_spectra で、Chandra gratings PHA ファイルを足しこむことができます。
参考ページ
- m字光ごとのスペクトル、arf, rmf を生成 : http://cxc.harvard.edu/ciao/threads/spectra_hetgacis/
高度な画像処理
イベントファイルから画像を生成し、そのあとに高度な画像処理を行うことも多い。
まずは、イベントファイルから画像ファイルを生成する方法を確認すると、
https://cxc.cfa.harvard.edu/ciao/guides/quick_start.html の ... make exposure corrected images の例にあるように、fluximage というコマンドでイベントファイルからイメージファイルを生成できる。
$ download_chandra_obsid 635
$ chandra_repro 635 635/repro
$ fluximage 635/repro/acisf00635_repro_evt2.fits rhoOph
Gallery: Smooth に色々なイメージ解析の方法が記載あるので遊んでみよう。
Deconvolution: arestore は、Lucy-Richardson deconvolution を計算してくれる。psf_x_center、psf_y_center が意味するのは、 image coordinates であり、DS9 の "physical" coordinates が、Chandra の "sky" coordinate に対応する。
参考ページ
- CHANDRA DATA ANALYSIS MANUAL OSAKA LOCAL, 最終更新日 2008/8/31, 記述がやや古い
- Chandra/HETGの位相分割スペクトル抽出の解析ログ by 湯浅君, 最終更新日 2013/8/28, たぶんこのまま動く
- python を用いて天体画像(Chandra, XMM etc.)から radial profile を作成する方法