「なんでもは知らないわよ。知ってることだけ。1」
これはR Advent Calendar2018の第一日目の記事です。"tidyverse"という言葉がUser!2016で爆誕してから早くも2年が経ちました。皆さんは普段どれだけtidyverseのパッケージを使って作業をしているでしょうか。
今日は、日々進化を続けるtidyverseに含まれるコアパッケージの、(私的に)あまり知られていない関数を紹介したいと思います。なお各パッケージのバージョンは全て現在CRANに登録されている最新版となります。
「あなたの好きなtidyverseの関数を教えてください!」のコーナーだ(謎)!! slice()わいも好き。 #tokyor
— Uryu Shinya (@u_ribo) October 20, 2018
皆さんの好きなtidyverseの関数は何ですか??
それでは行きます。
library(tidyverse)
## ─ Attaching packages ───────────────────────────────── tidyverse 1.2.1 ─
## ✔ ggplot2 3.1.0 ✔ purrr 0.2.5
## ✔ tibble 1.4.2 ✔ dplyr 0.7.8
## ✔ tidyr 0.8.2 ✔ stringr 1.3.1
## ✔ readr 1.2.1 ✔ forcats 0.3.0
## ─ Conflicts ─────────────────────────────────── tidyverse_conflicts() ─
## ✖ dplyr::filter() masks stats::filter()
## ✖ dplyr::lag() masks stats::lag()
ggplot2
NEWSというよりはtips的な紹介です。
gghighlightを使わないハイライト
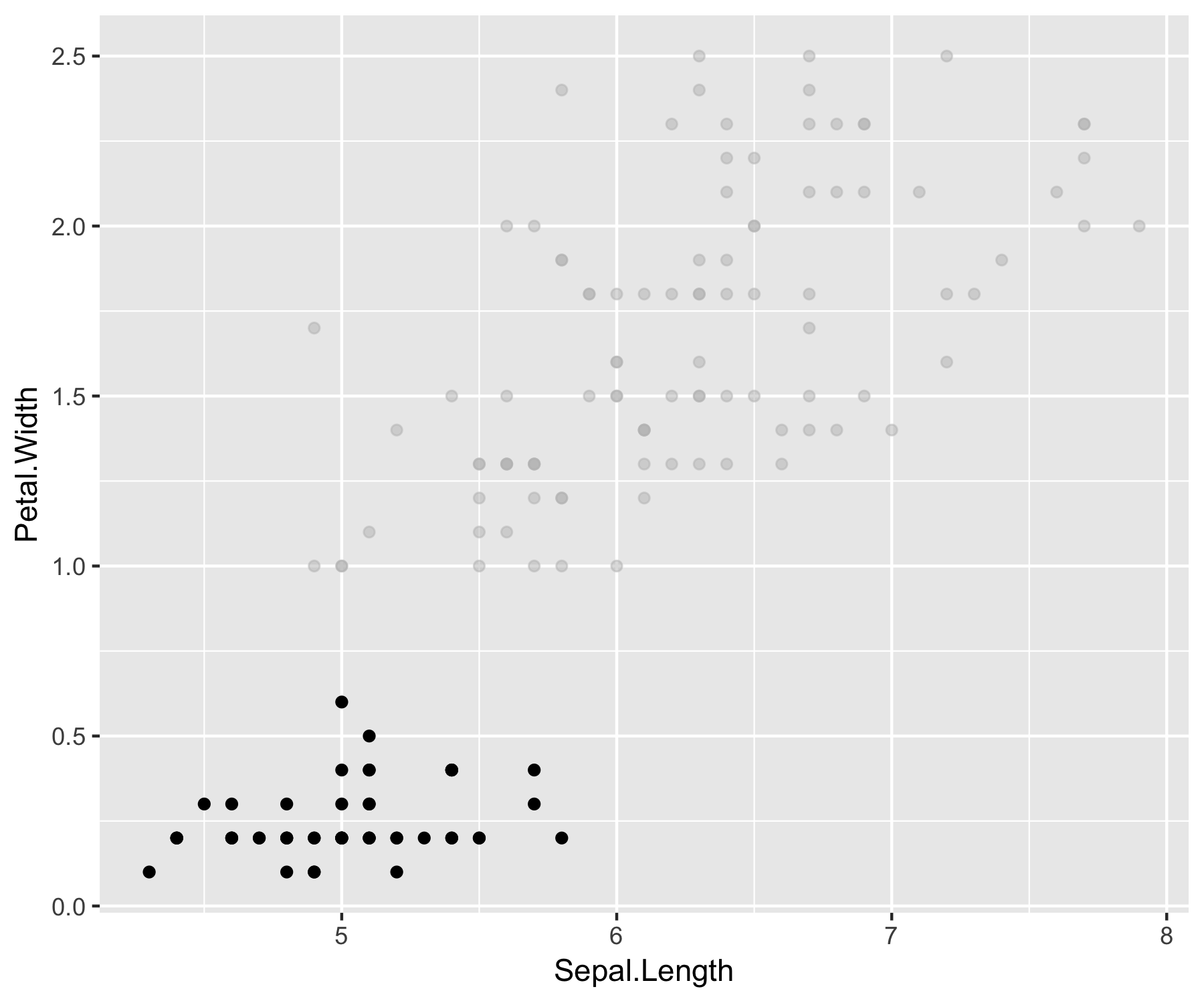
パイプ演算子を無名関数として利用することで、ハイライトのための用意を整えます。実際のデータの選択・抽出はgeom_*(data = )で行います。
例えば、gghighlightを使ってirisのSpecies == “setosa”をハイライトするには次のようにします。
library(gghighlight)
iris %>%
ggplot() +
geom_point(aes(Sepal.Length, Petal.Width)) +
gghighlight(Species == "setosa")
これをパイプ処理で再現します。
iris %>% {
ggplot(data = .,
aes(Sepal.Length, Petal.Width)) +
geom_point(data = . %>%
dplyr::filter(Species == "setosa"),
color = "black") +
geom_point(data = . %>%
dplyr::filter(Species != "setosa"),
color = "gray",
alpha = 0.5) +
guides(color = FALSE)
}
冗長ですが、細かな部分の調整やgghighlightで対応できない処理(があるかもしれない)があった時に覚えておくと便利です。
複数の図をまとめて表示する際に便利なタグ
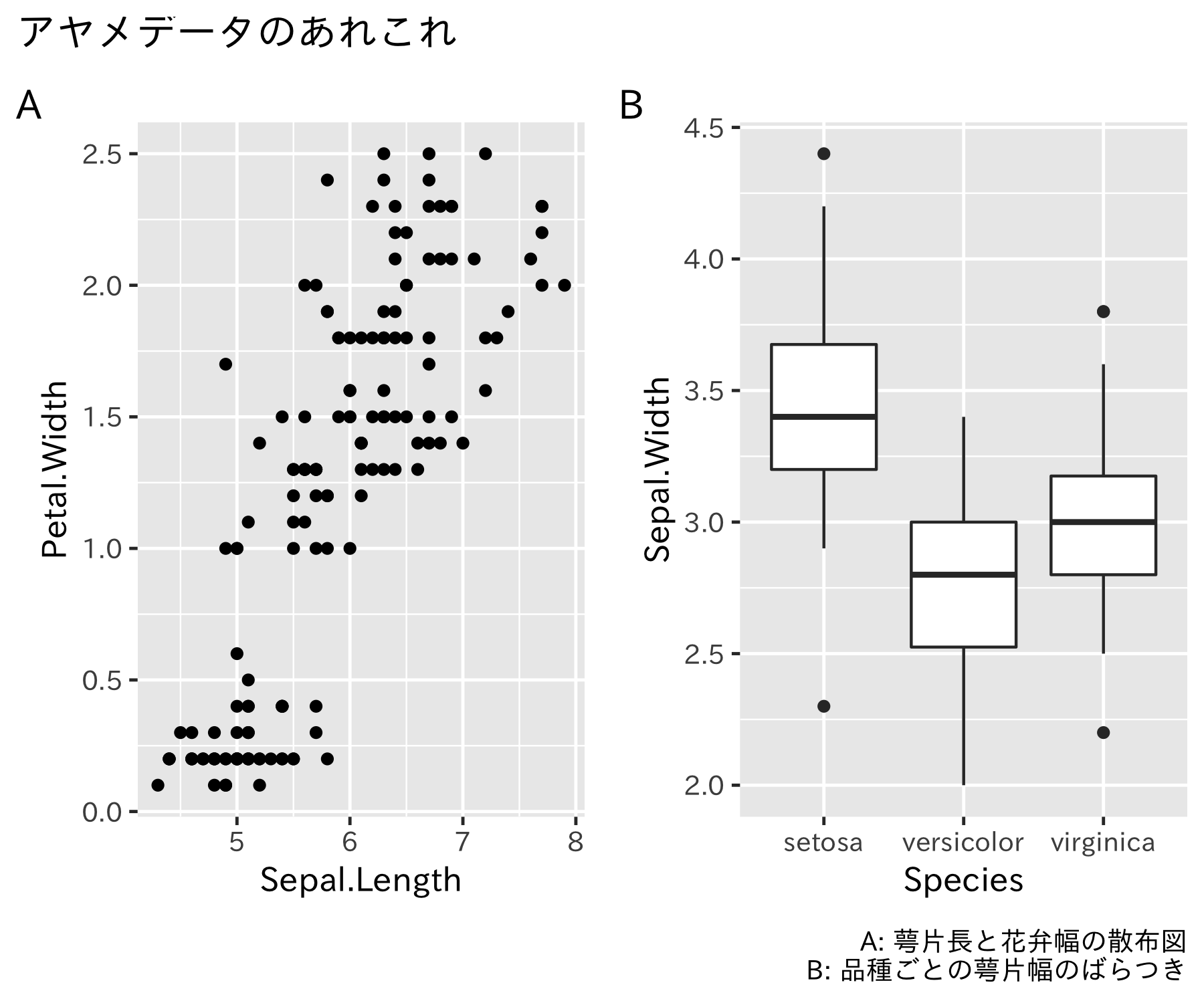
ggplot2には、図の説明用にtitle, subtitle, captionが用意されていることは知っていましたが、tagの存在に気が付いたのは比較的最近です。ggtitle()のような専用関数は用意されておらず、labs(tag = )で指定します。
library(patchwork)
p1 <-
iris %>%
ggplot(aes(Sepal.Length, Petal.Width)) +
geom_point() +
labs(tag = "A")
p2 <-
iris %>%
ggplot(aes(Species, Sepal.Width)) +
geom_boxplot() +
labs(tags = "B")
theme_set(theme_gray(base_size = 12, base_family = "IPAexGothic"))
p1 + p2 +
plot_layout(ncol = 2) +
plot_annotation(title = "アヤメデータのあれこれ",
caption = "A: 萼片長と花弁幅の散布図\nB: 品種ごとの萼片幅のばらつき")
patchwork パッケージの解説は @Atsushi776
さんによる資料に詳しい説明があります。そちらも要チェックです。
最後に描画したggplot2の図を復元する
大きな声では言えませんが
これも@Atsushi776さんに教えてもらいました。
直前に描画したggplot2での図をもう一度描画するために.Last.valueを使っていましたが、これあと別の処理を挟んだ後に復元することはできません。last_plot()を使いましょう。
# 作図デバイスを初期化する
dev.off()
# 先ほどの図をもう一度描画
last_plot()
これは確認のためにプロットして、ggsave()で保存する時などに便利です。
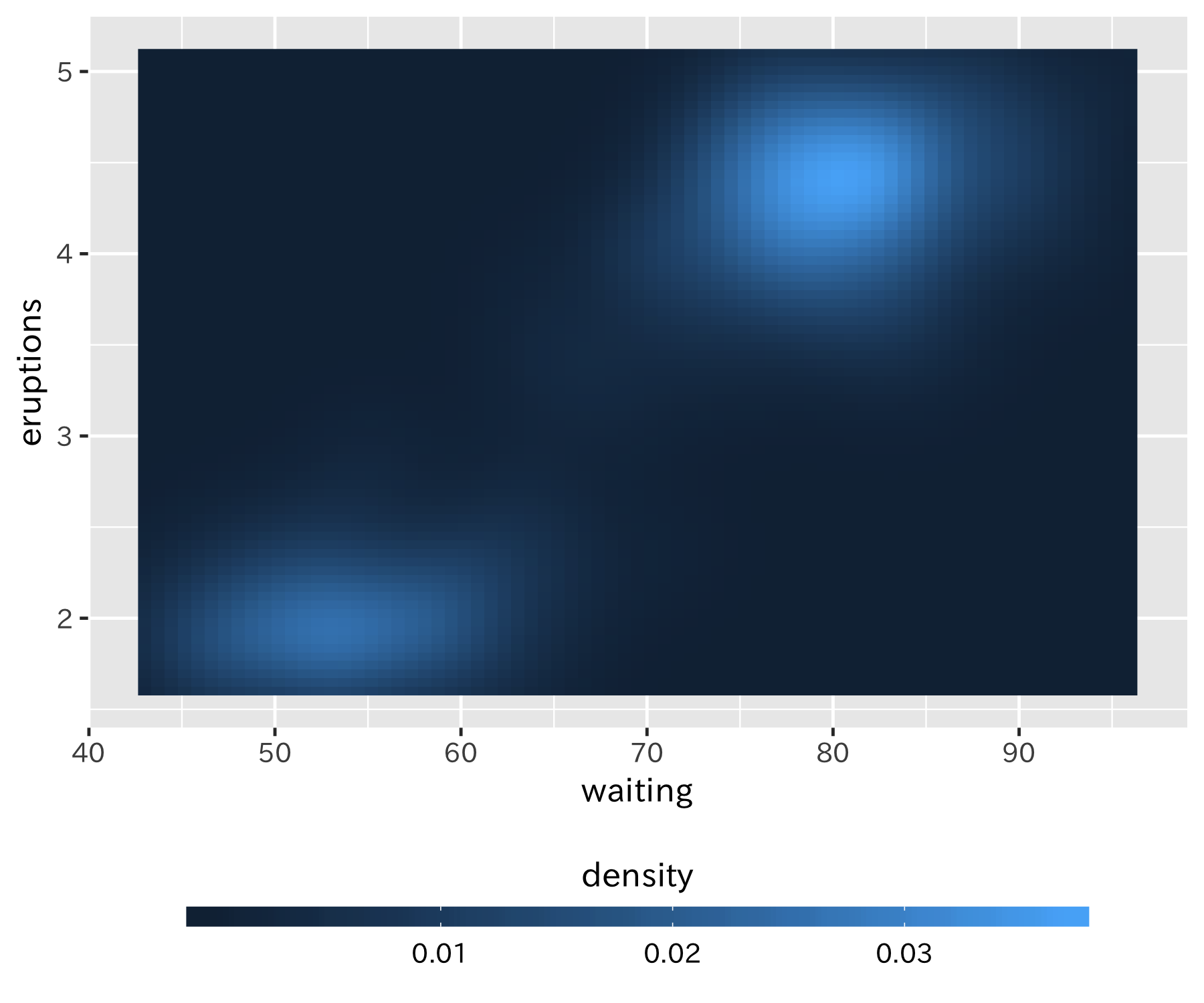
scaleの凡例を極める
連続値の凡例を横長に配置したり、長さをグラフの横の幅に合わせたりといった処理です。guides()およびtheme()に用意されている引数を適切に指定して次のような図が作れます。
Today's my #ggplot2 tips📈Adjust the position and size of the legend. Data are based on the 2015 census of the Ministry of Internal Affairs and Communications statistics Bureau small area population. pic.twitter.com/zGu00zOxyf
— Uryu Shinya (@u_ribo) November 21, 2018
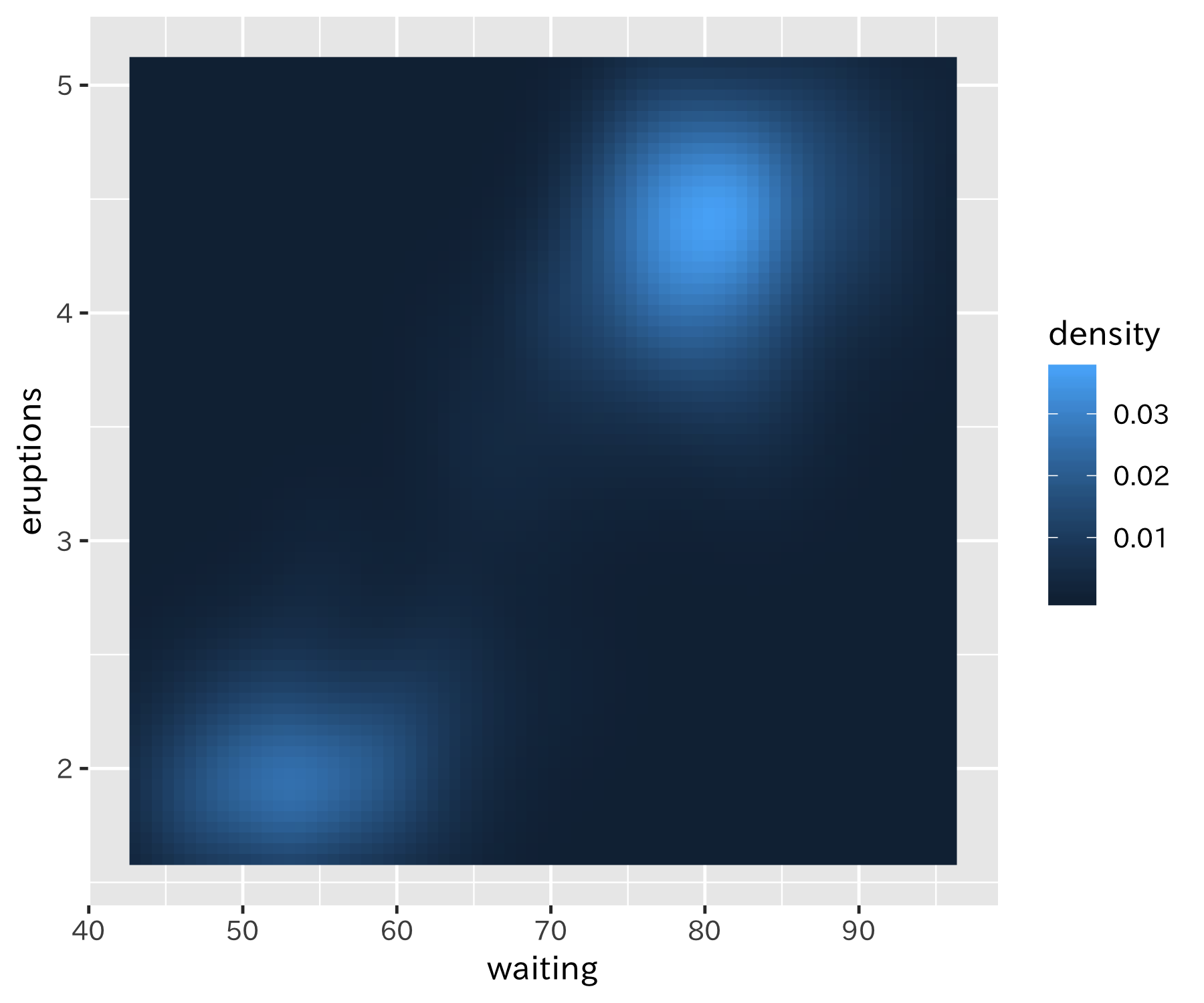
p <-
faithfuld %>%
ggplot(aes(waiting, eruptions)) +
geom_raster(aes(fill = density))
p
p +
guides(fill = guide_colorbar(title.position = "top",
title.hjust = 0.5)) +
theme(legend.position = "bottom",
legend.key.height = unit(0.5, "line"),
legend.key.width = unit(4.5 ,"line"))
tibble
データフレーム (data.frame)の使い勝手を改善したtbl_df(tibble data.frame)というオブジェクトのクラスを提供するパッケージです。そのため、tbl_dfとして実装するパッケージの内部で使われることが目立ちますが、データフレームに関するいくつかの処理も備えています2。
また、次期バージョンに関して @yutannihilationサンから注意勧告があります。
tibbleパッケージの次期リリースの変更点、as_tibble()がヤバ目なので確認したほうがよさげ。
— Hiroaki Yutani (@yutannihilation) November 29, 2018
・列名がついていないとエラーに(列名のない行列とか)
・列名が重複しているとエラーにhttps://t.co/QWRDOUwIxI
データフレームオブジェクトの作成
説明の時など、単純なデータフレームを作る際に重宝する関数です。
tibbleにはdata.frame()同様にベクトルでデータフレームの列を構築するdata_frame()がありますが、もう一つ、データフレーム作成用の関数、tribble()があります。tribble()(frame_data()という名前から変更されました)は、データフレームを記述的に作成します。一つ一つの値を入力するのでサイズの大きなデータフレームを作るのには向きませんが、一行ずつデータフレームを作っていく形式になっているのでうっかり桁数の揃わないデータを作ってしまうことを防ぐのに役立ちます。
tribble()では列名を~で表現し、引数の最初に与えます。~をつけた値が列名として使われます。列名、値はカンマで区切ります。次のように、データフレームの構造を意識しながらデータを作れます。
tribble(
~id, ~value,
"a", 1,
"b", 2)
## # A tibble: 2 x 2
## id value
## <chr> <dbl>
## 1 a 1
## 2 b 2
# こうした記述も可能ですが、tribble()のありがたみが薄れる気がします
tribble(
~id,
~value,
"a",
1,
"b",
2)
また、より単純なデータフレームを作る場合は、任意のベクトルからデータフレームを作成するenframe()が役立ちます。この関数は引数にベクトルを与えて実行します。すると、nameとvalueという2列のデータフレームが生成され、ベクトルの要素はvalue列に格納されます。nameには1から始まる正の自然数が与えられます。
seq_len(3) %>%
enframe()
## # A tibble: 3 x 2
## name value
## <int> <int>
## 1 1 1
## 2 2 2
## 3 3 3
生成されるデータフレームの列名は引数で指定できます。また、ベクトルの要素に名前が与えられている際にはそれがname列の値に使われます。
seq_len(3) %>%
enframe(name = "id",
value = "my_var")
## # A tibble: 3 x 2
## id my_var
## <int> <int>
## 1 1 1
## 2 2 2
## 3 3 3
x <-
seq_len(3) %>%
set_names(c("a", NA_character_, "c"))
enframe(x)
## # A tibble: 3 x 2
## name value
## <chr> <int>
## 1 a 1
## 2 <NA> 2
## 3 c 3
データフレームへの列・行の追加
一行ないし一列だけデータを追加したい…。そんなことが多々あります。これらの処理は標準関数を使っても可能ですが、パイプ処理の連結処理を中断させてしまいます。また任意の位置への挿入は困難です。add_row()、add_column()は、パイプ処理で記述可能な、位置指定によるデータの挿入を行う関数となります。
df <-
data_frame(
id = seq_len(3),
my_var = letters[seq_len(3)]
)
df
## # A tibble: 3 x 2
## id my_var
## <int> <chr>
## 1 1 a
## 2 2 b
## 3 3 c
このデータフレームに一行追加します。引数に列と値の組み合わせを与えて実行します。
df %>%
add_row(id = 4)
## # A tibble: 4 x 2
## id my_var
## <dbl> <chr>
## 1 1 a
## 2 2 b
## 3 3 c
## 4 4 <NA>
ベクトルの長さに応じて、追加する行は増やせます。
df %>%
add_row(id = c(4, 5), my_var = c("d", "e"))
## # A tibble: 5 x 2
## id my_var
## <dbl> <chr>
## 1 1 a
## 2 2 b
## 3 3 c
## 4 4 d
## 5 5 e
add_row()、add_column()には引数.before、.afterがあり、ここに挿入する位置を数値で指定することで、データフレーム内の自由な位置へデータを追加できます。
df %>%
add_row(id = 0, .before = 1)
## # A tibble: 4 x 2
## id my_var
## <dbl> <chr>
## 1 0 <NA>
## 2 1 a
## 3 2 b
## 4 3 c
df %>%
add_row(id = 2.5, .after = 2)
## # A tibble: 4 x 2
## id my_var
## <dbl> <chr>
## 1 1 a
## 2 2 b
## 3 2.5 <NA>
## 4 3 c
df %>%
add_column(new_var = c("あ", "い", "う"), .before = "my_var")
## # A tibble: 3 x 3
## id new_var my_var
## <int> <chr> <chr>
## 1 1 あ a
## 2 2 い b
## 3 3 う c
tbl_dfを継承するオブジェクトクラスの構築
上述の通り、tbl_dfはデータフレームの拡張です。as_tibble()を使って変換が可能です。一方で、同じくデータフレームを拡張したsfやtibbletimeクラスのオブジェクトに対してこの処理を適用するとクラスが上書きされてしまいます。
nc <-
system.file("shape/nc.shp", package = "sf") %>%
sf::st_read(quiet = TRUE)
class(nc)
## [1] "sf" "data.frame"
nc %>%
as_tibble() %>%
class()
## [1] "tbl_df" "tbl" "data.frame"
あら不思議、sfクラスではなくなってしまいます。しかし、オブジェクトを出力した際に全件出力されるのは煩わしい…。そんな時にnew_tibble()が役立ちます。
nc %>%
new_tibble(subclass = "sf") %>%
class()
## [1] "sf" "tbl_df" "tbl" "data.frame"
なおsfパッケージのデータ読み込み関数st_read()にはtibble形式への変換を行うas_tibbleオプションが備わっており、st_read(as_tibble = TRUEとしておくことで上記の処理が不要になります。
nc <-
system.file("shape/nc.shp", package = "sf") %>%
sf::st_read(quiet = TRUE,
as_tibble = TRUE)
class(nc)
## [1] "sf" "tbl_df" "tbl" "data.frame"
tidyr
データフレームをRで処理しやすいtidy形式へと変換するのがtidyrの主な機能です。dplyrがデータフレームの列や行の値を操作するのに対して、データフレーム全体に影響する関数が多いのが特徴です。
ここにない他の関数の挙動については@kazutanの記事にまとめられています。
カウント値を行に反映させる
特定の列の値をカウントした値をもつ列に対して、uncount()を実行するとカウント値の分だけ行が複製されたデータを作れます。例えば次のように“a”、“b”、“c”からなる値をx列に、それぞれの出現頻度をn列に記録したデータを用意し、これにuncount()を実行します。するとnの数に応じてx列の要素が複製されます。
df <-
data_frame(
x = c("a", "b", "c", "a", "b", "b", "a")
) %>%
count(x)
df
## # A tibble: 3 x 2
## x n
## <chr> <int>
## 1 a 3
## 2 b 3
## 3 c 1
df %>%
uncount(n)
## # A tibble: 7 x 1
## x
## <chr>
## 1 a
## 2 a
## 3 a
## 4 b
## 5 b
## 6 b
## 7 c
例で示したようにdplyr::count()した後の処理として使うことがありそうです。
欠損値を含む行を除外する
drop_na()は、欠損値を含む行をデータから除外するのに役立ちます。
df <-
tibble(x = c(1, 2, NA), y = c("a", NA, "b"))
df %>%
drop_na()
## # A tibble: 1 x 2
## x y
## <dbl> <chr>
## 1 1 a
欠損値が含まれた際に除外対象とする列を引数で指定可能です。
df %>%
drop_na(x)
## # A tibble: 2 x 2
## x y
## <dbl> <chr>
## 1 1 a
## 2 2 <NA>
この他、あまり知られていないと感じる関数にseparate_rows()がありますが、これについては以前記事を書いていますので、そちらを参照いただければと思います。
dplyr
データ操作に欠かせないdplyrパッケージの関数は、@matsuou1 さんの一連の記事にまとめられているもの使うことがほとんどですが、ちょくちょく追加がありますので紹介します。
データフレームの列を参照
パイプ処理の中で列を参照するためにmagrittr::use_series()を使っていましたが、dplyr::pull()で同様の処理ができます。
iris %>%
dplyr::pull(Species) %>%
levels()
## [1] "setosa" "versicolor" "virginica"
# magrittr::use_series()を使う場合... 同じ結果です
# iris %>%
# magrittr::use_series(Species) %>%
# levels()
データの一部を位置指定で取り出す
head()(先頭)やtail()(末尾)とは異なり、任意の位置でデータフレームの一部を参照するためにslice()を使います。引数に行番号を指定します。
df <-
data_frame(
x = seq_len(3))
df %>%
slice(2)
## # A tibble: 1 x 1
## x
## <int>
## 1 2
df %>%
slice(c(1, 3))
## # A tibble: 2 x 1
## x
## <int>
## 1 1
## 2 3
カウント値を列に追加する
出現頻度をかぞえあげる関数にはcount()、tally()が使えますが、add_count()およびadd_tall()は、対象のデータフレームの構造を維持したまま、列を追加する形でカウントを行います。
mtcars %>%
count(cyl)
## # A tibble: 3 x 2
## cyl n
## <dbl> <int>
## 1 4 11
## 2 6 7
## 3 8 14
mtcars %>%
select(mpg, cyl) %>%
add_count(cyl)
## # A tibble: 32 x 3
## mpg cyl n
## <dbl> <dbl> <int>
## 1 21 6 7
## 2 21 6 7
## 3 22.8 4 11
## 4 21.4 6 7
## 5 18.7 8 14
## 6 18.1 6 7
## 7 14.3 8 14
## 8 24.4 4 11
## 9 22.8 4 11
## 10 19.2 6 7
## # ... with 22 more rows
今日は国内Rイベントにおいて最大規模を誇るJapan.Rの開催日です。私も会場手伝いとして参加します。ぜひ会場でお会いしましょう。
明日は@utaka233の記事です。お楽しみに!それでは、Enjoy!