Pythonで2要因混合計画の分散分析を行います。
2要因混合計画自体の説明は割愛します。
勉強中のためもし間違いなどありましたらコメントで教えていただけますと幸いです。
pingouin
pingouinを使用すると簡単に2要因混合計画の分散分析を行うことができます。もちろん他の分析もできます。
インストール
pip install pingouin
使い方
データセットの形式
データセットは、Pandasのデータフレームが対応しています。
例としてサンプルデータセットを読み込みます。mixed_anovaはpingouinが提供するサンプルデータセットです。
import pingouin as pg
df = pg.read_dataset('mixed_anova')
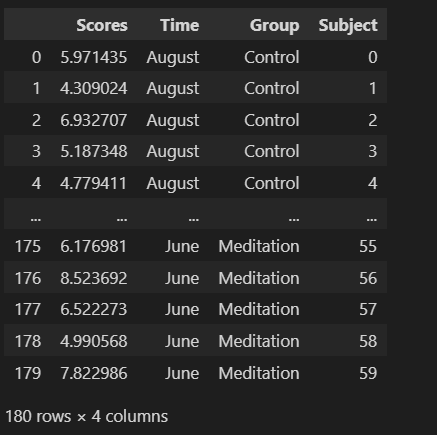
df
読み込まれたデータフレームの列名は、左からScores、Time、Group、Subjectです。Timeは3水準、Groupは2水準、Subjectは被験者番号で重複セルがあります。
pingouinでは列名を指定するので、列の順序は関係ないと思います。
形式をまとめると以下の通りです。被験者内要因の水準ごとに1からn番目の被験者が順番に並んでいます。
| 被験者番号 | 被験者間要因 | 被験者内要因 | 従属変数 (dv) |
|---|---|---|---|
| 1番 | A | 1 | 3 (何らかの値) |
| 2番 | B | 上のセルと同じ値 (以降、〃) |
6 |
| ... | ... | 〃 | ... |
| n番 | B | 〃 | 8 |
| 1番 | A (被験者1番の他の行と同じ値) |
2 | 4 |
| 2番 | B (被験者2番の他の行と同じ値) |
〃 | 7 |
| ... | ... | 〃 | ... |
| n番... | B (他の被験者n番の行と同じ値) |
〃 | 9 |
| --- | 以下同じパターン | --- | --- |
分析
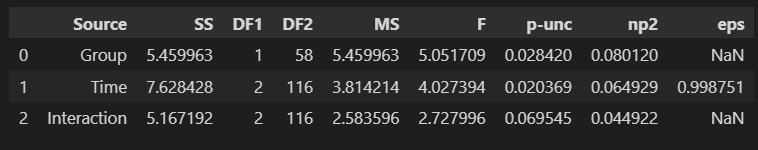
mixed_anovaの返り値が分散分析表です。形式はデータフレームです。
aov = pg.mixed_anova(data=df, dv='Scores', between='Group', within='Time',
subject='Subject', correction=False, effsize="np2")
aov
引数は、
- data :データフレーム
- dv : 従属変数の列名
- between : 被験者間要因の列名
- within : 被験者内要因の列名
- subject : 被験者番号の列名
- correction : Greenhouse-Geisser補正をするかどうか
- "auto" : デフォルト値。Mauchlyの球面性検定を行い、p値を修正するか判断される。
- True : する
- False : しない
- effsize :
- "np2" : 偏イータ二乗 デフォルト値。
- "n2" : イータ二乗
- "ng2" : 一般化イータ二乗
実行結果
他の方法でもやってみる
pingouinの記事が少なく、実行結果を信用していいのか不安だったので同じような結果になるか別の方法で確認します。
参考
次の記事に従って実行します。
余談
R_HOME環境変数があればrpy2ならRのパスを指定しないで実行可能です(後述)。
そしてrpy2をインポートした後ならpyperもRCMD引数でパスを指定する必要がありません(???)
データフレーム整形
対応している形式が違うのでデータフレームを変形します。
df.reindex(columns=['Subject', 'Group', 'Time','Scores'])
grouped = df.groupby(['Subject','Group'])['Scores'].apply(list).reset_index()
max_len = grouped['Scores'].str.len().max()
for i in range(max_len):
grouped[f'Scores{i+1}'] = grouped['Scores'].apply(lambda x: x[i] if i < len(x) else None)
grouped.drop(columns=['Subject','Scores'], inplace=True)# Subject列と最初のリスト列(Scores)を削除
grouped
groupedはデータフレーム形式です↓。列の順序は後の分析に影響します。
| 被験者間要因A | 被験者内要因Bの 水準1 |
被験者内要因Bの 水準2 |
|---|---|---|
| A | 3 (何らかの値) |
4 |
| B | 6 | 7 |
| ... | ... | ... |
| B | 8 | 9 |
行数はn(被験者数)で、横方向に被験者内要因の水準が追加されます。また、被験者番号用の列は必要ありません。
分析
#
# anovakunを実行するまでは、参考記事と同じ。
#
result = r('anovakun(df, "AsB", 2, 3)')
print(result)
実行結果
AがGroupでBがTimeです。四捨五入されてはいますが、SS、MS、F、p値が一致していると思います。df1とdf2が同じdf列内にありますが、df1がA、B、AxB行、df2がsxA、sxAxB行なので一致しています。
他参考
- http://riseki.php.xdomain.jp/index.php?ANOVA%E5%90%9B
- http://riseki.php.xdomain.jp/index.php?ANOVA%E5%90%9B/ANOVA%E5%90%9B%E3%81%AE%E4%BD%BF%E3%81%84%E6%96%B9
rpy2の場合
pyperではなくrpy2を使う場合
from rpy2.robjects.conversion import localconverter
from rpy2.robjects.conversion import py2rpy
import rpy2.robjects as robjects
from rpy2.robjects import pandas2ri
with localconverter(robjects.default_converter + pandas2ri.converter):
r_df = py2rpy(grouped)
robjects.r.assign("df", r_df)
robjects.r("df")
robjects.r('source(file="D:/anovakun_489.txt")')
robjects.r('anovakun(df, "AsB", 2, 3)')