この記事はEnigmo Advent Calendar 2018の10日目です。
はじめに
OptunaはPFN社が公開したハイパーパラメータ自動最適化フレームワークです。
目的関数さえ決めれば、直感的に最適化を走らせることが可能のようです。
今回、最適化自体の説明は割愛させていただきますが、機械学習の入門ということを考えるとハイパーパラメータの調整としては、gridsearchやRandomizedSearchCVで行う機会が多いと思います。
スキル、あるいはリソースでなんとかするということになるかと思いますが、特に、kaggleのような0.X%の精度が向上が重要になるような状況では、ハイパーパラメータのチューニングが大きなハードルの一つになります。
そこで、titanicでのsubmitはあるものの、Kaggleの経験がほぼゼロな筆者でも、Optunaで簡単にチューニングができるかどうかを試してみようと思います。
今回の対象コンペ
既にcloseしているコンペの中で、下記のPorto Seguro’s Safe Driver Predictionを選びました。
https://www.kaggle.com/c/porto-seguro-safe-driver-prediction
選定理由は以下の通りです。
- データがそれほど大きくない
- 手元(自宅)のラップトップのRAMは8GBと大きくないので、XGboostではなくメモリ消費が抑えられるLightGBMでやってみたい
- 解法がシンプルかつ、LightGBMで上位のスコアを解法を公開しているカーネルがすぐに見つかった
公開解法の再現
上記をそのままコピペして一回submitします。
Python2対応のようなので、下記のようにPython3で動くように修正しました。
# part of 2nd place solution: lightgbm model with private score 0.29124 and public lb score 0.28555
import lightgbm as lgbm
from scipy import sparse as ssp
from sklearn.model_selection import StratifiedKFold
import numpy as np
import pandas as pd
from sklearn.preprocessing import LabelEncoder
from sklearn.preprocessing import OneHotEncoder
def Gini(y_true, y_pred):
# check and get number of samples
assert y_true.shape == y_pred.shape
n_samples = y_true.shape[0]
# sort rows on prediction column
# (from largest to smallest)
arr = np.array([y_true, y_pred]).transpose()
true_order = arr[arr[:, 0].argsort()][::-1, 0]
pred_order = arr[arr[:, 1].argsort()][::-1, 0]
# get Lorenz curves
L_true = np.cumsum(true_order) * 1. / np.sum(true_order)
L_pred = np.cumsum(pred_order) * 1. / np.sum(pred_order)
L_ones = np.linspace(1 / n_samples, 1, n_samples)
# get Gini coefficients (area between curves)
G_true = np.sum(L_ones - L_true)
G_pred = np.sum(L_ones - L_pred)
# normalize to true Gini coefficient
return G_pred * 1. / G_true
cv_only = True
save_cv = True
full_train = False
def evalerror(preds, dtrain):
labels = dtrain.get_label()
return 'gini', Gini(labels, preds), True
path = "input/"
train = pd.read_csv(path+'train.csv')
train_label = train['target']
train_id = train['id']
test = pd.read_csv(path+'test.csv')
test_id = test['id']
NFOLDS = 5
kfold = StratifiedKFold(n_splits=NFOLDS, shuffle=True, random_state=218)
y = train['target'].values
drop_feature = [
'id',
'target'
]
X = train.drop(drop_feature,axis=1)
feature_names = X.columns.tolist()
cat_features = [c for c in feature_names if ('cat' in c and 'count' not in c)]
num_features = [c for c in feature_names if ('cat' not in c and 'calc' not in c)]
train['missing'] = (train==-1).sum(axis=1).astype(float)
test['missing'] = (test==-1).sum(axis=1).astype(float)
num_features.append('missing')
for c in cat_features:
le = LabelEncoder()
le.fit(train[c])
train[c] = le.transform(train[c])
test[c] = le.transform(test[c])
enc = OneHotEncoder(categories='auto')
enc.fit(train[cat_features])
X_cat = enc.transform(train[cat_features])
X_t_cat = enc.transform(test[cat_features])
ind_features = [c for c in feature_names if 'ind' in c]
count=0
for c in ind_features:
if count==0:
train['new_ind'] = train[c].astype(str)+'_'
test['new_ind'] = test[c].astype(str)+'_'
count+=1
else:
train['new_ind'] += train[c].astype(str)+'_'
test['new_ind'] += test[c].astype(str)+'_'
cat_count_features = []
for c in cat_features+['new_ind']:
d = pd.concat([train[c],test[c]]).value_counts().to_dict()
train['%s_count'%c] = train[c].apply(lambda x:d.get(x,0))
test['%s_count'%c] = test[c].apply(lambda x:d.get(x,0))
cat_count_features.append('%s_count'%c)
train_list = [train[num_features+cat_count_features].values,X_cat,]
test_list = [test[num_features+cat_count_features].values,X_t_cat,]
X = ssp.hstack(train_list).tocsr()
X_test = ssp.hstack(test_list).tocsr()
learning_rate = 0.1
num_leaves = 15
min_data_in_leaf = 2000
feature_fraction = 0.6
num_boost_round = 10000
params = {"objective": "binary",
"boosting_type": "gbdt",
"learning_rate": learning_rate,
"num_leaves": num_leaves,
"max_bin": 256,
"feature_fraction": feature_fraction,
"verbosity": 0,
"drop_rate": 0.1,
"is_unbalance": False,
"max_drop": 50,
"min_child_samples": 10,
"min_child_weight": 150,
"min_split_gain": 0,
"subsample": 0.9
}
x_score = []
final_cv_train = np.zeros(len(train_label))
final_cv_pred = np.zeros(len(test_id))
for s in range(16):
cv_train = np.zeros(len(train_label))
cv_pred = np.zeros(len(test_id))
params['seed'] = s
if cv_only:
kf = kfold.split(X, train_label)
best_trees = []
fold_scores = []
for i, (train_fold, validate) in enumerate(kf):
X_train, X_validate, label_train, label_validate = \
X[train_fold, :], X[validate, :], train_label[train_fold], train_label[validate]
dtrain = lgbm.Dataset(X_train, label_train)
dvalid = lgbm.Dataset(X_validate, label_validate, reference=dtrain)
bst = lgbm.train(params, dtrain, num_boost_round, valid_sets=dvalid, feval=evalerror, verbose_eval=100,
early_stopping_rounds=100, )
best_trees.append(bst.best_iteration)
cv_pred += bst.predict(X_test, num_iteration=bst.best_iteration)
cv_train[validate] += bst.predict(X_validate)
score = Gini(label_validate, cv_train[validate])
print(score)
fold_scores.append(score)
cv_pred /= NFOLDS
final_cv_train += cv_train
final_cv_pred += cv_pred
print("cv score:")
print(Gini(train_label, cv_train))
print("current score:", Gini(train_label, final_cv_train / (s + 1.)), s+1)
print(fold_scores)
print(best_trees, np.mean(best_trees))
x_score.append(Gini(train_label, cv_train))
print(x_score)
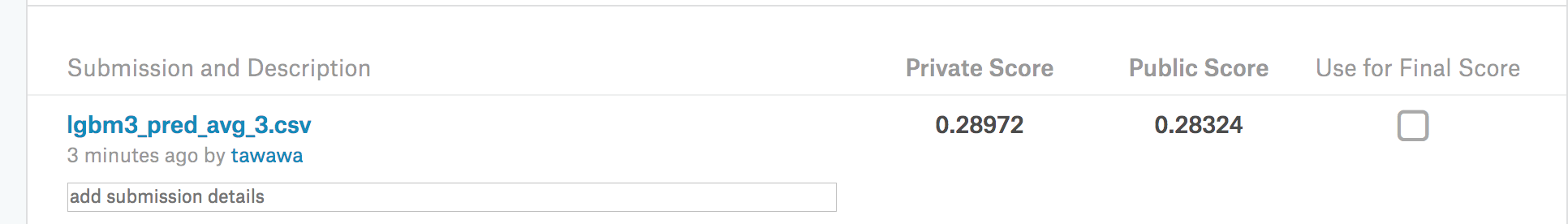
pd.DataFrame({'id': test_id, 'target': final_cv_pred / 16.}).to_csv('model/lgbm3_pred_avg.csv', index=False)
pd.DataFrame({'id': train_id, 'target': final_cv_train / 16.}).to_csv('model/lgbm3_cv_avg.csv', index=False)
公開解法でのsubmit
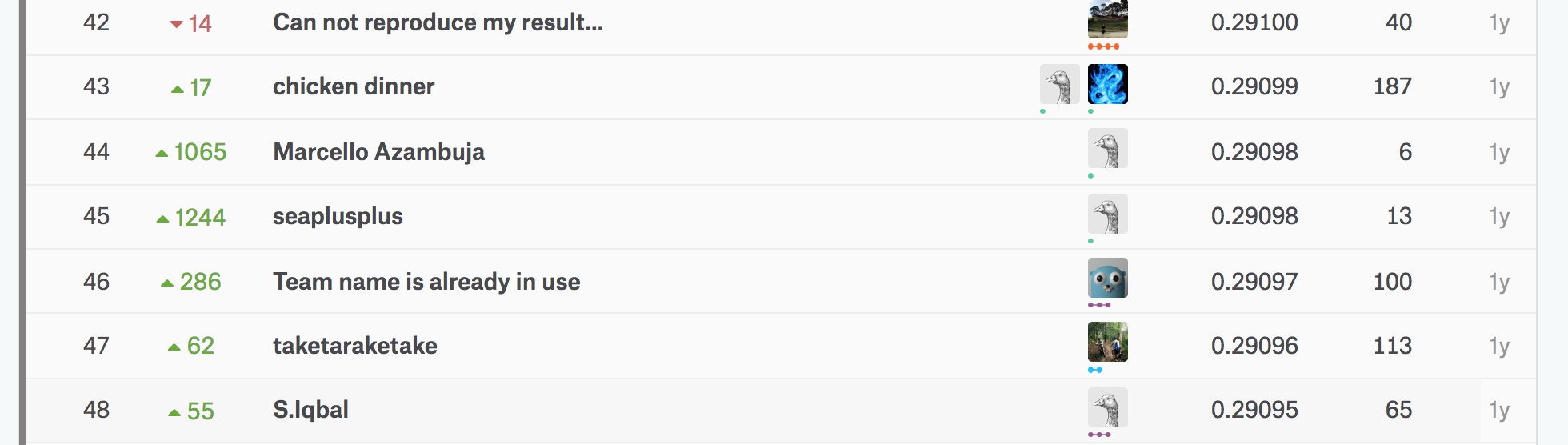
Private Scoreで0.29097。5169チーム中46位のスコアとなり、シルバーメダル圏内に入りました。
コンペは終了しているので、もちろんスコアボードの本体は更新はされません。
なお、実際のコンペでは、カーネルの著書から他のNeral Networkでの予測値の平均と記載があるので、2位のsubmitの再現というわけにならないようです。
しかし、このようなシンプルな方法でシルバーメダルのスコアを取れるのは、個人的にもKaggleに積極してみたいという励みになったと感じています。
ハイパーパラメータのチューニング
さて、ハイパーパラメータのチューニングをフレームワークの力を借りて、ハードルをぐっと下げようという、本題に移ります。
他のKaggleのコンペや、Stack over flowで雑に調査し、パラメータの範囲を決めました。
そうしてできた修正したソースコードが、以下のようになります。
import lightgbm as lgbm
import optuna
from scipy import sparse as ssp
from sklearn.model_selection import StratifiedKFold
import numpy as np
import pandas as pd
from sklearn.preprocessing import LabelEncoder
from sklearn.preprocessing import OneHotEncoder
def Gini(y_true, y_pred):
# check and get number of samples
assert y_true.shape == y_pred.shape
n_samples = y_true.shape[0]
# sort rows on prediction column
# (from largest to smallest)
arr = np.array([y_true, y_pred]).transpose()
true_order = arr[arr[:, 0].argsort()][::-1, 0]
pred_order = arr[arr[:, 1].argsort()][::-1, 0]
# get Lorenz curves
L_true = np.cumsum(true_order) * 1. / np.sum(true_order)
L_pred = np.cumsum(pred_order) * 1. / np.sum(pred_order)
L_ones = np.linspace(1 / n_samples, 1, n_samples)
# get Gini coefficients (area between curves)
G_true = np.sum(L_ones - L_true)
G_pred = np.sum(L_ones - L_pred)
# normalize to true Gini coefficient
return G_pred * 1. / G_true
cv_only = True
save_cv = True
full_train = False
def evalerror(preds, dtrain):
labels = dtrain.get_label()
return 'gini', Gini(labels, preds), True
path = "input/"
train = pd.read_csv(path+'train.csv')
# train = train.sample(frac=0.1, random_state=0).reset_index(drop=True)
train_label = train['target']
train_id = train['id']
test = pd.read_csv(path+'test.csv')
# test = test.sample(frac=0.1, random_state=0).reset_index(drop=True)
test_id = test['id']
NFOLDS = 4
kfold = StratifiedKFold(n_splits=NFOLDS, shuffle=True, random_state=218)
y = train['target'].values
drop_feature = [
'id',
'target'
]
X = train.drop(drop_feature,axis=1)
feature_names = X.columns.tolist()
cat_features = [c for c in feature_names if ('cat' in c and 'count' not in c)]
num_features = [c for c in feature_names if ('cat' not in c and 'calc' not in c)]
train['missing'] = (train==-1).sum(axis=1).astype(float)
test['missing'] = (test==-1).sum(axis=1).astype(float)
num_features.append('missing')
train.shape
for c in cat_features:
le = LabelEncoder()
le.fit(train[c])
train[c] = le.transform(train[c])
test[c] = le.transform(test[c])
# 事前にlabelEncoderを行っているから、この使い方でユニークな値で割り当てられる。引数categories = 'auto'で警告を消す
enc = OneHotEncoder(categories='auto')
enc.fit(train[cat_features])
X_cat = enc.transform(train[cat_features])
X_t_cat = enc.transform(test[cat_features])
ind_features = [c for c in feature_names if 'ind' in c]
count=0
for c in ind_features:
if count == 0:
train['new_ind'] = train[c].astype(str)+'_'
test['new_ind'] = test[c].astype(str)+'_'
count += 1
else:
train['new_ind'] += train[c].astype(str)+'_'
test['new_ind'] += test[c].astype(str)+'_'
cat_count_features = []
for c in cat_features+['new_ind']:
d = pd.concat([train[c],test[c]]).value_counts().to_dict()
train['%s_count'%c] = train[c].apply(lambda x:d.get(x,0))
test['%s_count'%c] = test[c].apply(lambda x:d.get(x,0))
cat_count_features.append('%s_count'%c)
train_list = [train[num_features+cat_count_features].values, X_cat]
test_list = [test[num_features+cat_count_features].values, X_t_cat]
X = ssp.hstack(train_list).tocsr()
X_test = ssp.hstack(test_list).tocsr()
def objective(trial):
drop_rate = trial.suggest_uniform('drop_rate', 0, 1.0)
feature_fraction = trial.suggest_uniform('feature_fraction', 0, 1.0)
learning_rate = trial.suggest_uniform('learning_rate', 0, 1.0)
subsample = trial.suggest_uniform('subsample', 0.8, 1.0)
num_leaves = trial.suggest_int('num_leaves', 5, 1000)
verbosity = trial.suggest_int('verbosity', -1, 1)
num_boost_round = trial.suggest_int('num_boost_round', 10, 100000)
min_data_in_leaf = trial.suggest_int('min_data_in_leaf', 10, 100000)
min_child_samples = trial.suggest_int('min_child_samples', 5, 500)
min_child_weight = trial.suggest_int('min_child_weight', 5, 500)
params = {"objective": "binary",
"boosting_type": "gbdt",
"learning_rate": learning_rate,
"num_leaves": num_leaves,
"max_bin": 256,
"feature_fraction": feature_fraction,
"verbosity": verbosity,
"drop_rate": drop_rate,
"is_unbalance": False,
"max_drop": 50,
"min_child_samples": min_child_samples,
"min_child_weight": min_child_weight,
"min_split_gain": 0,
"min_data_in_leaf": min_data_in_leaf,
"subsample": subsample
}
x_score = []
final_cv_train = np.zeros(len(train_label))
final_cv_pred = np.zeros(len(test_id))
cv_train = np.zeros(len(train_label))
cv_pred = np.zeros(len(test_id))
params['seed'] = 0
kf = kfold.split(X, train_label)
best_trees = []
fold_scores = []
for i, (train_fold, validate) in enumerate(kf):
print('kfold_index:', i)
X_train, X_validate, label_train, label_validate = \
X[train_fold, :], X[validate, :], train_label[train_fold], train_label[validate]
dtrain = lgbm.Dataset(X_train, label_train)
dvalid = lgbm.Dataset(X_validate, label_validate, reference=dtrain)
bst = lgbm.train(params, dtrain, num_boost_round, valid_sets=dvalid, feval=evalerror, verbose_eval=100,
early_stopping_rounds=100)
best_trees.append(bst.best_iteration)
cv_pred += bst.predict(X_test, num_iteration=bst.best_iteration)
cv_train[validate] += bst.predict(X_validate)
score = Gini(label_validate, cv_train[validate])
print(score)
fold_scores.append(score)
cv_pred /= NFOLDS
final_cv_train += cv_train
final_cv_pred += cv_pred
print("cv score:")
print(Gini(train_label, cv_train))
print("current score:", Gini(train_label, final_cv_train / (s + 1.)), s+1)
print(fold_scores)
print(best_trees, np.mean(best_trees))
x_score.append(Gini(train_label, cv_train))
print(x_score)
pd.DataFrame({'id': test_id, 'target': final_cv_pred / 16.}).to_csv('model/lgbm3_pred_avg_2.csv', index=False)
pd.DataFrame({'id': train_id, 'target': final_cv_train / 16.}).to_csv('model/lgbm3_cv_avg_2.csv', index=False)
return (1 - x_score[0])
study = optuna.create_study()
study.optimize(objective, n_trials=150)
パラメータの設定の範囲を抜粋すると以下のようになります。
drop_rate = trial.suggest_uniform('drop_rate', 0, 1.0)
feature_fraction = trial.suggest_uniform('feature_fraction', 0, 1.0)
learning_rate = trial.suggest_uniform('learning_rate', 0, 1.0)
subsample = trial.suggest_uniform('subsample', 0.8, 1.0)
num_leaves = trial.suggest_int('num_leaves', 5, 1000)
verbosity = trial.suggest_int('verbosity', -1, 1)
num_boost_round = trial.suggest_int('num_boost_round', 10, 100000)
min_data_in_leaf = trial.suggest_int('min_data_in_leaf', 10, 100000)
min_child_samples = trial.suggest_int('min_child_samples', 5, 500)
min_child_weight = trial.suggest_int('min_child_weight', 5, 500)
なお、Optuna自体の使用方法は、下記の記事と公式リファレンスを参考させていただきした。
https://qiita.com/ryota717/items/28e2167ea69bee7e250d
https://optuna.readthedocs.io/en/stable/index.html
(18/12/11 19:41追記)
コメントいただけた通り、'verbosity'は、警告レベルの表示を制御するパラメータであり、予測性能の最適化としては意味の無いパラメータでした。ですので、チューニングの対象にはすべきではありませんでした。
以下のように試行回数を定めていますが、
n_trials=150
時間が足りなくなった関係で、その時点で計算されたパラメータで最適化を中断しております。
20時間ほど回し回しましたが、ハイパーパラメータによって検証の時間は1分から60分程度となり、
100回くらいの試行数だったようです。
そうしてできてパラメータが、以下のように、2位の解法と比較すると以下のようになります。
| ハイパーパラメータ | 今回のチューニング結果 | 2位の解法 |
|---|---|---|
| drop_rate | 0.3015600134599976 | 0.1 |
| feature_fraction | 0.46650703511665226 | 0.6 |
| learning_rate | 0.004772377676601769 | 0.1 |
| subsample | 0.8080720420805803 | 0.9 |
| num_leaves | 718 | 15 |
| verbosity | -1 | 0 |
| num_boost_round | 1942 | 10000 |
| min_data_in_leaf | 212 | 150 |
| min_child_samples | 68 | 10 |
| min_child_weight | 151 | 150 |
2位コンペとの解法とは、雰囲気が異なるセットとなり、公開解法の再現ということにはならないようです。
K_fold=4 でやっていることも異なる要因になると思います。
算出できたハイパーパラメータでsubmit
最初のpython3のスクリプトからパラメータを入れ替え、予測値を算出しました。
K_fold =4,
また、ランダムシートの数を16から4に減らしております。
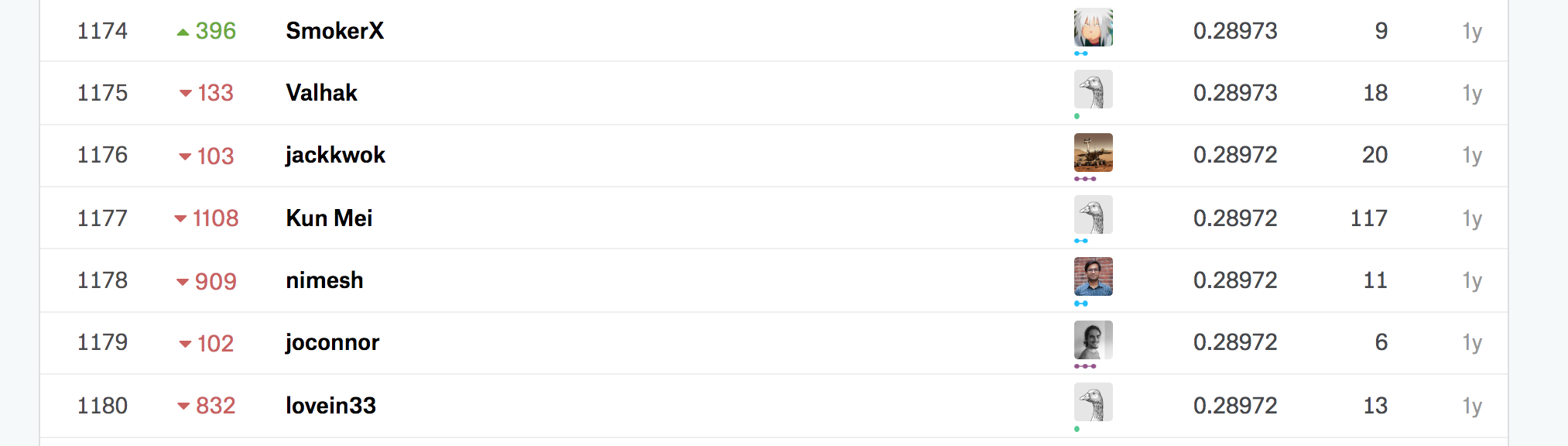
結果
スコアは下がってます。
1176位相当。。ハイパーパラメータ次第でシルバーメダル圏内ということを考えると、微妙な結果です。
所感
結果としては残念ですが、grid searchだけに頼らない、ハイパーパラメータの最適化方法の導入のきっかけになりました。
また、非常に手軽に使えたというのもあり、今後もチューニングの場面でOptunaを活用してみたいと思います。
反省としては、探索するハイパーパラメータの設定が悪く、計算の効率化が著しく悪くなった恐れがあります。
validationの際に、fold数の全て計算するのではなく、スコアが下がらなそうなら、そのハイパーパラメータの計算をやめるとか、一定時間以上かかってしまったらまた、次に試行に移るとかできれば効率化できたように思えます。
フレームワークはブラックボックスでもある程度は動かすことができますが、やはり中身をある程度理解しないと遠回りしてしまうというのは、当然の結果と言えます。
もっと使いこなせるよう精進しなければと思いました。
公式リファレンスでも、OptunaでLightGBMをチューニングする例が出ており、そちらの例も参考にしながらリベンジしたいと思います。
最後にですが、この記事が何かの役に経てば幸いです。