始めに:pandasの作者であるWes McKinneyさんがPythonのデータツール関連でとても興味深いblogを書かれているので、翻訳して日本のPyDataコミュニティに公開してもいいでしょうか、とお聞きしたところ、快諾をいただきましたので少しずつ訳して公開していこうと思っています。
翻訳元: Native Hadoop file system (HDFS) connectivity in Python
2017/1/3
これまで、Hadoop File SystemことHDFSとのやりとりするためのPythonライブラリが数多く開発されてきました。HDFSのWebHDFSゲートウェイ経由のものもあれば、ネイティブのProtocol BufferベースのRPCインターフェースもあります。このポストでは、既存のライブラリの概要をお伝えし、Arrowのエコシステム開発の中で高パフォーマンスのHDFSインターフェースを提供するために私がやってきたことを紹介します。
このblogは、2017年のロードマップに関するポストのフォローアップです。
Hadoop file systemのプロトコル
HDFSはApache Hadoopの一部であり、もともとその設計はオリジナルのMapReduceの論文に述べられているGoogle File Systemに基づいています。HDFSは、リモートプロシージャコール、すなわちRPCでネイティブのワイヤープロトコルとしてGoogleのProtocol Buffers(短く"protobufs"と呼ばれることもあります)を使っています。
通常、HDFSとやりとりをするシステムは、メインのJavaのクライアントと同様に、ProtobufのメッセージングフォーマットとRPCプロトコルを実装しているでしょう。低負荷のアプリケーションがファイルを簡単に読み書きできるようにするために開発されたのがWebHDFSで、これはprotobufsのRPCの代わりにPUTやGETリクエストを使えるHTTPあるいはHTTPSのゲートウエイを提供します。
低負荷のアプリケーションでは、WebHDFSとネイティブのprotobufs RPCのデータスループットは同等になりますが、概してネイティブの接続はスケーラビリティに優れ、実働環境での利用に適していると考えられています。
Pythonには、私が使ったことのあるWebHDFSインターフェースが2つあります:
- pywebhdfs
- hdfscli
この後この記事では、ネイティブのRPCクライアントインターフェースに注目していきます。
ネイティブRPCでのPythonからのアクセス
PythonのようにCとの相性のいい言語からネイティブなやり方でHDFSに接続する場合、Apache Hadoopでの「公式」なやり方はlibhdfsを使うことです。libhdfsは、HDFS JavaクライアントのJNIベースのCのラッパーです。libhdfsの主なメリットは、主要なHadoopのベンダーから配布され、サポートされており、Apache Hadoopプロジェクトの一部であることです。欠点としては、JNIを使っており(Pythonのプロセス内からJVMが起動されます)、クライアント側にHadoopのJavaディストリビューションが一式必要になることがあります。クライアントによっては、これは受け入れられない条件であり、他のクライアントと違ってプロダクションレベルのサポートは必要なものもあります。例えば、C++のアプリケーションであるApache Impala(インキュベーションプロジェクト)は、HDFS上のデータへのアクセスにlibhdfsを使っています。
libhdfsがもともと重いことから、代わりとなるHDFSへのネイティブインターフェースが開発されてきました。
-
libhdfs3は現在Apache HAWQ (インキュベーションプロジェクト)の一部となっている、純粋にC++のライブラリです。libhdfs3はPivotal Labsによって、SQL-on-HadoopシステムのHAWQで使用するために開発されました。libhdfs3が便利なのは、CのAPIレベルでlibhdfsと高い互換性を持っていることです。一時はlibhdfs3は公式にApache Hadoopの一部になりそうでしたが、現在はそうなることはなさそうになっています(HDFS-8707で新しいC++のライブラリが開発中なので、参照してください)。
-
snakebite: Hadoopのprotobuf RPCインターフェースの純粋なPython実装で、Spotifyによって開発されました。
snakebiteはクライアントのAPIを包括的に提供しているわけではなく(例えばファイルを書くことはできません)、パフォーマンスも良くない(純粋にPythonで実装されています)ので、ここからはlibhdfsとlibhdfs3に注目していきます。
libhdfs及びlibhdfs3へのPythonのインスターフェース
これまでにも、JNIライブラリのlibhdfsへのCのレベルでのインターフェースを構築しようとする動きはたくさんありました。それらの中には、cyhdfs(Cythonを利用)、libpyhdfs(通常のPythonのCによるエクステンション)、pyhdfs(SWIGを利用)などがあります。libhdfsへのCのエクステンションを構築する際の課題の1つは、共有ライブラリのlibhdfs.soがHdoopのディストリビューションに含まれて配布されているので、この共有ライブラリをロードできるよう、$LD_LIBRARY_PATHを適切に設定しなければならないことです。加えて、インポートの際にはJVMのlibjvm.soもロードできなければなりません。これらの条件が組み合わさると、「設定地獄」にはまっていくことになります。
Apache Arrow(そしてPyArrow経由でPythonでも)で使うC++のHDFSインターフェースを構築しようと考えているときに、私はTuriのSFrameプロジェクトのlibhdfsの実装を見つけました。これは、実行時にJVMとlibhdfsをロードする際に、この両方をどちらも賢明なアプローチで見つけるようになっていました。私はArrowでこのアプローチを採用し、それはうまくいきました。この実装を使うことで、Arrowのデータシリアライゼーションツール群(Apache Parquetのような)でのI/Oのオーバーヘッドは非常に低くなり、便利なPythonのファイルインターフェースも提供してくれました。
libhdfsとlibhdfs3のドライバライブラリのCのAPIはほとんど同じなので、Pythonのキーワード引数に従ってドライバを切り替えることができました。
from pyarrow import HdfsClient
# libhdfsを使用
hdfs = HdfsClient(host, port, username, driver='libhdfs')
# libhdfs3を使用
hdfs_alt = HdfsClient(host, port, username, driver='libhdfs3')
with hdfs.open('/path/to/file') as f:
...
これを並行して、Daskプロジェクトの開発者たちはlibhdfs3に対する純粋なPythonのインターフェースであるhdfs3を作成しました。これは、Cのエクステンションを回避するためにctypesを使っていました。hdfs3は、Pythonのファイルインターフェースとともに、libhdfs3の他の機能へのアクセスも提供しています。
from hdfs3 import HDFileSystem
hdfs = HDFileSystem(host, port, user)
with hdfs.open('/path/to/file', 'rb') as f:
...
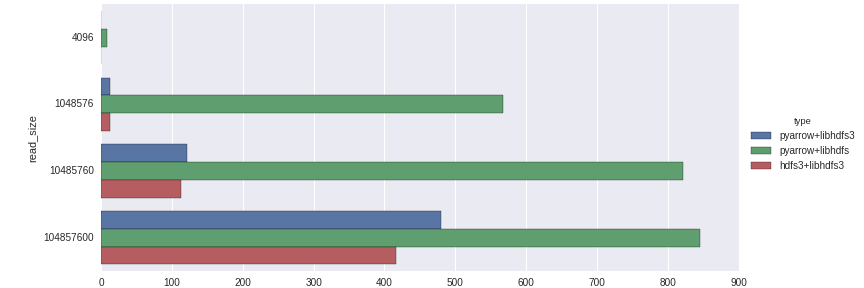
pyarrow.HdfsClientとhdfs3のデータアクセスパフォーマンス
ローカルのCDH 5.6.0 HDFSクラスタに対し、3種類の設定で4KBから100MBのサイズのファイル群の読み取りパフォーマンスの集合平均を計算してみました。
- hdfs3(常にlibhdfs3を使用)
- driver='libhdfs'でのpyarrow.HdfsClient
- driver='libhdfs3'でのpyarrow.HdfsClient
これらのパッケージはすべて以下のようにすれば取得できます。
conda install pyarrow hdfs3 libhdfs3 -c conda-forge
注意:pyarrowのconda-forgeパッケージは、現在Linuxでのみ提供されています。理論上は、この問題は2017年1月20日には解決されているはずです。Windowsサポートを手助けしてくださる方がおられましたら、お知らせください。
パフォーマンスの数値は、メガバイト/秒です("throughput")。ベンチマークのコードはこのポストの最後にあります。もっと多彩なプロダクション環境やHadoopの設定でこの結果がどうなるか、私は興味を持っています。
少なくとも私が行ったテストでは、以下のような興味深い結果が得られました。
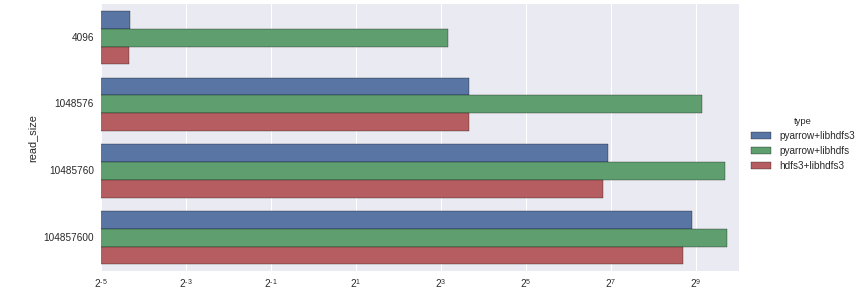
- libhdfsは、Java及びJNIベースであるにもかかわらず、このテストでは最高のスループットを示しました。
- libhdfs3は、小さいサイズの読み取りのパフォーマンスが良くありませんでした。これは、RPCのレイテンシーか、私が認識できていない問題が設定にあるためかもしれません。
- 厳密にlibhdfs3と比較すれば、pyarrowはhdfs310-15%程度上回っています。これは主に、ctypes(hdfs3)とC++(pyarrow)の違いによるメモリのハンドリング/コピーによるものだと思われます。
以下は、時間を対数軸にしたものです。
Apache ArrowにおけるネイティブのC++ I/O
pyarrowライブラリ内でHDFSのようなI/Oインターフェースを構築する理由の1つは、そういったインターフェースはすべて共通のメモリ管理のレイヤーを使用して、非常に低い(場合によってはゼロ)コピーのオーバーヘッドだけで、データを渡せるためです。これに対して、Pythonのfileインターフェースだけを公開しているライブラリは、メモリの処理をPythonインタープリタ内のバイト列のオブジェクトで行うため、多少のオーバーヘッドが生じます。
ArrowのC++のI/Oシステムの詳細はこの記事の範囲を超えますが、将来このブログにそれに関するポストを書くことにしましょう。
ベンチマークのコード
import gc
import random
import time
import pyarrow as pa
import hdfs3
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
DATA_SIZE = 200 * (1 << 20)
data = 'a' * DATA_SIZE
hdfs = pa.HdfsClient('localhost', 20500, 'wesm')
hdfscpp = pa.HdfsClient('localhost', 20500, 'wesm', driver='libhdfs3')
hdfs3_fs = hdfs3.HDFileSystem('localhost', port=20500, user='wesm')
hdfs.delete(path)
path = '/tmp/test-data-file-1'
with hdfs.open(path, 'wb') as f:
f.write(data)
def read_chunk(f, size):
# do a random seek
f.seek(random.randint(0, size))
return f.read(size)
def ensemble_average(runner, niter=10):
start = time.clock()
gc.disable()
data_chunks = []
for i in range(niter):
data_chunks.append(runner())
elapsed = (time.clock() - start) / niter
gc.enable()
return elapsed
def make_test_func(fh, chunksize):
def runner():
return read_chunk(fh, chunksize)
return runner
KB = 1024
MB = 1024 * KB
chunksizes = [4 * KB, MB, 10 * MB, 100 * MB]
iterations = [100, 100, 100, 10]
handles = {
('pyarrow', 'libhdfs'): hdfs.open(path),
('pyarrow', 'libhdfs3'): hdfscpp.open(path),
('hdfs3', 'libhdfs3'): hdfs3_fs.open(path, 'rb')
}
timings = []
for (library, driver), handle in handles.items():
for chunksize, niter in zip(chunksizes, iterations):
tester = make_test_func(handle, chunksize)
timing = ensemble_average(tester, niter=niter)
throughput = chunksize / timing
result = (library, driver, chunksize, timing, throughput)
print(result)
timings.append(result)
results = pd.DataFrame.from_records(timings, columns=['library', 'driver', 'read_size', 'timing', 'throughput'])
results['MB/s'] = results['throughput'] / MB
results
results['type'] = results['library'] + '+' + results['driver']
plt.figure(figsize=(12, 6))
g = sns.factorplot(y='read_size', x='MB/s', hue='type', data=results, kind='bar', orient='h', size=(10))
g.despine(left=True)
# g.fig.get_axes()[0].set_xscale('log', basex=2)
g.fig.set_size_inches(12, 4)
plt.savefig('results2.png')