SeleneはPyTorchベースのDeepLearningライブラリで、シークエンスデータ解析に特化しています。
医療画像でいうNiftyNetのような感じで、コードを書くことなくcofigファイルを設定をするだけでDeepLearningが出来るようになっています。
サンプリングなどを同じ条件で合わせて行うことが出来るので、モデル性能の比較がしやすいです。
また、Seleneで作ったモデルは前回の記事のKipoiによって共有することも出来ます。
github:https://github.com/FunctionLab/selene
論文:https://www.nature.com/articles/s41592-019-0360-8
ドキュメント:https://selene.flatironinstitute.org/
ちなみにWeb系のライブラリで全く同じ名前のPythonライブラリがあるので注意です。
インストール
Python 3.6以上と、PyTorchのインストールが前提です。
環境はUbuntsu16.04、CUDA10、Anaconda、Python3.6、PyTorch1.2.0で行いました。
pip install selene-sdk
これでインストール出来ました。バージョンは0.4.8でした。
動かしてみる
基本的にはconfigファイルで設定を書いてrunするだけです。
configファイル(YAML形式)の書き方はこちらにあります。
オペレーション、モデル、サンプラー、パラメータの4つを設定していきます。
データは、トレーニングデータ・バリデーションデータ・テストデータに再現性を保って振り分けられます。
まずはgit cloneします。
git clone https://github.com/FunctionLab/selene.git
git cloneに結構時間がかかりました。
config_examplesフォルダ内にはconfigファイルの例があって参考になります。
今回はチュートリアルのquickstart_trainingを試してみます。
cd selene/tutorials/quickstart_training
wget https://zenodo.org/record/1443558/files/selene_quickstart.tar.gz
tar -zxvf selene_quickstart.tar.gz
mv selene_quickstart_tutorial/* .
データのダウンロードには時間がかかります。
selene_quickstart.tar.gzのダウンロードができない時はこちらの方法に代えることもできます。
configはdeepSEAをより深くしたモデルを使っています。RandomPositionsSamplerに変更しています。「simple_train.yml」
ops: [train, evaluate]
model: {
path: ./deeperdeepsea.py,
class: DeeperDeepSEA,
class_args: {
sequence_length: 1000,
n_targets: 1,
},
non_strand_specific: mean
}
sampler: !obj:selene_sdk.samplers.RandomPositionsSampler {
reference_sequence: !obj:selene_sdk.sequences.Genome {
input_path: ./male.hg19.fasta
},
features: !obj:selene_sdk.utils.load_features_list {
input_path: ./distinct_features.txt
},
target_path: ./sorted_GM12878_CTCF.bed.gz,
seed: 100,
sequence_length: 1000,
center_bin_to_predict: 200,
test_holdout: [chr8, chr9],
validation_holdout: [chr6, chr7],
feature_thresholds: 0.5,
mode: train,
save_datasets: [validate, test]
}
train_model: !obj:selene_sdk.TrainModel {
batch_size: 64,
max_steps: 8000,
report_stats_every_n_steps: 1000,
n_validation_samples: 32000,
n_test_samples: 120000,
cpu_n_threads: 10,
use_cuda: True,
data_parallel: False
}
random_seed: 1445
output_dir: ./training_outputs
create_subdirectory: False
load_test_set: False
runするには、Pythonで以下のコードを実行するだけです。
from selene_sdk.utils import load_path, parse_configs_and_run
parse_configs_and_run(load_path("./simple_train.yml"), lr=0.01)
GPUメモリを2GBほど使用して、数分で学習を終えることができました。
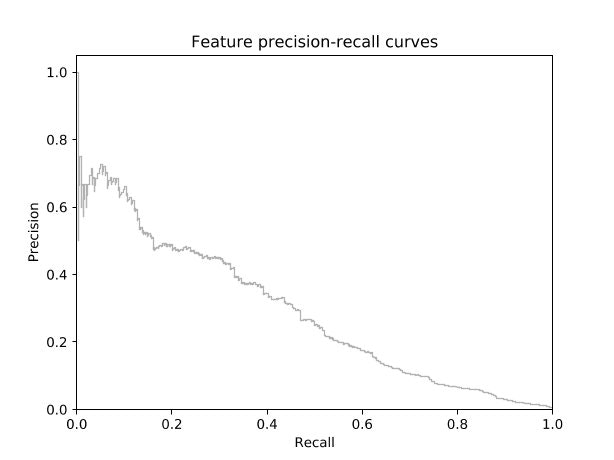
結果は以下です。(テストデータでの精度評価)
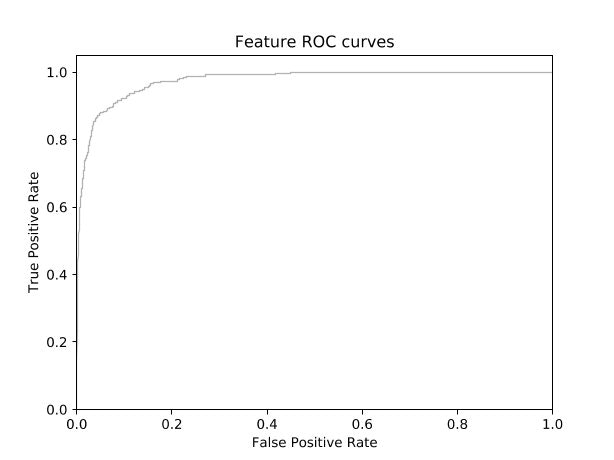
モデルはbest_model.pth.tarに保存されました。
DeepSEAのモデルが出来ると、変異を入れたシークエンスデータを入力とすることでその変異の影響を予測することが出来ます。
その予測によってGWASの結果をシミュレーションするようなことが出来るわけですね。
感想
Kipoiにも対応していて、非常に便利なライブラリだと感じました。
今回はサンプルを試すことしかできなかったので、次は自分でモデルを設定してやりたいですね。