はじめに
久しぶりに Quiita に投稿します。
最近、新型コロナウイルスのパンデミックに関する統計データを分析することを始めました(仕事ではなく個人のライフワーク?として)。
そして幾つかブログに記事にアップしています。
- 新型コロナの救命率から見た各国の状況:救命率が極めて低いあの先進国がとった政策とは?│YUUKOU's 経験値
- 新型コロナの救命率の推移から読み解く:米国の底力、危機的なイギリス、オランダ、推移が美し過ぎる中国│YUUKOU's 経験値
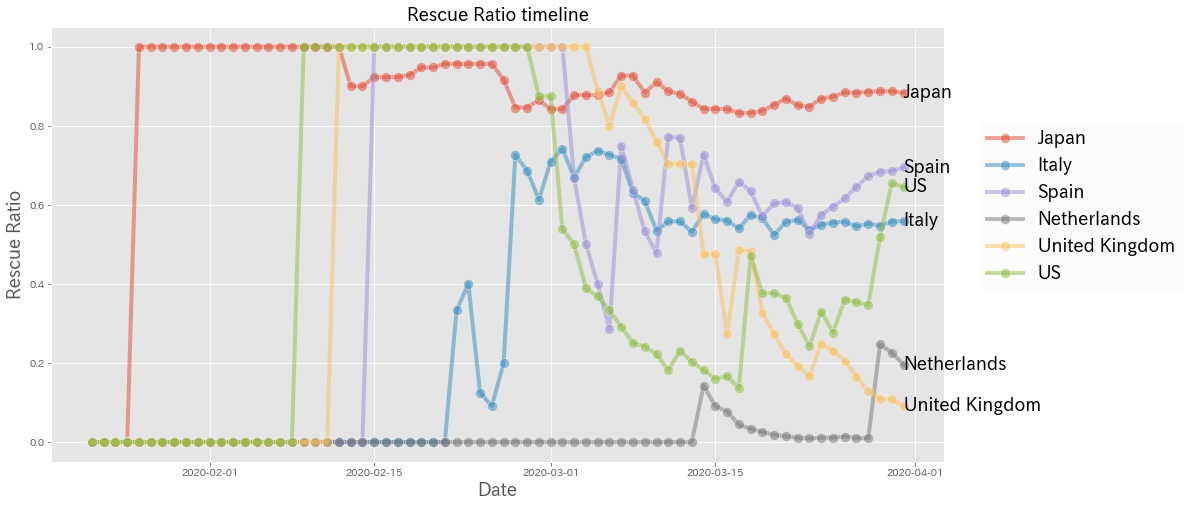
例えば、救命率についての時系列推移をプロットしたチャートをデータ分析の成果として掲載していたりします。(感染者のカウント基準が国によって違う点はあるものの、データを見る限り日本は世界的に見ても医療現場が優秀であることがうかがえます)
今回は、ジョンズホプキンス大学が提供している新型コロナウイルス統計データを分析するための下準備のコードを共有したいと思います。
このコードを使えば、新型コロナウイルス統計のデータフレームが生成され、思い思いのデータ分析に取り組む準備ができると思います。
ぜひご活用いただけると、微力ながら貢献できると幸いです。
データダウンロード&加工処理
ジョンズホプキンス大学は、github上に全世界の新型コロナウイルス感染の統計データ(しかも時系列で!)で公開しています。
処理全体の流れとしては、urllib を使ってデータを取得した後、加工します。
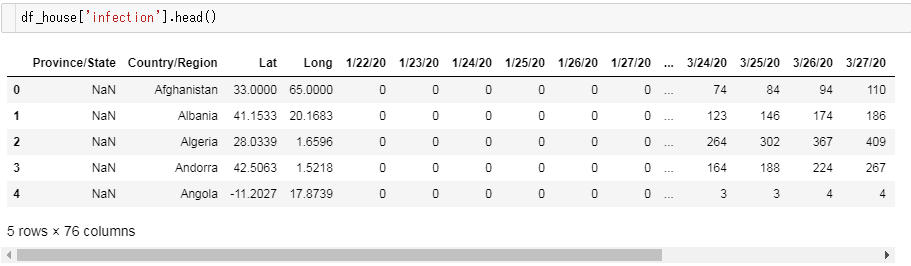
ジョンズホプキンス大学が公開している統計データには、感染確認者数(confirmed)、死亡者数(deaths)、治癒者数(recovered)の3つが含まれています。
また、その粒度は、各国の地域単位まで記録しているレコードもあります。
今回は国単位に集約して分析します。
ただし、注意点が一つあります。
時系列といっても、列方向に日付ごとのカラムがズラリと何十個も並んでいるので、それを使いやすい構造に変換しなければなりません。
例えば、こんなデータフレームです。(感染確認者数の場合)
日付らしきカラムが並んでいるのが分かりますね。
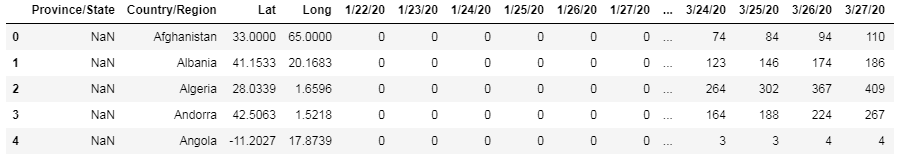
時系列カラムを行方向に構造変換し、国ごとに集約すれば、扱いやすいオーソドックスなデータフレームに落ち着きます。
今回は、Jupyter Notebook 上で実装しました。
なので、エントリに掲載したコードをそのまま上から順番に張り付けて実行すれば動くと思います。
クローラークラスの実装
クローラークラスを定義します。
名前はそのまんまですね。他のノートブックでも使いまわしそうなので、とりあえずクラス化しておきました。
import urllib
import json
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
import numpy as np
import io
from dateutil.parser import parse
from tqdm import tqdm, tqdm_notebook
class Crowler():
def __init__(self):
"""
クローラークラス
"""
self._ua = 'Mozilla/5.0 (Macintosh; Intel Mac OS X 10_12_3) '\
'AppleWebKit/537.36 (KHTML, like Gecko) '\
'Chrome/55.0.2883.95 Safari/537.36 '
def fetch(self, url):
"""
URLを指定し、HTTPリクエストを実行する。
:param url:
:return: リクエスト結果(html)
"""
req = urllib.request.Request(url, headers={'User-Agent': self._ua})
return urllib.request.urlopen(req)
各種設定
クローラーインスタンスの宣言と、各データソースのURLを定義します。
# クローラーインスタンス
cr = Crowler()
# 感染者推移の時系列データ
url_infection = 'https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_confirmed_global.csv'
# 死亡者の時系列データ
url_deaths = 'https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_deaths_global.csv'
# 治癒者の時系列データ
url_recover = 'https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_recovered_global.csv'
各データソースの取得
3つのデータソースをクローリングして、いったんデータフレームに変換します。
url_map = {'infection': url_infection,
'deaths': url_deaths,
'recover': url_recover}
df_house = {}
for _k, _url in url_map.items():
_body_csv = cr.fetch(_url)
df_house[_k] = pd.read_csv(_body_csv)
df_house は、3つのデータフレームを格納する辞書です。
中身は次のようになっています。
テーブル構造の変換
日付型に変換する関数の準備
時系列カラムは 3/27/20 のような形式で、Python の dateutil.parser.parse ではそのままでは変換できません。
泥臭いのですが、いったん標準的な YYYY-mm-dd形式に変換するための関数を用意します。
def transform_date(s):
"""
'3/15/20' 形式の日付を '2020-03-15' のように 'YYYY-mm-dd' 形式に直す
"""
_chunk = str(s).split('/')
return '20{year}-{month:02d}-{day:02d}'.format(year=_chunk[2], month=int(_chunk[0]), day=int(_chunk[1]))
各データフレームを変換する
3つのデータフレームのそれぞれにおいて、時系列のカラムを行に変換します。
date というカラムに時系列を持たせるように変換します。
df_buffer_house = {}
for _k, _df in df_house.items():
df_buffer_house[_k] = {'Province/State':[],
'Country/Region':[],
'date': [],
_k: []}
_col_dates = _df.columns[4:]
for _k_date in tqdm(_col_dates):
for _idx, _r in _df.iterrows():
df_buffer_house[_k]['Province/State'].append(_r['Province/State'])
df_buffer_house[_k]['Country/Region'].append(_r['Country/Region'])
df_buffer_house[_k]['date'].append(transform_date(_k_date))
df_buffer_house[_k][_k].append(_r[_k_date])
Jupyter Notebook上で実行すると、次のようにプログレスバーを表示しながら変換を進めて行きます。
100%|██████████████████████████████████████████| 72/72 [00:05<00:00, 12.37it/s]
100%|██████████████████████████████████████████| 72/72 [00:05<00:00, 12.89it/s]
100%|██████████████████████████████████████████| 72/72 [00:05<00:00, 13.27it/s]
3つのデータフレームの構造がだいぶマシになったので、あとは結合するだけなのですが、そこで注意点があります。
感染者数(infection)や死亡者数(deaths)では、Province/Stateが複数記録されているが、治癒数(recover) では国単位という記録がされていることがあります。
例) Canada
よって、各データフレームごとに国ごとに集約してから結合する必要があるのです。
df_integrated = pd.DataFrame()
col_integrated = ['Country/Region', 'date']
df_chunk = {}
for _k, _df_dict in df_buffer_house.items():
_df_raw = pd.DataFrame.from_dict(_df_dict)
# 'Country/Region' ごとに集約する
_df_grouped_buffer = {'Country/Region':[], 'date':[] , _k:[]}
for _idx, _grp in tqdm(_df_raw.groupby(col_integrated)):
_df_grouped_buffer['Country/Region'].append(_idx[0])
_df_grouped_buffer['date'].append(_idx[1])
_df_grouped_buffer[_k].append(_grp[_k].sum())
df_chunk[_k] = pd.DataFrame.from_dict(_df_grouped_buffer)
df_integrated = df_chunk['infection'].merge(df_chunk['deaths'], on=col_integrated, how='outer')
df_integrated = df_integrated.merge(df_chunk['recover'], on=col_integrated, how='left')
実行します。
100%|██████████████████████████████████| 13032/13032 [00:08<00:00, 1621.81it/s]
100%|██████████████████████████████████| 13032/13032 [00:08<00:00, 1599.91it/s]
100%|██████████████████████████████████| 13032/13032 [00:07<00:00, 1647.02it/s]
動作確認
先ほどの例で挙げたカナダがちゃんとしたデータに変換されているか確かめてみましょう。
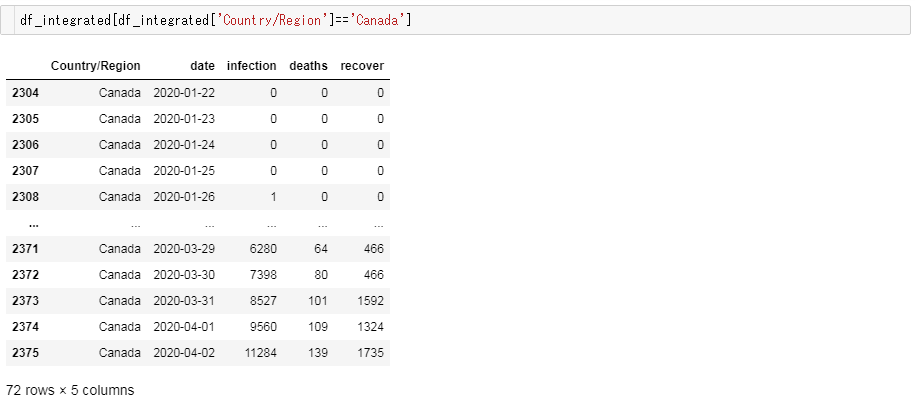
大丈夫そうですね!
Nan で欠落するレコードが多発している様子もなく、時系列順に数値が推移している様子も確認できました!
変換後の統計データを使った分析例
こうして変換して得られた新型コロナウイルスの統計データを使った分析コードの例を紹介したいと思います。
救命率、感染終息度の計算
救命率の計算
救命率とは、ここでは治療が終了した患者数(Closed Cases)(※)のうち、治癒した患者数(Total Recovered Cases)の割合と定義したいと思います。
※治療が終了した患者(Closed Cases)は、次のように分けられます。
(1) 治癒した患者(Recovered Cases)
(2) 死亡した患者(Death Cases)
$$Resuce Ratio(救命率) = \frac{TotalRecovered(治癒した患者数)}{ClosedCases(治療が終了した患者数)}$$
感染終息度の計算
各国の感染が、どれくらい終息に近づいているのかを表した数字です。
感染者累計に対して、どれくらいの患者の治療が終了しているのか、その割合で表します。
$$Phase Position(感染終息度) = \frac{Closed Case(治療が終了した患者数)}{Total Case(感染者数累計)}$$
Phase Position は、0.0 ~ 1.0 の値を取ります。
0.0 に近いほど感染フェーズが序盤であることを表します。
1.0に近づくほど感染フェーズが終盤であることを表します。
計算コード例
df_grouped = df_integrated
df_grouped['date'] = pd.to_datetime(df_grouped['date'])
# 救命率の計算
df_grouped['rescue_ratio'] = df_grouped['recover']/(df_grouped['recover'] + df_grouped['deaths'])
df_grouped['rescue_ratio'] = df_grouped['rescue_ratio'].fillna(0)
# 感染終息度の計算
# 治療終了患者数 = 治癒した患者数 + 死亡した患者数
df_grouped['phase_position'] = (df_grouped['recover'] + df_grouped['deaths'])/df_grouped['infection']
計算結果の確認
米国を例に、計算結果を確認してみましょう。
すると以下のようなデータフレームが表示されます。
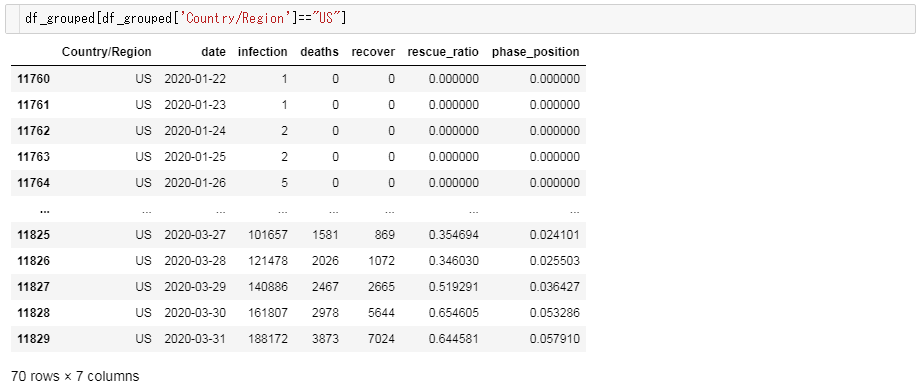
米国はまだまだ感染の序盤であり、救命率は持ち直しつつあるものの依然として厳しい状況にあることがうかがえます。
まとめと分析エントリの紹介
ということで、新型コロナウイルスの統計データを分析するための下準備のコードを紹介させていただきました。
ジョンズホプキンス大学の統計データは、現在世界で注目されているデータソースの一つですので、ぜひ皆さんの様々な分析アイディアを試行錯誤して、積極的に情報発信していただけると良いのではないかと思います!
ということで、まずは言いだしっぺからということで、私が執筆した新型コロナ分析エントリをご紹介して、締めくくりたいと思います。
- 新型コロナの救命率から見た各国の状況:救命率が極めて低いあの先進国がとった政策とは?│YUUKOU's 経験値
- 新型コロナの救命率の推移から読み解く:米国の底力、危機的なイギリス、オランダ、推移が美し過ぎる中国│YUUKOU's 経験値
- 新型コロナウイルス・世界各国の感染フェーズを数値化した結果:ぶっちぎりで米国ヤバイ│YUUKOU's 経験値