本記事は、AI/ML on AWS Advent Calendar 2022の18日目の記事です。
以前の記事で、TabBERTモデル(IBM論文の付属コード)の環境構築と学習(事前学習、Fine-Tuning)をEC2上で行いました。
動作確認くらいであればこれで問題なかったのですが、いざ本番運用を考えてみると、以下のような問題がありそうでした。
- 学習中以外の時間にかかるEC2のコスト
- 推論環境を別途たてることになったときの環境再構築コスト
- 入出力データの管理の手間
調べてみたところ、Amazon SageMakerというAWSの機械学習用マネジメントサービスを使えばこれらを解決できそうだったので、技術書展で購入した書籍を参考に、勉強がてらSageMakerを触ってみることにしました。
いざ触ってみるとつまづきポイントが多くて心が折れかけたのですが、1週間粘ってようやくJobを成功させることができました。
今回の記事はSageMaker Training Jobsの構築手順が主ですが、その中につまづいたポイントの記述も行っています。
同じ部分で悩んでいる方の参考になれば幸いです。
- Dockerfileのベースイメージ選択
- conda環境切替のためのDockerfile
- ノートブックインスタンスの作成
- コンテナとS3の入出力データのやりとり
- Training Job実行時のコンテナのパス(/opt/ml/~)
- Training Job実行前のconda環境切替(conda activate)
- 環境変数によるhyperparametersの引き渡し
Docker環境構築
Dockerfile作成、イメージビルド、ECRプッシュまでの手順をまとめました。
Dockerfile作成
SageMakerでは、Training Jobを実行する際にコンテナイメージから学習用のインスタンスを起動します。
以前、EC2で環境構築した際はDeep Learning AMIを使用し、ymlファイルからconda環境をつくりました。
コンテナで同様の環境をつくるにあたり、いろいろ試行錯誤しました。
先にDockerfile全文を記載します。
FROM 763104351884.dkr.ecr.ap-northeast-1.amazonaws.com/pytorch-training:1.12.1-gpu-py38-cu113-ubuntu20.04-sagemaker
COPY environment.yml .
RUN pip install --upgrade pip && \
conda update -n base -c defaults conda && \
conda env create -f environment.yml && \
conda init bash && \
echo ". /opt/conda/etc/profile.d/conda.sh" >> ~/.bashrc && \
echo "conda activate tabformer-opt-sagemaker" >> ~/.bashrc
ENV CONDA_DEFAULT_ENV tabformer-opt-sagemaker && \
PATH /opt/conda/envs/tabformer-opt-sagemaker/bin:$PATH
ENV LD_LIBRARY_PATH $LD_LIBRARY_PATH:/opt/conda/envs/tabformer-opt-sagemaker/lib
RUN echo $LD_LIBRARY_PATH
ベースイメージ選定
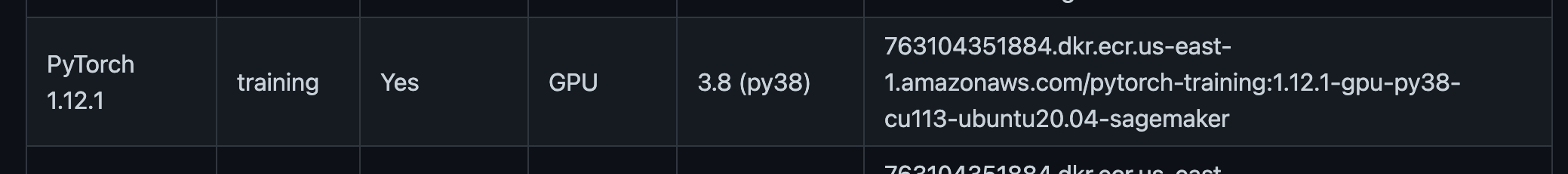
FROM 763104351884.dkr.ecr.ap-northeast-1.amazonaws.com/pytorch-training:1.12.1-gpu-py38-cu113-ubuntu20.04-sagemaker
EC2ではDeep Learning AMI GPU PyTorch 1.13.0 (Ubuntu 20.04) 20221110を使っていたので、それに相当するベースイメージを探しました。
調査に時間がかかってしまったのですが、AWSが公開しているdeep-learning-containersというものをみつけました。
SageMaker Framework Containersから、以下を選択しました(PyTorch 1.13.0がなく、1.12.1を選択しました)。
今回は以上のベースイメージを使用しましたが、Pyhonが動き、build-essentialとsagemaker-trainingが入っている環境であれば、Training Jobは動作するとのことです。
conda環境の構築
RUN pip install --upgrade pip && \
conda update -n base -c defaults conda && \
conda env create -f environment.yml && \
conda init bash && \
echo ". /opt/conda/etc/profile.d/conda.sh" >> ~/.bashrc && \
echo "conda activate tabformer-opt-sagemaker" >> ~/.bashrc
ENV CONDA_DEFAULT_ENV tabformer-opt-sagemaker && \
PATH /opt/conda/envs/tabformer-opt-sagemaker/bin:$PATH
以下の記事でも書いたのですが、元コードのsetup.ymlから動作するconda環境をつくれず、追加でライブラリを導入するなどいろいろいじりました。
そのため、最終的に動作したconda環境を新しいymlファイルenvironment.ymlに書き出し、そのファイルを元にconda環境をつくるようにしました。
また、コンテナを立ち上げたときにconda環境も立ち上がるよう、.bashrcにconda activate ~を書き込み、デフォルト環境CONDA_DEFAULT_ENVとして設定しました。
※結局これだけではconda環境が立ち上がらず、後述のbashスクリプト内でconda activateしました。
補足
ENV LD_LIBRARY_PATH $LD_LIBRARY_PATH:/opt/conda/envs/tabformer-opt-sagemaker/lib
RUN echo $LD_LIBRARY_PATH
以上のパスを通さないと学習が実行できなかったので追記しました。
私の環境だと必要でしたが、通常であればこちらの記述は不要です。
イメージビルドとECRへのプッシュ
deep-learning-containersのイメージをベースとして使う場合、イメージをビルドする前にリージョンとECRレジストリを指定して、認証トークンの取得とDockerクライアントの認証を行う必要があります。
$ aws ecr get-login-password --region ap-northeast-1 | docker login --username AWS --password-stdin 763104351884.dkr.ecr.ap-northeast-1.amazonaws.com
これでイメージビルドを行うことができます。
$ docker build -t <アカウントID>.dkr.ecr.ap-northeast-1.amazonaws.com/tabformer-opt:<タグ> .
また、イメージプッシュする際にも、自アカウントのECRレジストリへの認証を行います。
$ aws ecr get-login-password --region ap-northeast-1 | docker login --username AWS --password-stdin <アカウントID>.dkr.ecr.ap-northeast-1.amazonaws.com
これでイメージプッシュを行うことができます。
$ docker push <アカウントID>.dkr.ecr.ap-northeast-1.amazonaws.com/tabformer-opt:<タグ>
Training Jobの作成
Training Jobを作成するにあたり、ノートブックインスタンスに学習用のコードとJobを実行するコードを配置する必要があります。
ノートブックインスタンスの作成
以下を付与したIAMロールを作成し、ノートブックインスタンス作成時にアタッチします。
- AmazonSageMakerFullAccess
- AmazonS3FullAccess
- AmazonEC2ContainerRegistryFullAccess
フォルダ構成
作成したノートブックインスタンスからJupyterを開き、以下のようにフォルダやファイルを配置します(画像のshell.shは不要です)。
-
tabformer-opt:学習用のコード -
pre-training_jobs.ipynb:Jobの実行コード -
pre-training.sh:conda activate、Python実行用bashスクリプト
pre-training_jobs.ipynbの作成
pre-training_jobs.ipynbを作成し、前項で作成したコンテナイメージを読み込んでモデルの学習を実行できるようにします。
モデルの学習にはsagemaker.estimator.Estimatorを使用します。
こちらがコード全文です。
import sagemaker
from sagemaker.estimator import Estimator
session = sagemaker.Session()
role = sagemaker.get_execution_role()
estimator = Estimator(
image_uri=<作成したコンテナイメージのURL>,
role=role,
instance_type="ml.g4dn.2xlarge",
instance_count=1,
base_job_name="tabformer-opt-pre-training",
output_path="s3://<バケット名>/sagemaker/output_data/pre_training",
code_location="s3://<バケット名>/sagemaker/output_data/pre_training",
sagemaker_session=session,
entry_point="pre-training.sh",
dependencies=["tabformer-opt"],
hyperparameters={
"mlm": True,
"do_train": True,
"field_hs": 64,
"output_dir": "/opt/ml/model/",
"data_root": "/opt/ml/input/data/input_data/",
"data_fname": "<ファイル名>"
}
)
estimator.fit({
"input_data": "s3://<バケット名>/sagemaker/input_data/<ファイル名>.csv"
})
コンテナパスとS3パスの対応
SageMakerではコンテナ起動時に/opt/ml/~のパスが使用されます。
S3とのデータ入出力を考えるとき、コンテナでのデータ配置とS3パスとの対応を理解しておく必要があります。
/opt/ml/model
こちらのパスにデータを保存すると、学習終了時にS3にデータが自動でアップロードされます。
S3の保存先は、Estimatorの引数output_pathで指定します。
学習終了時ではなくリアルタイムでS3にデータをアップロードする場合は、/opt/ml/checkpointsにデータを保存します。
このとき、S3の保存先はcheckpoint_s3_uriで指定します。
/opt/ml/code
pre-training_jobs.ipynbと同じディレクトリにあるフォルダとファイル(今回の場合tabformer-optとtabformer-opt.sh)がこちらに配置されます。
/opt/ml/input
estimator.fitで入力データのあるS3パスを<key>: <S3 URL>で指定すると、コンテナでは/opt/ml/input/data/<key>以下に配置されます。
estimator.fit({
"input_data": "s3://<バケット名>/sagemaker/input_data/<ファイル名>.csv"
})
pre-training.shの作成
こちらのbashスクリプトでは、conda環境の切替(conda activate)、フォルダtabformer-optの移動、学習を行います。
#!/bin/bash
"===conda activate==="
conda init bash
source /opt/conda/etc/profile.d/conda.sh
conda activate tabformer-opt-sagemaker
"===move directory==="
mv ./tabformer-opt/* .
"===pre-training==="
python main.py
conda activate
Dockerfileにもconda activateを記述したのですが、conda init bash後のCommandNotFoundErrorを解消できませんでした。
そのため、bashスクリプト内でsource /opt/conda/etc/profile.d/conda.shを実行し、conda activateされるようにしました。
move directory
学習のためにはtabformer-opt/main.pyを実行する必要があります。
main.pyを実行するにはtabformer-opt内の依存ファイルも読み込む必要があり、そのためにEstimatorでdependencies=["tabformer-opt"]を記述します。
あとはentry_point: python tabformer-opt/main.pyとすればよさそうですが、これだとライブラリのカレントディレクトリが変わってしまい、importがうまくいかずJobが失敗します。
そのため、mv ./tabformer-opt/* .でtabformer-opt以下をカレントディレクトリに移動させています。
環境変数によるhyperparametersの引き渡し
main.pyを実行するにあたり、今回のコードでは--do_train --mlmなどをコマンドライン引数として渡す必要があります。
しかし、tabformer-opt.shがentry_pointになったことにより、Estimatorのhyperparametersをコマンドライン引数としてmain.pyに渡すことができません。
ここでSageMakerの環境変数SM_HPSを利用します。
SM_HPSにはhyperparametersのJSONがテキストに変換されたものが入っています。
SM_HPS={"data_fname":"summary","data_root":"/opt/ml/input/data/input_data/","do_train":"True","field_hs":64,"mlm":"True","output_dir":"/opt/ml/model/"}
これをmain.pyで以下のように読み込むと、SageMakerを使用しているとき(Training Jobsを実行しているとき)のみ、hyperparametersを利用するようになります。
if __name__ == "__main__":
parser = define_main_parser()
opts = parser.parse_args()
if "SM_HPS" in os.environ.keys():
hps = json.loads(os.environ["SM_HPS"])
for key, value in hps.items():
if opts.__contains__(key):
opts.__setattr__(key, value)
main(opts)
Training Jobの実行
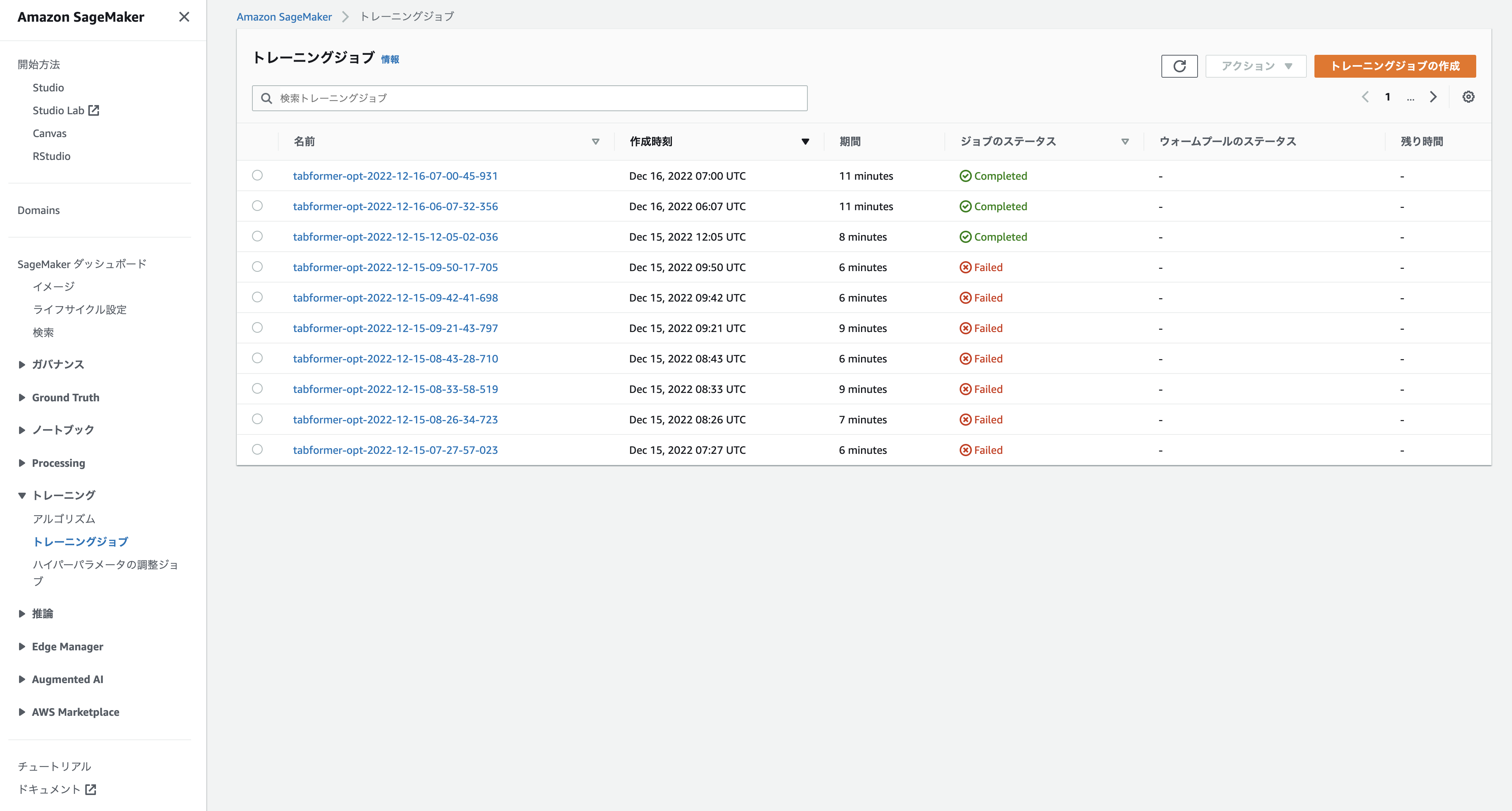
pre-training_jobs.ipynbを実行すると、Training Jobが実行されます。
Jobが成功すると、ジョブのステータスがCompletedとなります。
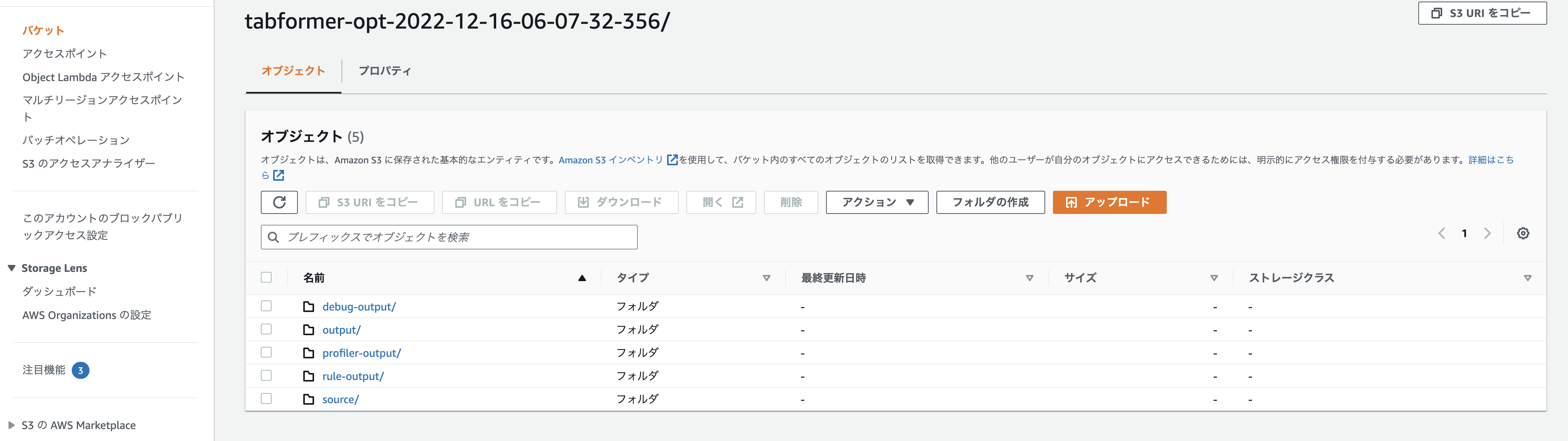
Job実行後にS3を確認すると複数のオブジェクトがあり、outputのmodel.tar.gz内にモデルが保存されています。
参考資料