前回の
SVMをJupyter Notebook上のscikit-learnで試す
の続編みたいな記事です。
これ以降は、
Jupyter NotebookをDockerを使って簡単にインストールし起動(nbextensions、Scalaにも対応) - Qiita
に従って準備したJupyter Notebookの環境で試しています。
このJupyterの環境でブラウザで8888番ポートにアクセスして、Jupyter Notebookを使うことができます。右上のボタンのNew > Python3をたどると新しいノートを開けます。
適当にランダムで作成したCSVファイル
https://github.com/suzuki-navi/sample-data/blob/master/sample-data-2.csv
も使っています。
データ確認
sample-data-2.csv で試してみます。
import pandas as pd
from sklearn import model_selection
df = pd.read_csv("sample-data-2.csv", names=["id", "target", "data1", "data2"])
こんなデータです。
%matplotlib inline
import matplotlib.pyplot as plt
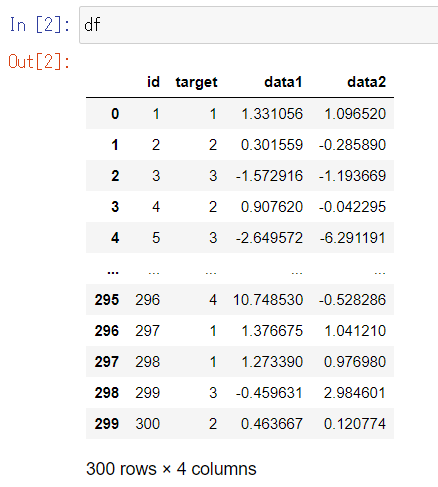
plt.scatter(df["data1"], df["data2"])
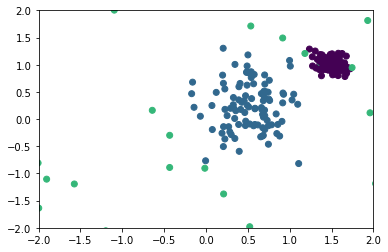
これだけだと伝わらないのですが、4つのクラスタがあります。4つがわかるようにこのあと散布図をだらだら並べますが、先に要約すると、次の4つです。
A. まわりに散らばってる少量のデータ
B. 中央付近の大きなデータの塊
C. 中央付近のさらに中心部に小さくまとまっているデータの塊
D. 中央付近のさらに中心部のやや右上にごく小さく固まっているデータの塊
以下、色分けした散布図です。
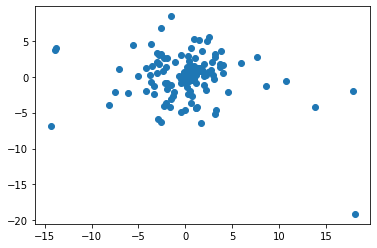
plt.scatter(df["data1"], df["data2"], c = df["target"])
大多数の中央付近のデータ(B緑, C青, D紫)と、少ないですがまわりに散らばっているデータ(A黄)とがあります。
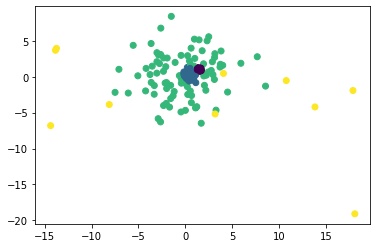
中央付近を拡大してみます。
plt.xlim(-5, 5)
plt.ylim(-5, 5)
plt.scatter(df["data1"], df["data2"], c = df["target"])
緑のエリアの中には2つの濃い塊(C青, D紫)があります。
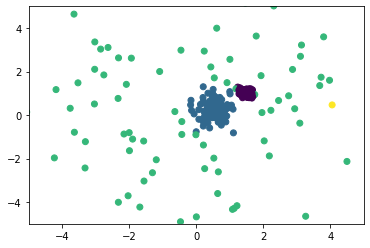
さらに大きく拡大してみます。
plt.xlim(-2, 2)
plt.ylim(-2, 2)
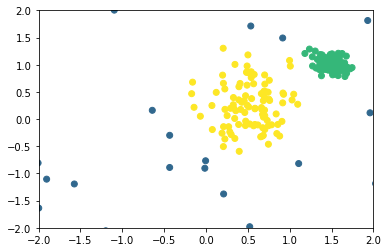
plt.scatter(df["data1"], df["data2"], c = df["target"])
中心部がC青で右上がD紫です。
このデータを生成した以下のコードを見ていただければわかるのですが、色ごとに正規分布でランダムに分布させたのを4色重ね合わせただけです。
この4色を教師なしでクラスタリングしたいです。
K-meansを試してみる
混合ガウスモデルの前に、同じく教師なしクラスタリングのK-meansを試してみます。データの分布状況からしてK-meansでは無理そうなのですが、やりかたを把握するためにいちおう。
とりあえず
feature = df[["data1", "data2"]]
target = df["target"]
K-meansのモデルで学習します。クラスタ数は4つと指定しておきます。
from sklearn import cluster
model = cluster.KMeans(n_clusters=4)
model.fit(feature)
参考
sklearn.cluster.KMeans — scikit-learn 0.21.3 documentation
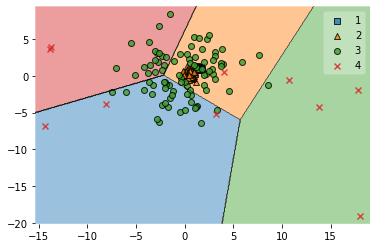
mlxtendというパッケージに含まれるplotting.plot_decision_regionsを使うとどのように分類しているのかを散布図で視覚化してくれます。plot_decision_regionsにはPandasのオブジェクトではなくNumPyの配列を渡す必要がありますので、to_numpy()というメソッドで変換します。
from mlxtend import plotting
plotting.plot_decision_regions(feature.to_numpy(), target.to_numpy(), clf=model)
見てのとおり、やはりまったくだめでした。
参考
plot_decision_regions - Mlxtend.plotting - mlxtend
pandas.DataFrame.to_numpy — pandas 0.25.3 documentation
混合ガウスモデルを試してみる
本題の混合ガウスモデルを試してみます。
参考
sklearn.mixture.GaussianMixture — scikit-learn 0.21.3 documentation
from sklearn.mixture import GaussianMixture
model = GaussianMixture(n_components=4, covariance_type='full')
model.fit(feature)
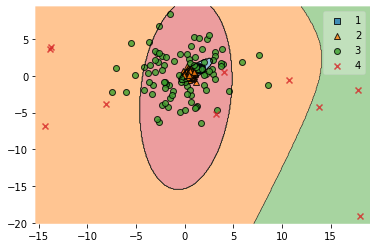
plotting.plot_decision_regions(feature.to_numpy(), target.to_numpy(), clf=model)
うーん、期待していたほどではない・・・
plot_decision_regionsでの拡大の仕方がわからなかったので普通にmatplotlibで分類結果の中央部を見てみます。
pred = model.predict(feature)
plt.xlim(-5, 5)
plt.ylim(-5, 5)
plt.scatter(feature["data1"], df["data2"], c = pred)
中心部の2つの塊が1つにまとまってしまいました。
学習にランダムな要素があるので、なんどか試してみたのですが、中心部の2つの塊を分離することはなく、それ以外のところは毎度バラバラな境界線を描くようです。
独自(?)ロジック
唐突ですがここで、正規分布に従うクラスタを分離できるように、K-meansをもとに独自にコードを書いてみました。
独自のコードはとりあえずScalaで書いたのですが、詳細はここでは省略します。余裕があったら別の記事で紹介します。
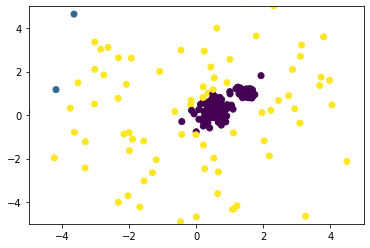
独自のコードでクラスタリングした結果を pred1.csv に保存して、散布図で見てみます。
pred1 = pd.read_csv("pred1.csv", names=["pred"])
plt.scatter(feature["data1"], feature["data2"], c = pred1["pred"])
よさそうです。
中央部を拡大します。
plt.xlim(-5, 5)
plt.ylim(-5, 5)
plt.scatter(feature["data1"], feature["data2"], c = pred1["pred"])
plt.xlim(-2, 2)
plt.ylim(-2, 2)
plt.scatter(feature["data1"], feature["data2"], c = pred1["pred"])
4つにきれいに分離できました。
経緯
K-meansではきれいに分離できないこのサンプルデータのケースで教師なしで分離したいってのがこの記事の動機ではあったのですが、記事の順番は違っていまして、混合ガウスモデルが最初よくわからなくて、さきに独自ロジックを書きました。K-meansを改良したものです。
独自ロジックであまりにもきれいに分離できて感動したのですが、きっと既知のアルゴリズムを再発明したにすぎないと思って @stkdev さんに相談したら、混合ガウスモデルでできると指摘を受けたものです。混合ガウスモデルのアルゴリズムがまだ理解しきれていなくて、私が書いた独自ロジックと同じものなのかどうかはよくわかっていないのです。同じかもしれないですが、少なくとも似てそうだとまでは思ってます。
ただscikit-learnで混合ガウスモデルで試してみてもいまいちきれいに出なかったのは、チューニングの問題なのか、これもよくわかってないです。
独自ロジックは余裕があったら別に投稿します。