今回は{caret}パッケージの基本的な使い方をまとめようと思います。
caretとは、Classification And Regression Trainingを略したもので、
機械学習に必要な
・データの分割(trainデータとtestデータの分割)
・前処理(ダミー変数化、少数の特定の値しかとらない連続変数の処理、相関のある特徴量の削除、線形関係のある特徴量の削除、scalingなど)
・特徴量選択
・モデルの訓練・評価(グリッドサーチなど)
の一連の操作を簡単に行えるパッケージです。非常に多くのアルゴリズムがサポートされています。
英語版の解説サイトおよびそれを和訳してくださっている記事があり、参考にさせていただいています。
背景
今回は{mlbench}パッケージのSonar データを利用します。
このデータはGormanとSejnowskiが行った、ソナー信号を利用して金属(Metal)と岩(Rock)を分類しようとした実験のデータで、V1~V60のデータは金属もしくは岩に当てたソナーから帰ってきたエネルギーを表しているらしいです。詳しくは、mlbenchを参考にしてください。
今回は{randomForest}パッケージを使用して、金属(Metal)と岩(Rock)を上記の60種類の特徴量を使って分類してみます。
使用するpackageの読み込み
まず、使用するパッケージを読み込みます。
library(tidyverse)
library(caret)
library(doParallel)
library(tictoc)
library(mlbench)
library(e1071)
library(modelr)
データの読み込みとデータ内容の確認
data(Sonar)
Sonar %>% head()
## V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
## 1 0.0200 0.0371 0.0428 0.0207 0.0954 0.0986 0.1539 0.1601 0.3109 0.2111
## 2 0.0453 0.0523 0.0843 0.0689 0.1183 0.2583 0.2156 0.3481 0.3337 0.2872
## 3 0.0262 0.0582 0.1099 0.1083 0.0974 0.2280 0.2431 0.3771 0.5598 0.6194
## 4 0.0100 0.0171 0.0623 0.0205 0.0205 0.0368 0.1098 0.1276 0.0598 0.1264
## 5 0.0762 0.0666 0.0481 0.0394 0.0590 0.0649 0.1209 0.2467 0.3564 0.4459
## 6 0.0286 0.0453 0.0277 0.0174 0.0384 0.0990 0.1201 0.1833 0.2105 0.3039
Class
## 1 R
## 2 R
## 3 R
## 4 R
## 5 R
## 6 R
V11~V60は長いので省略します。
データの可視化
df <-
Sonar %>%
gather(key = key,value = value,-c(Class)) %>%
group_by(key) %>%
mutate(ScaledValue=(value-min(value))/(max(value)-min(value)))
このコードの意味は以下の記事を参考にしてください。
tidyr::gather( )をとggplot2::facet_wrap()使って目的変数vs説明変数の散布図の一覧を描く
箱ひげ図を描いてみます。
ggplot(df,aes(x=Class,y=ScaledValue))+
theme_bw()+
geom_boxplot()+
geom_jitter(aes(color=Class),alpha=.3)+
facet_wrap(~key)
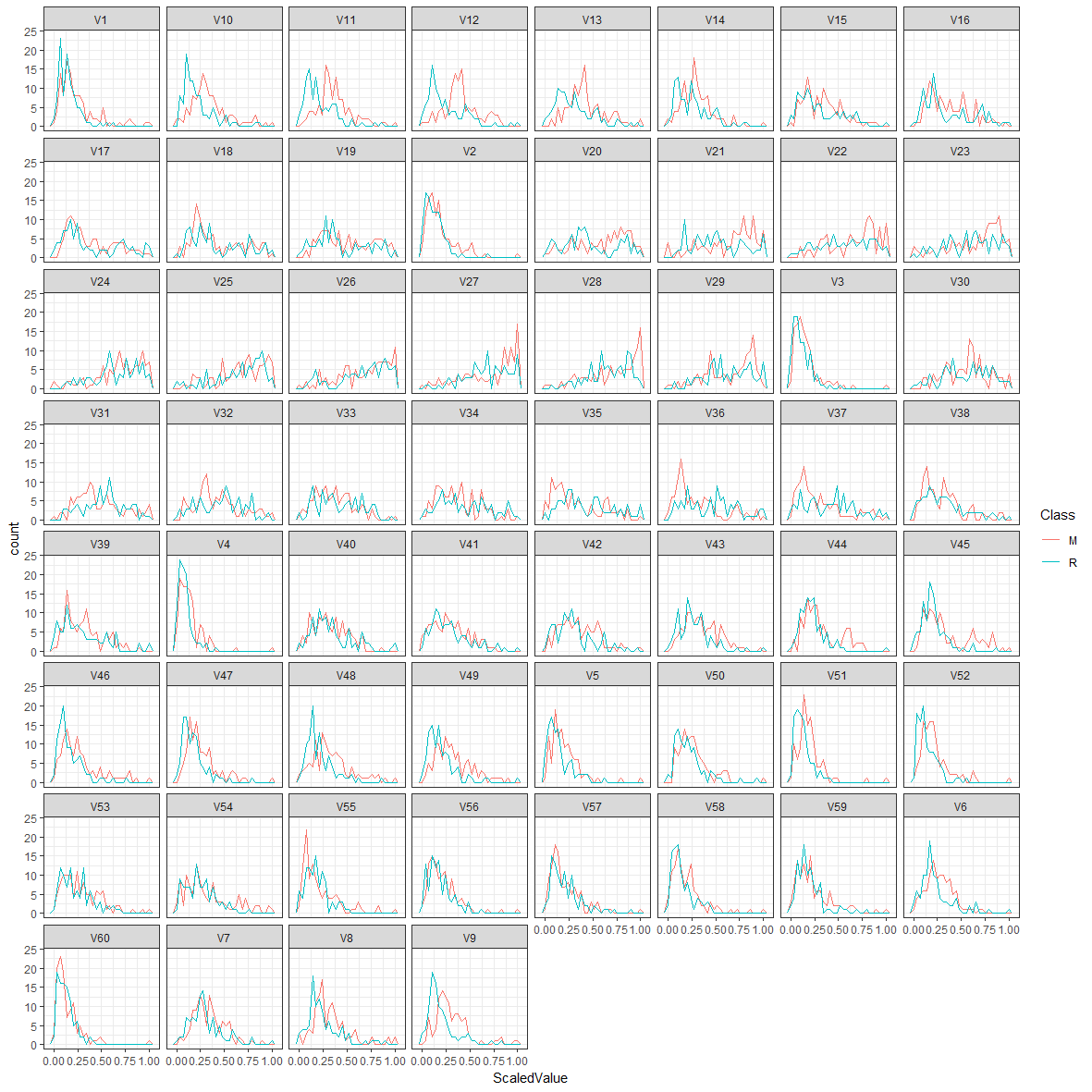
geom_freqpoly()も描いてみます。
ggplot(df,aes(color=Class,x=ScaledValue))+
theme_bw()+
geom_freqpoly()+
facet_wrap(~key)
これらの図から、V11、V12などは金属(Metal)か岩(Rock)かによって特徴量の分布が異なるので、分類に役立ちそうだと予想できますね。
対数変換したら予測精度が上がるかもしれないという印象も持ちます。
訓練データとテストデータに分ける
分類を行う際には、層化抽出を行います。
今回で言えば、金属と岩の比率を訓練データとテストデータで同じ割合にします。
層化抽出を行うには、**createDataPartition()**を使用します。
訓練データ:テストデータ=75:25に分けます。
set.seed(423)
inTrain <- createDataPartition(y = Sonar$Class,p = .75,list = F)
df_Train <- Sonar[inTrain,]
df_Test <- Sonar[-inTrain,]
{rf}methodのtuning可能なパラメータを確認
{randomForest}パッケージを使用する際に、caretでは
mothod="rf"
で指定します。
各手法の指定の方法(必要なpackcageや、method引数に指定する文字列、分類に使用できるか回帰に使用できるか、tuningできるパラメータはなにか)は、このページに書いてあります。以下の方法でも確認できます。
まず、{rf}methodでチューニングできるパラメータを**modelLookup()**で確認します。
modelLookup("rf")
## model parameter label forReg forClass probModel
## 1 rf mtry #Randomly Selected Predictors TRUE TRUE TRUE
チューニングできるパラメータはmtry(決定木群を作成する際に、いくつの特徴量を選択するか)で、label列にそのパラメータの説明があります。
また、forRegは回帰に使用できるか、forClassは分類に使用できるかを表しています。
grid_searchで最適なハイパーパラメータを決める
Rで機械学習するならチューニングもグリッドサーチ関数orオプションでお手軽に
こちらのサイトでもチューニングについて解説されています。
今回はmtryのみをチューニングします(しかできないという表現が正しい?)。
mtryを1から8にして訓練したときのモデルを比較して、最適なモデルを選択します。
ここではチューニングするハイパーパラメータが1つなので、**expand.grid()**を使う必要はないのですが、一応使ってみます。
grid <-
expand.grid(mtry=1:8)
grid
## mtry
## 1 1
## 2 2
## 3 3
## 4 4
## 5 5
## 6 6
## 7 7
## 8 8
trainControlでモデルの評価方法を決める
mtryを1から8にしたときの8つのモデルから、最適なモデルを選ぶ際の、評価条件を指定します。
**trainControl()**を使用します。
機械学習アルゴリズム〜caretパッケージ〜
こちらのページが詳しく解説されています。
今回は、繰り返し10分割交差検証(10-fold cross validation)を行います。
selectionFunction引数は、モデルを選ぶ際の基準を指定する引数です。"best"は指定した8つのモデルの中で最高精度を示したモデルを選択します。
今回使用している"oneSE"は標準的な誤りの範囲(1SE)以内でもっとも単純なモデルを選択します。
複雑なモデルはデータに過学習している可能性があり、同程度の精度であれば単純なモデルの方が汎化性能が高いという前提をもとにした選択方法です。1SEルールというらしいです。
fitControl <-
trainControl(method = "repeatedcv",
number = 10,#10-fold
repeats=3,#3回繰り返す
selectionFunction = "oneSE")
並列演算で訓練
ランダムフォレストは個々の決定木を独立させて学習するので、並列で処理させて訓練を高速化できます。
{doParallel}packageで複数コアを使用して計算させます。
{tictoc}packageは、処理にかかった時間を計算してくれます。
※この処理を実行する際に、なぜか{e1071}packageを要求されたので、必要かどうかはわからないのですが、一応読み込んでいます。
※私のパソコンのコア数が4コアだったので、makePSOCKcluster(4)としています。
**train()**を使用します。
set.seed(423)
cl <- makePSOCKcluster(4)
registerDoParallel(cl)
tic()
model_rf <- train(Class~.,
data=df_Train,
mothod="rf",
trControl=fitControl,
tuneGrid=grid)
toc()
stopCluster(cl)
## 27.4 sec elapsed
この処理に27.4秒かかっています。
モデルの確認
model_rf
## Random Forest
##
## 157 samples
## 60 predictor
## 2 classes: 'M', 'R'
##
## No pre-processing
## Resampling: Cross-Validated (10 fold, repeated 3 times)
## Summary of sample sizes: 140, 141, 142, 142, 142, 141, ...
## Resampling results across tuning parameters:
##
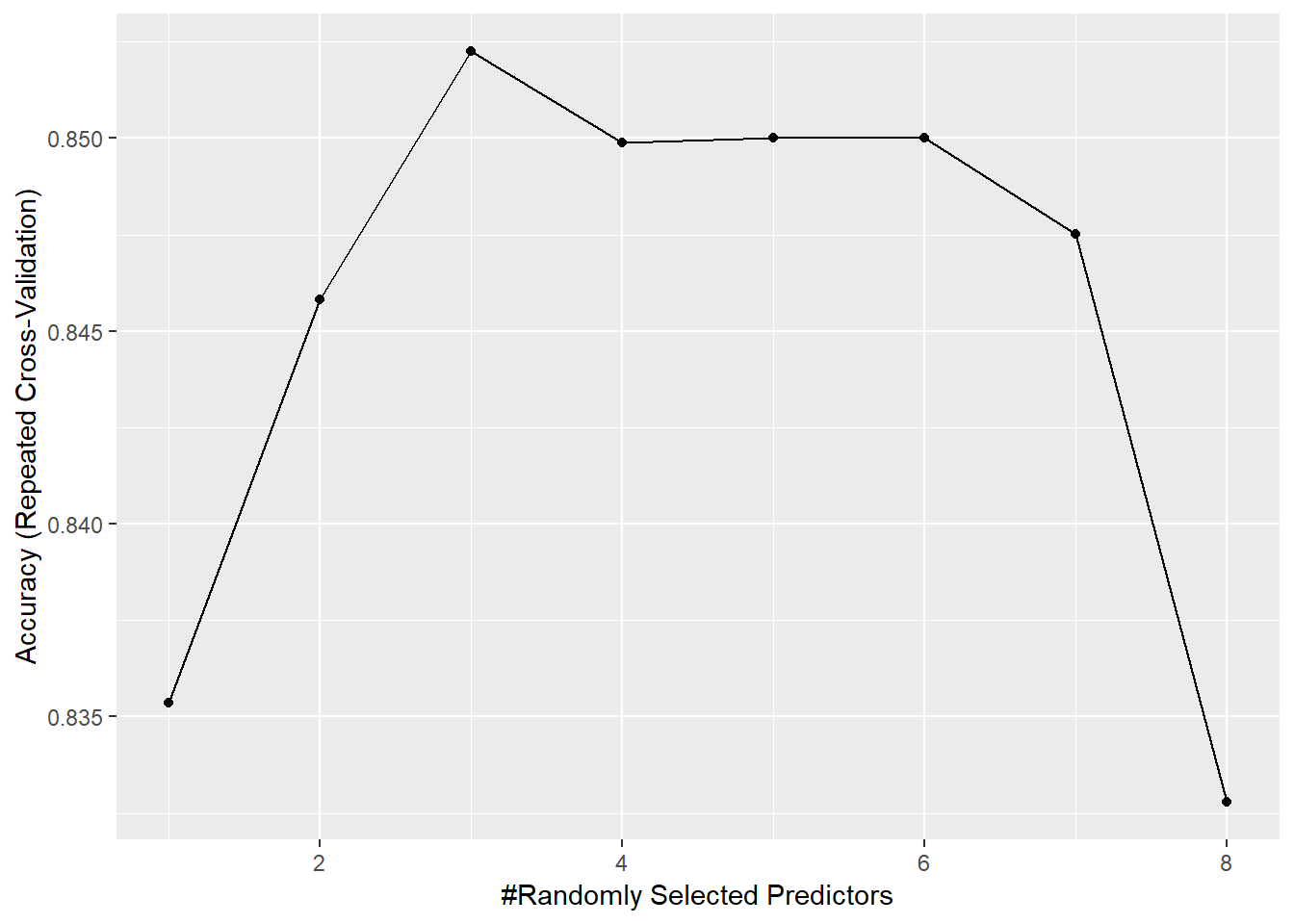
## mtry Accuracy Kappa
## 1 0.8353513 0.6648858
## 2 0.8458170 0.6862801
## 3 0.8522549 0.7000312
## 4 0.8498775 0.6961437
## 5 0.8500163 0.6972934
## 6 0.8500163 0.6967044
## 7 0.8475163 0.6917979
## 8 0.8327778 0.6620027
##
## Accuracy was used to select the optimal model using the one SE rule.
## The final value used for the model was mtry = 2.
訓練したモデルの情報が表示されます。
手法はRandom Forest
サンプルは157個
特徴量は60種類
分類したクラスは"M"と"R"の2種類
などです。
結果として、mtry=2のモデルが選ばれています。Accuracy、Kappaでベストなのはmtry = 3なのですが、1SEルールによりmtry = 2のモデルが選択されています。
訓練結果を**plot()もしくはggplot()**で表示できます。
ggplot(model_rf)
変数重要度の確認
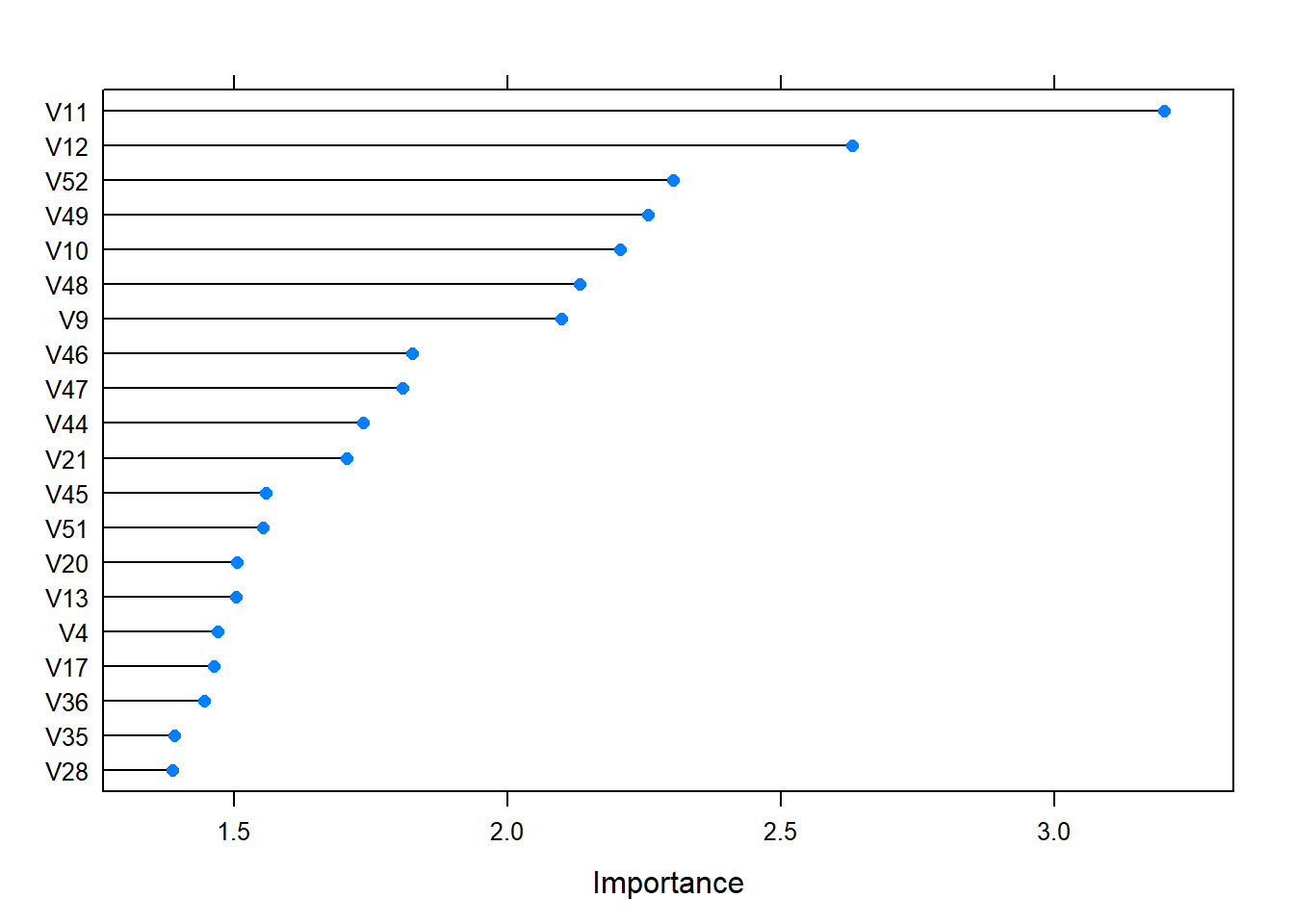
ランダムフォレストは、特徴量の重要度を計算できるアルゴリズムなので、確認します。
**varImp()**を使用します。上位20この特徴量の重要度を表示します。
varImp(model_rf,scale = F) %>%plot(top=20)
上記のboxplotやfreqpolyから予想していたように、V11やV12の重要度が高くなっていますね。
訓練したモデルで予測
訓練したモデルで新しいデータを予測する際には**predict()**を使用します。
pred_rf <- predict(model_rf,newdata = df_Test)
pred_rf
## [1] M M M R R M R M R R M R M R R R R R R R M M R R M M R M R M M M M M M
## [36] M M M M M R M M M M M M M M M M
## Levels: M R
テストデータでの精度の確認
混同行列を計算します。
**confusionMatrix()**で計算できます。
(参考)混同しやすい混同行列
confusionMatrix(data = df_Test$Class, pred_rf)
## Confusion Matrix and Statistics
##
## Reference
## Prediction M R
## M 24 3
## R 9 15
##
## Accuracy : 0.7647
## 95% CI : (0.6251, 0.8721)
## No Information Rate : 0.6471
## P-Value [Acc > NIR] : 0.05035
##
## Kappa : 0.5211
## Mcnemar's Test P-Value : 0.14891
##
## Sensitivity : 0.7273
## Specificity : 0.8333
## Pos Pred Value : 0.8889
## Neg Pred Value : 0.6250
## Prevalence : 0.6471
## Detection Rate : 0.4706
## Detection Prevalence : 0.5294
## Balanced Accuracy : 0.7803
##
## 'Positive' Class : M
##
{modelr}packageで予測結果の確認
**predict()と違い、{modelr}packageのgather_predictions()**を使うと、元データのデータフレームに予測結果を追加した形で結果を返してくれます。
Sonar_pred <-
Sonar %>% gather_predictions(model_rf,.pred="rf_pred") %>%
mutate(Judge=if_else(rf_pred==Class,"CORRECT","WRONG")) %>%
select(Judge,rf_pred,Class,everything())
Sonar_pred %>%head()
## Judge rf_pred Class model V1 V2 V3 V4 V5 V6
## 1 CORRECT R R model_rf 0.0200 0.0371 0.0428 0.0207 0.0954 0.0986
## 2 CORRECT R R model_rf 0.0453 0.0523 0.0843 0.0689 0.1183 0.2583
## 3 WRONG M R model_rf 0.0262 0.0582 0.1099 0.1083 0.0974 0.2280
## 4 CORRECT R R model_rf 0.0100 0.0171 0.0623 0.0205 0.0205 0.0368
## 5 CORRECT R R model_rf 0.0762 0.0666 0.0481 0.0394 0.0590 0.0649
## 6 CORRECT R R model_rf 0.0286 0.0453 0.0277 0.0174 0.0384 0.0990
※V7~V60は省略
今回は予測が当たっていたかを示すJudge列を追加しています。
モデル改善
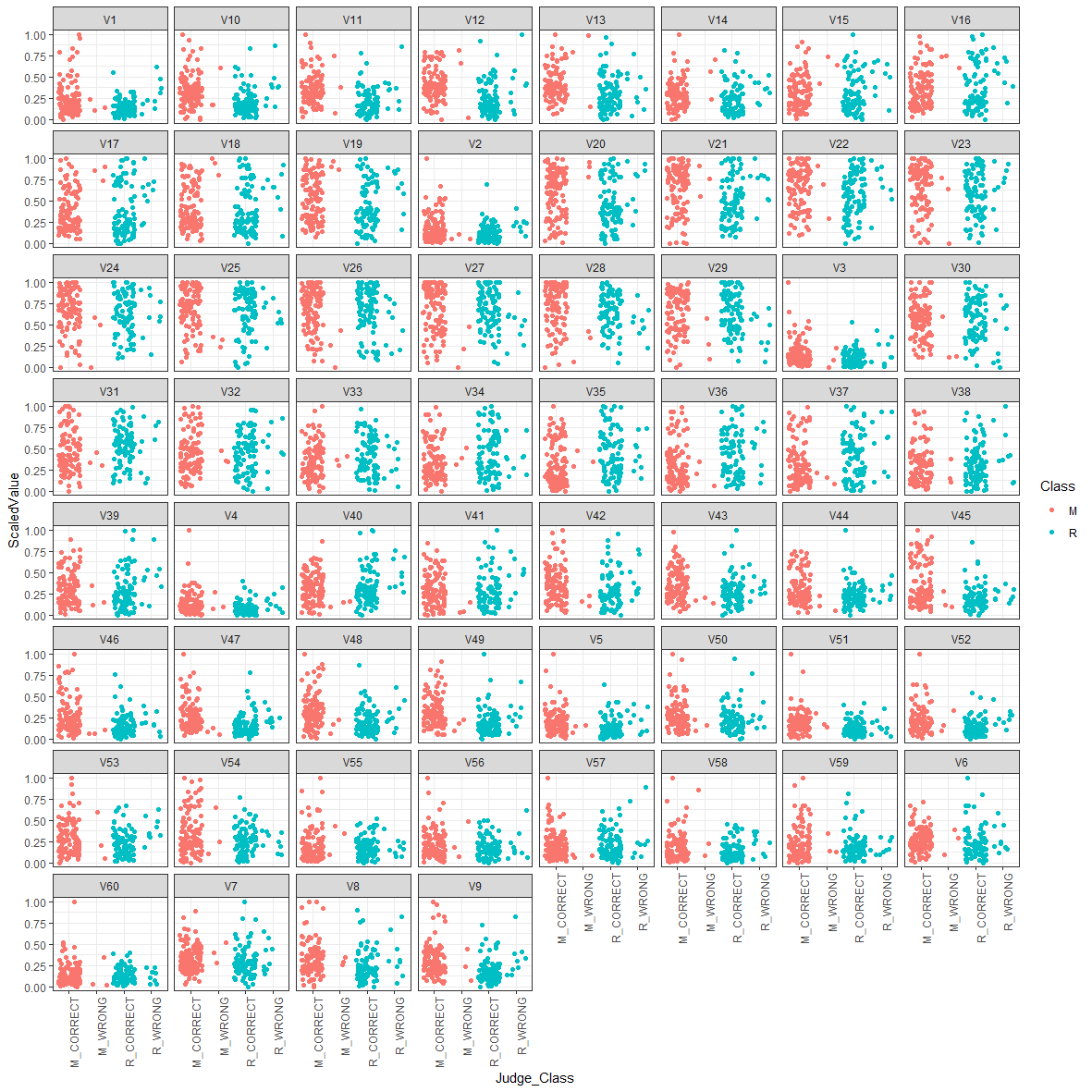
モデルの改善を試みるために分類が間違っていたサンプルを確認します。
Sonar_pred2 <- Sonar_pred %>%
gather(key = key,value = value,-c(Judge,rf_pred,Class,model)) %>%
mutate(Judge_Class=str_c(Class,"_",Judge)) %>%
group_by(key) %>%
mutate(ScaledValue=(value-min(value))/(max(value)-min(value)))
Sonar_pred2%>%head()
## # A tibble: 6 x 8
## # Groups: key [1]
## Judge rf_pred Class model key value Judge_Class ScaledValue
## <chr> <fct> <fct> <chr> <chr> <dbl> <chr> <dbl>
## 1 CORRECT R R model_rf V1 0.02 R_CORRECT 0.136
## 2 CORRECT R R model_rf V1 0.0453 R_CORRECT 0.323
## 3 WRONG M R model_rf V1 0.0262 R_WRONG 0.182
## 4 CORRECT R R model_rf V1 0.01 R_CORRECT 0.0627
## 5 CORRECT R R model_rf V1 0.0762 R_CORRECT 0.551
## 6 CORRECT R R model_rf V1 0.0286 R_CORRECT 0.200
ggplot(Sonar_pred2,aes(x=Judge_Class,y=ScaledValue,group=Judge_Class))+
theme_bw()+
geom_jitter(aes(color=Judge_Class),alpha=.3)+
facet_wrap(~key)
うむー。
よくわからない・・・。
何か良いアイデア、手法があれば追加したいと思います!
(参考サイト)
caret解説(英語)
caret解説の和訳_Rで学ぶベイズ統計学
2018/12/25