サイトをいくつか探して実行してみましたが、次の2つのサイトではうまく実行できました。
1https://colab.research.google.com/github/sokrypton/ColabFold/blob/main/AlphaFold2.ipynb
2https://colab.research.google.com/github/sokrypton/ColabFold/blob/main/beta/AlphaFold2_advanced.ipynb
実際に使ってみる
AlphaFold2のGoogle Colab版サイトを開きます(上記の1番目)
サンプルのアミノ酸配列が表示されているので、お試しでよければそのまま実行することもできます。
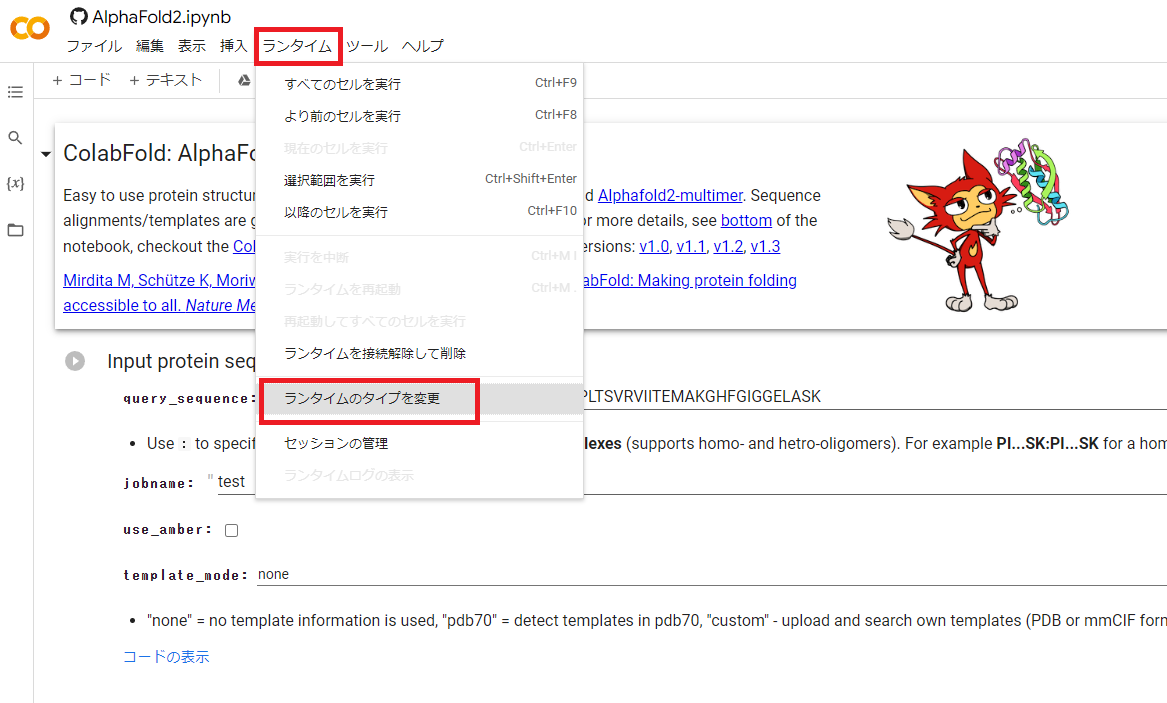
1.実行する前に ランタイムよりランタイムのタイプを変更を選択
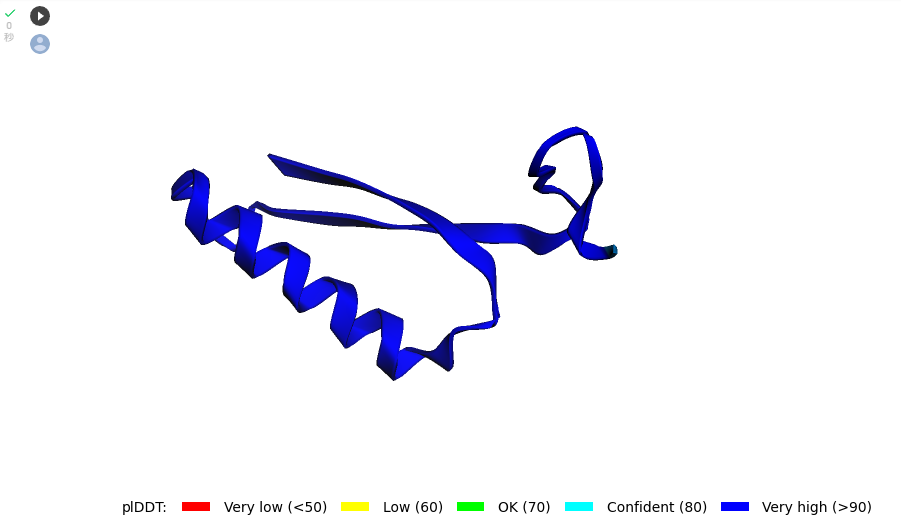
データはダウンロードフォルダにjobname-****.result.zipで保存されます。
実行内容詳細
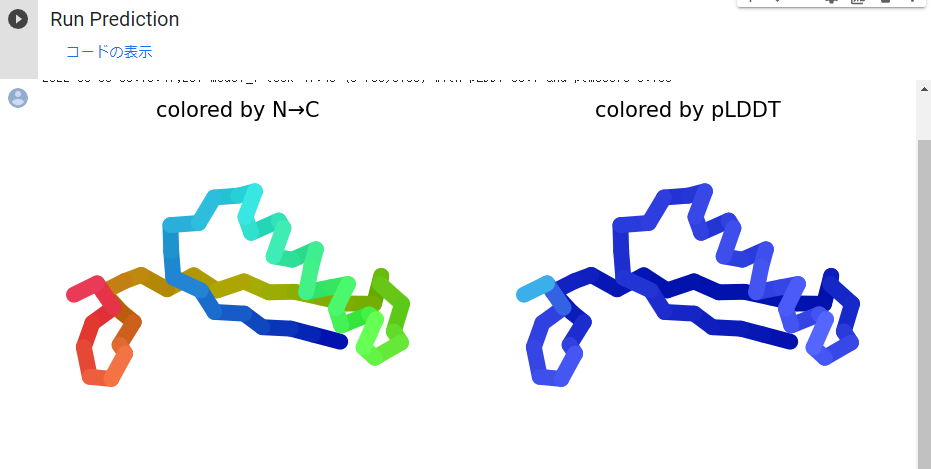
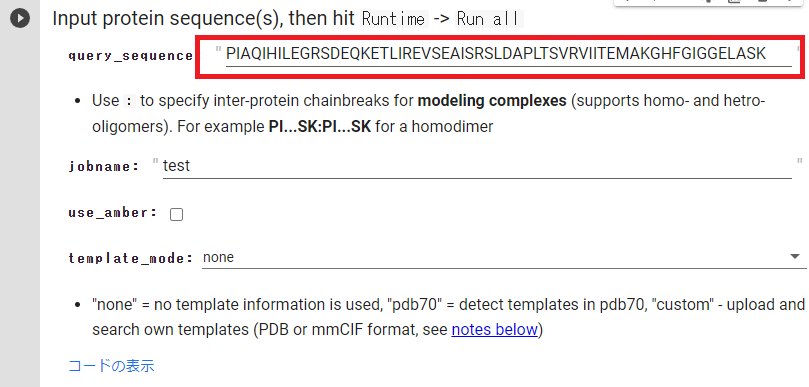
Input protein sequence
- query_sequence:アミノ酸配列を入力(タンパク質内の鎖切断は/ タンパク質間の鎖切断は:を指定)
- jobname: 好きな名前を入力
- use_amber:
- template_mode:none テンプレート情報を使用しない
:custom 独自のテンプレートを使用
※アミノ酸配列はPDBのサイトより入手できます
PDBサイトにアクセスし、PDB ArchiveにPBD IDかタンパク質名を入れて検索します
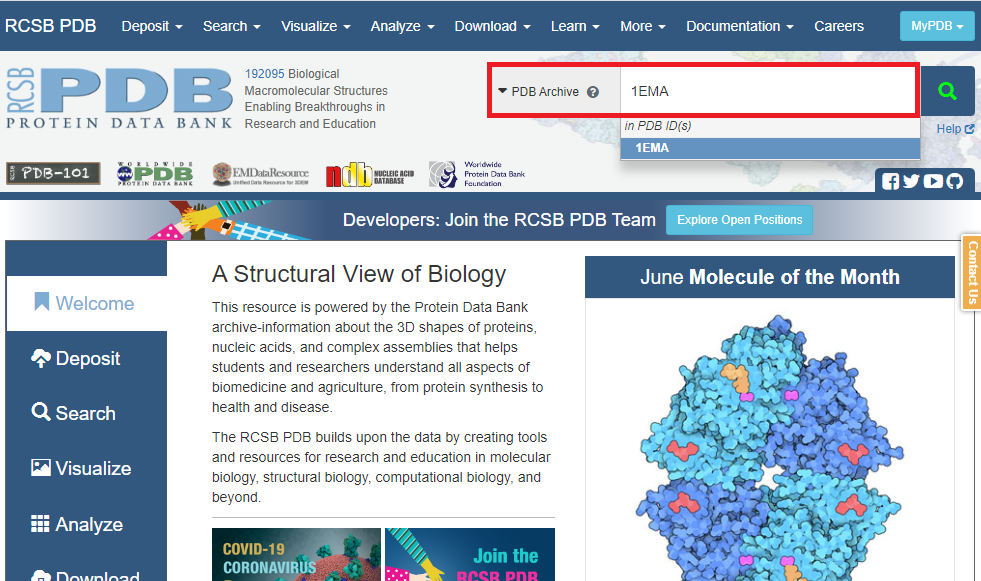
Display FilesよりFASTA Sequenceを選択
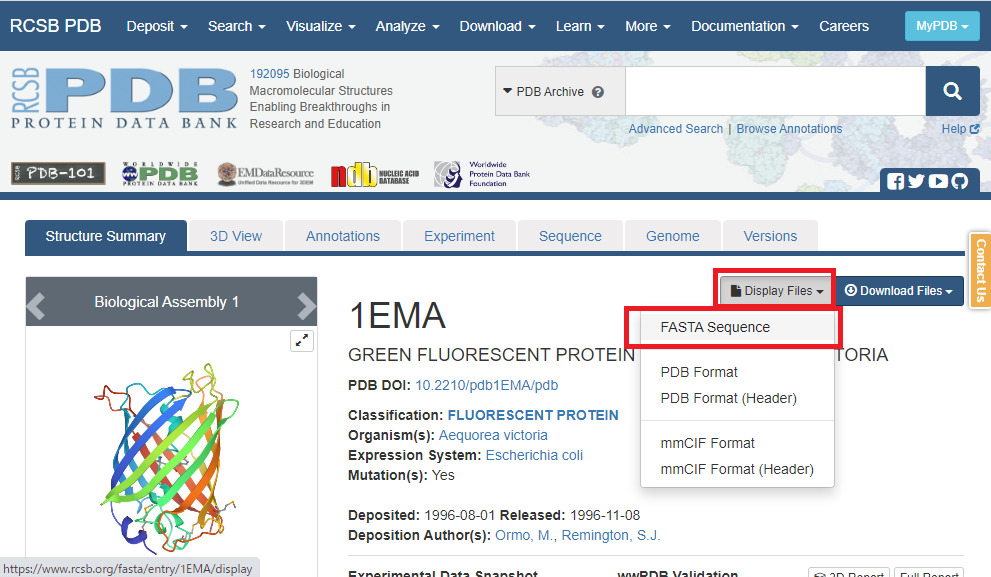
アミノ酸配列が表示されるので、コピーします。
※1行目は説明文になってますので、2行目以降をコピー

MSAのオプション指定
- Msa_mode:mmseqs2
- Pair_mode:unpaired+paired・・・同一種からのペア配列
:paired・・・ペア配列のみを使用
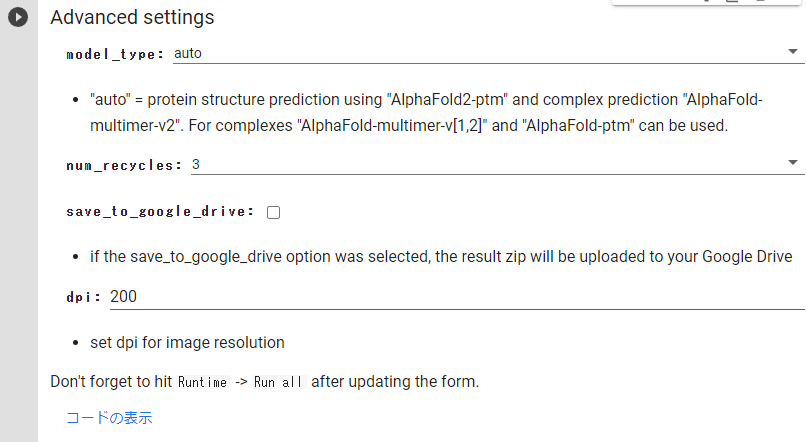
Advanced Settings
- model_type:auto・・・-ptm "を用いたタンパク質構造予測
:AlphaFold-multimer-v2・・・phaFold-multimer-v2" を用いたタンパク質構造予測 - num_recycles:
- save_to_google_drive:結果のzipファイルがgoogle driveにアップロード
- dpi:画像の解像度
今回参考にさせていただいたサイトは
http://cedro3.com/ai/alphafold2/
https://togotv.dbcls.jp/en/20220517.html