ついにPandasの1.0.0rc版が公開されたようなので、早速アップデート内容の確認や挙動を試していきたいと思います。
元のツイート :
Pandas 1.0.0rc0 is now available for testing! Please try it out and report any issues.https://t.co/iTjBtzy3WA
— pandas (@pandas_dev) January 10, 2020
TLDR
極端にがらっと変わったという感じでもありませんが、細かいところが色々とたくさんブラッシュアップされたような印象を受けました。
- 整数の列に欠損値が含まれていた際に、カラムの型を整数のまま保持するためのpd.NAのシングルトンが追加された。
- 文字列のカラムがobjectではなく文字列専用の型を指定できるようになった。
- 真偽値のカラムでも、カラムの型を維持したまま欠損値を含められるようになった。
- 窓関数でのapplyで高速なNumbaを利用できるようになった。
- データフレームを直接マークダウンで出力できるようになった。
- 文字列変換(to_string)で、文字列のカラムの最大文字数を簡単に調整できるようになった。
- Json変換(to_json)で、インデントと改行を付与して見やすい形で変換できるようになった。
- ソート処理(sort_values・sort_index)や重複削除の処理(drop_duplicates)で同時にインデックスのリセットができるようになった。
- pickleの読み込みがURLの指定をサポートした。
- 数値計算や比較など様々なパフォーマンスが改善した。
- 170件くらいのバグのissueの修正がマージされた
- etc
試す環境とインストール周り
使うもの :
- Windows10
- Anaconda
- Python3.7
- Jupyter notebook (jupyterlab-lspとかの影響なのか、LabだとなぜかPandas使おうとするとフリーズしたので普通のJupyterを使っていきます)
試すための環境をAnacondaで作っておきます(Pandas公式でもAnacondaとかが手っ取り早いよと書いてあるので)。
Pythonバージョンは、3.6.1以降をサポートしているそうなので、今回は3.7を使っていきます。
$ conda create -n pandas1.0.0rctest python=3.7 anaconda
環境を有効化します。
$ conda activate pandas1.0.0rctest
これだけだとpandas==0.25.3がインストールされている(0.x.x系の最終バージョン?)ので、pipで1.0.0rcにアップデートします。
Pandasのメーリングリストの内容を参考にします。
$ python -m pip install --upgrade --pre pandas==1.0.0rc0
...
Installing collected packages: pandas
Found existing installation: pandas 0.25.3
Uninstalling pandas-0.25.3:
Successfully uninstalled pandas-0.25.3
Successfully installed pandas-1.0.0rc0
Jupyter上でimportでエラーにならないこととバージョンの表示・最低限のデータフレームの作成など動くことを確認します。
>>> import pandas as pd
>>> pd.__version__
'1.0.0rc0'
>>> df = pd.DataFrame(data=[{'a': 100, 'b': 200}, {'a': 300, 'b': 400}])
>>> df
とりあえずは無事インストールできたようです。
事前の注意事項
1.0.0になって、多くのDeprecationWarningに指定されていたものが切り落とされたそうです。
実務で使う際などには、必要に応じて先に0.25.3にアップデートしてみて、テストなどを流してみてDeprecationWarningやFutureWarningが出ていない(1.0.0以降で切り落としになっているAPIを参照していない)ことを事前にご確認ください。
整数における欠損値の型の追加 : pd.NA
現状、Pandasでは複数の欠損値が存在しています。
例えば、floatのカラムに対してはnp.nanが、objectの型のカラムにはnp.nanもしくはNoneが、そして日時のカラムにはpd.NaTといった具合です。
今回のバージョンから、pd.NAというシングルトンが追加されます。主に整数や真偽値・新しい文字列の型(後述)などのカラムで使われるそうです。
以下のようにdtype='Int64'と指定してデータフレームを作ると、Noneの値などが<NA>になります。
>>> import numpy as np
>>> df = pd.DataFrame(data=[{'a': 100, 'b': None}], dtype='Int64')
>>> df
dtypesを見ていると、欠損値を含んでいるにも関わらずカラムの型がint64を維持している(今まではNoneなどが含まれているとobjectになっていた)のが分かります。
>>> df.dtypes
a Int64
b Int64
dtype: object
※もしくは、文字列などの代わりにpd.Int64Dtypeなどを指定しても同じ挙動になります。
>>> df = pd.DataFrame(data=[{'a': 100, 'b': None}], dtype=pd.Int64Dtype())
>>> df.dtypes
a Int64
b Int64
dtype: object
データフレーム内の値の型を確認してみると、NATypeという型になります。
>>> type(df.loc[0, 'b'])
pandas._libs.missing.NAType
注意点として、np.int64などとpd.Int64Dtype()などは挙動がずれるようです。
>>> df = pd.DataFrame(data=[{'a': 100, 'b': None}], dtype=np.int64)
>>> df
>>> df.dtypes
a int64
b object
dtype: object
>>> type(df.loc[0, 'b'])
NoneType
現状ではデフォルトではpd.NAは使われず、前述のようにpd.Int64Dtype()などを明示的に指定した際に使われるようになるとドキュメントに書かれていたので、挙動の差異は互換性を配慮したため、といったところでしょうか。
Currently, pandas does not yet use those data types by default (when creating a DataFrame or Series, or when reading in data), so you need to specify the dtype explicitly.
Experimental NA scalar to denote missing values
pd.NAの値と計算の演算子での挙動は、基本的に<NA>の値を引き継ぎつつ、一部で特殊な挙動をするものがあるようです。
<NA>を引き継ぐ例 :
>>> df = pd.DataFrame(data=[{'a': 100, 'b': None}], dtype=pd.Int64Dtype())
>>> pd_na = df.loc[0, 'b']
>>> pd_na + 1
<NA>
>>> pd_na / 2
<NA>
>>> 'a' * pd_na
<NA>
累乗の特殊な挙動をする例 :
あまり欠損値に対して累乗の計算をすることは少なさそうですが、累乗の場合は一部を除いて<NA>を継承したりはせず、整数などの値が返ります。
<NA>の0乗は1になります。
>>> pd_na ** 0
1
※ドキュメントでは0になっていますが、npも1が返るので1が正しい・・・のでしょうか?
<NA>の1乗は<NA>になるようです。
>>> pd_na ** 1
<NA>
1の<NA>乗は1になります。
>>> 1 ** pd_na
1
-1の<NA>乗も同様に-1になります。
>>> -1 ** pd_na
-1
その他の値の<NA>乗は<NA>になるようです。
>>> 2 ** pd_na
<NA>
この辺りの挙動はNumPyに近い形に合わせた、という感じでしょうか。
>>> np.nan ** 0
1.0
>>> 1 ** np.nan
1.0
>>> 2 ** np.nan
nan
比較演算子では<NA>を引き継ぐ
比較演算子ではすべて<NA>で返ってきます。
>>> pd_na == 1
<NA>
>>> pd_na == pd.NA
<NA>
>>> pd_na < 5
<NA>
返却値が真偽値じゃないのは結構特殊に感じるのと、NumPy側は真偽値で返されるので、NumPyとも挙動が変わるようです。
>>> np.nan == 1
False
pd.NAの欠損値かどうかの判定
isnaという関数で真偽値を取れます。昔からあったisnullと近い挙動になるようです(違いがぱっと見良くわからない・・・)。それぞれ、np.nanやNoneなどもTrueになります。
>>> pd.isna(pd_na)
True
>>> pd.isna(np.nan)
True
>>> pd.isna(300)
False
>>> pd.isnull(pd_na)
True
pd.NAの論理演算の挙動
Three-valued logicという、JuliaやRなどとも近いらしい挙動に準じているようです。
OR演算子の場合且つ片側がTrueであればTrueになります。
>>> pd_na | True
True
OR演算子の場合且つ片側がFalseの場合は<NA>になります。
>>> pd_na | False
<NA>
AND演算子の場合且つ片側がTrueの場合には<NA>になります。
>>> pd_na & True
<NA>
AND演算子の場合且つ片側がFalseの場合はFalseになります。
>>> pd_na & False
False
文字列専用のカラムの型が追加された
※こちらも、pd.NAと同様現在はまだ実験的な追加となるため将来内容が変更になる可能性があります。
今までは、文字列を含んだカラムではNumPyの挙動に合わせる形でobjectなどの型が割り振られていました(NumPyは別途固定長の文字列とかが絡んできたりしますが・・)。
>>> df = pd.DataFrame(data=[{'a': 'apple'}])
>>> df.dtypes
a object
dtype: object
今回のバージョンからpd.StringDtypeが追加され、明示的に「このカラムは文字列(とpd.NA)のみを格納する」という制御ができるようになりました。
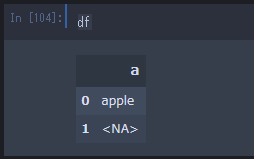
>>> df = pd.DataFrame(data=[{'a': 'apple'}, {'a': pd.NA}], dtype=pd.StringDtype())
>>> df
>>> df.dtypes
a string
dtype: object
主に以下のような意図で追加されたようです。
- カラムがobject型だとちょっとしたミスなどで文字列以外の値を入れれてしまう(今回追加した文字列のカラムの型には他の数値などを入れれない)。
- object型だと内容が分かりづらい(明示的に文字列だけのカラムなのかそうでは無いのか等)。
地味に嬉しい(より安全に文字列のカラムを扱える)アップデートですね。
試しに、以下のように数値を入れてみると、ちゃんと弾かれることが分かります。
>>> df.loc[0, 'a'] = 100
ValueError: Cannot set non-string value '100' into a StringArray.
なお、型の指定でpd.Int64Dtypeと'Int64'の指定のように、pd.StringDtypeだけでなく'string'と指定しても同様の挙動になります。
>>> df = pd.DataFrame(data=[{'a': 'apple'}, {'a': pd.NA}], dtype='string')
>>> df.dtypes
a string
dtype: object
シリーズに設定されるstr属性を経由して、色々操作できるのは今まで同様です。
>>> df.a.str.upper()
0 APPLE
1 NaN
Name: a, dtype: string
>>> df.a.str.split('l')
0 [app, e]
1 <NA>
Name: a, dtype: object
明示的な文字列の型が追加されたので、str属性関係で色々操作する際にはカラムの型でpd.StringDtypeを使うのが推奨されています(objectのカラムで扱って予期せぬ挙動を避けるため)。
真偽値のカラムでも、カラムの型を維持したまま欠損値を扱えるようになった
前述の整数と文字列同様、真偽値でも欠損値が<NA>の値で扱われて、カラムの型で真偽値の状態を維持できるようになりました。
dtypeに'boolean'と指定するか、pd.BooleanDtype()と指定することで使えます。
>>> df = pd.DataFrame(data=[{'a': True}, {'a': None}], dtype=pd.BooleanDtype())
>>> df
>>> df.dtypes
a boolean
dtype: object
窓関数で任意の関数を指定できるapplyで、高速なNumbaの利用ができるようになった
移動平均などを算出する際に利用するPandasの窓関数で、任意の関数を指定する際にNumba(NumPyなどのために作られた高速化のためのライブラリ)が指定できるようになりました。
条件を満たした場合に、Pythonで書いたapply用の関数がネイティブコードに近いレベルに速度が速くなります。
※Anaconda以外を利用している場合、Numba関係でのインストールなどが恐らく必要になります。
>>> df = pd.DataFrame(columns=['a'], index=np.arange(0, 10000))
>>> df['a'] = np.random.randint(low=0, high=101, size=df.shape)
>>> rolling = df.rolling(window=10)
applyに指定する関数は適当に、fという名前で用意しました。
def f(x):
return np.mean(x) + 5
apply関数でNumbaを利用する場合にはengine='numba', raw=Trueと引数を設定します。
%%timeit
rolling_df = rolling.apply(func=f, engine='numba', raw=True)
2.02 ms ± 97.4 µs per loop (mean ± std. dev. of 7 runs, 1 loop each)
なお、Numbaの特徴として、JIT(Just In Time)で処理されるという挙動をします。これは、対象の関数が最初に実行された際にコンパイルされ、その後はそのコンパイル後の状態で高速化された形で処理される都合、最初の1回だけ遅い(通常と変わらない速度)という特徴があります。
そのため、もう一度実行してみたりすると、%%timeitのマジックコマンドの±の部分が小さくなっている(もう一度コンパイルされたので、その分ぶれが少なくなっている)のが分かります。
%%timeit
rolling_df = rolling.apply(func=f, engine='numba', raw=True)
2.01 ms ± 38.2 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
Pandasを経由せずに直接Numbaを使う際には、Numbaのデコレーターを関数に付けたりしますが、そのデコレーターへの引数はengine_kwargsで指定できます。
例えば、Numbaで最適化できない行を含まないという制限を課すnopythonとかを指定する場合以下のようになります。
>>> rolling_df = rolling.apply(func=f, engine='numba', raw=True, engine_kwargs={'nopython': True})
nopython以外では、現状ではnogilとparallelをサポートしているそうです。
※Numbaに関しては以前「Pythonを速くするTIPS集(計測・ビルドインの各機能・Cython・Numba・etc)」で色々触れたので必要な場合そちらやドキュメントなどをご確認ください。
Numbaを指定しない形での普通のapplyを同条件で試してみると、大分遅くなります。
%%timeit
rolling_df = rolling.apply(func=f)
1.06 s ± 9.9 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
また、以前からあるCythonで試してみても、ケースバイケースだとは思いますが、Numbaの方が早くなりえます。
%%timeit
rolling_df = rolling.apply(func=f, engine='cython', raw=True)
63.3 ms ± 6.78 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
データフレームを直接マークダウンに変換できるようになった
今まではQiitaやGithubなどのマークダウンにデータフレームの内容を載せたい場合、Pandas以外の変換用のライブラリを挟むか、to_htmlでHTMLの状態で持ってくるなどが必要でした。
1.0.0からはto_markdownメソッドが追加されており、直接マークダウンの文字列を得ることができます。
・・・が、ドキュメントに書いてなかったのですが、実行してみたらtabulateの依存ライブラリが足りないよ、と出ました。
どうやらオプション扱いのライブラリらしく、現状自分で入れる必要があるようです。
$ pip install tabulate
...
Successfully installed tabulate-0.8.6
tabulatel入れた後はto_markdownメソッドを呼び出すだけです。
ただ、Jupyter上とかだとprintを挟まないと改行が\n表示となってしまうので、printを挟んでおきます。
df = pd.DataFrame(
data=[{
'a': 100,
'b': 200,
}, {
'a': 300,
'b': 400,
}])
print(df.to_markdown())
| | a | b |
|---:|----:|----:|
| 0 | 100 | 200 |
| 1 | 300 | 400 |
これでQiitaとかに持ってくるのがお手軽になりますね。素晴らしい![]()
to_stringでの文字列のカラムの最大文字数を指定できるようになった
通常、データフレームをJupyter上などで表示すると、文字列のカラムは一定も文字数で省略表示(...)になります(pd.set_optionなどで条件の調整はできます)。
df = pd.DataFrame(
data=[{
'a': 'aaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa'
'bbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbb'
'cccccccccccccccccccccccccccccccccccccc',
'b': 100
}])
df
一方で、今まではデータフレームのto_stringで文字列に変換した場合には全てが含まれる形で変換され、他の箇所にサンプルなどとして持ってくる場合に見づらいケースがありました。
>>> print(df.to_string())
a b
0 aaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaabbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbbcccccccccccccccccccccccccccccccccccccc 100
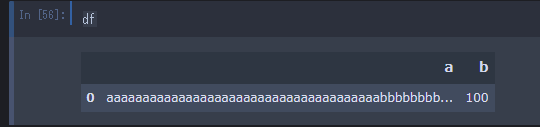
今回、max_colwidthのオプションが加わったため、任意の見やすい長さに直接調整できるようになりました。
>>> print(df.to_string(max_colwidth=30))
a b
0 aaaaaaaaaaaaaaaaaaaaaaaaaa... 100
JSON変換(to_json)で、インデント数が指定できるようになった
通常、データフレームをto_jsonで変換すると、1行のJSONになります。
df = pd.DataFrame(
data=[{
'a': 100,
'b': 200,
}, {
'a': 300,
'b': 400,
}])
>>> print(df.to_json())
{"a":{"0":100,"1":300},"b":{"0":200,"1":400}}
今回、indentというオプションが追加となり、インデントと改行が付与できるようになりました。
>>> print(df.to_json(indent=2))
{
"a":{
"0":100,
"1":300
},
"b":{
"0":200,
"1":400
}
}
to_stringやto_htmlにencodingのオプションが追加された
to_stringやto_htmlで、第一引数にファイルパスなどを指定してファイル保存するときのために、encodingのオプションが追加になりました。ただ、デフォルトではutf-8のようなので、utf-8以外を指定しないといけない特殊なケースを除いてあまり意識することは無いと思われます。
ファイル保存用なので、第一引数にファイルパスなどを指定しない場合はエラーで怒られます。
>>> df.to_html('./to_html_test.html', encoding='utf-8')
Stata のデータの読み込みのインターフェイスが追加された
Stataってなんだろう・・・と調べたら、統計分析用のソフトウェアらしいです。
Stata(ステータ、スタータ)は、1985年、StataCorp社により開発された統計分析のソフトウェアである。主に経済学、社会学、政治学、医学(臨床疫学)、疫学の分野で用いられている。
Stata - Wikipedia』
使ったことがないので割愛しますが、read_stataというインターフェイスが追加になったそうです。
sort_valuesで同時にインデックスのリセットができるようになった
今までソート(sort_values)を使うと、インデックスの値は元の値を維持するため、昇順になっていませんでした。
df = pd.DataFrame(
data=[{
'a': 100,
}, {
'a': 200,
}, {
'a': 300,
}])
df
>>> df.sort_values(by='a', inplace=True, ascending=False)
>>> df
locなどで使う際などにインデックスを再度割り振りたい場合、別途reset_indexを呼べば特に問題ないのですが、今回sort_valuesにignore_indexオプションが加わってよりシンプルに1行でいけるようになりました。
>>> df.sort_values(by='a', inplace=True, ascending=False, ignore_index=True)
>>> df
sort_indexで同時にインデックスのリセットができるようになった
これもsort_valuesと同様ですね。
インデックスの値の昇順や降順てソートした後に、インデックスを0からの昇順の値にリセットするためのオプション(ignore_index)が追加になりました。
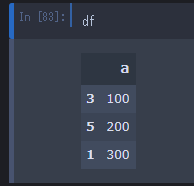
df = pd.DataFrame(
data=[{
'a': 100,
}, {
'a': 200,
}, {
'a': 300,
}],
index=[3, 5, 1])
df
>>> df.sort_index(inplace=True, ignore_index=True)
>>> df
drop_duplicatesの処理で同時にインデックスのリセットができるようになった
ソートと同様に、重複行などを切り落とすdrop_duplicatesでも、処理後にインデックスが飛び飛びになったりして、別途reset_indexが必要になるケースがありました。
こちらもignore_indexオプションが追加となり、同時にインデックスのリセットができるようになりました。
df = pd.DataFrame(
data=[{
'a': 100,
}, {
'a': 200,
}, {
'a': 100,
}, {
'a': 300,
}])
df.drop_duplicates(inplace=True, ignore_index=True)
df
pickleの読み込みがURLの指定をサポートした
read_pickleがURLの指定をサポートしたそうです。
ドキュメントにto_pickleも、と書かれていましたが、issueを見る感じread_pickleだけ?の気配がします。
少し手間がかかるので今回は検証をスキップしますが、S3などに設置したpickleの読み込みなども楽になりそうですね。
renameのオプションが厳密になった
インデックスやカラムのリネーム用のrename関数の引数の指定が厳密になりました。
普段columnsなどのキーワード引数を指定していたので私は特に影響を受けませんでしたが、今までは例えば以下のように2つの辞書の引数を指定して、且つそれらのキーで同じインデックスを指定しているケースが動いてしまっていたようです(0.25.xのバージョンなどではFutureWarningになっていたようです)。
df = pd.DataFrame(
data=[{
'a': 100,
}, {
'a': 200,
}, {
'a': 300,
}])
df.rename({0: 1}, {0: 2})
現在では、上記のコードはエラーで弾かれるようになっています。
TypeError: rename() takes from 1 to 2 positional arguments but 3 were given
同様に、第一引数に辞書を指定して別途indexやcolumnsの引数を指定するのが弾かれるようになりました。
>>> df.rename({0: 1}, index={0: 2})
TypeError: Cannot specify both 'mapper' and any of 'index' or 'columns'
infoメソッドの表示が見やすくなった
データフレームの情報を表示するinfoメソッドに、行番号や表のヘッダーなどが追加され、見やすくなりました。
0.25.xまでの表示 :
>>> df = pd.DataFrame({"int_col": [1, 2, 3],
... "text_col": ["a", "b", "c"],
... "float_col": [0.0, 0.1, 0.2]})
>>> df.info(verbose=True)
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 3 entries, 0 to 2
Data columns (total 3 columns):
int_col 3 non-null int64
text_col 3 non-null object
float_col 3 non-null float64
dtypes: float64(1), int64(1), object(1)
memory usage: 152.0+ bytes
※環境切り替えが手間なため、前述のコードはExtended verbose info output for DataFrameから引用。
バージョン1.0.0以降 :
df = pd.DataFrame(
data=[{
'fruit name': 'apple',
'fruit price': 200,
}, {
'fruit name': 'orange',
'fruit price': 300,
}, {
'fruit name': 'melon',
'fruit price': 500,
}])
df.info()
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 3 entries, 0 to 2
Data columns (total 2 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 fruit name 3 non-null object
1 fruit price 3 non-null int64
dtypes: int64(1), object(1)
memory usage: 176.0+ bytes
pd.Categoricalの最小値でnanが対象外になった
pd.Categoricalを使ったことがないので私は特に使っていないのですが、もし使われている方で且つ値にnanが入りうる実装の場合注意が必要そうです。
pd.Categoricalのインスタンスのminメソッドを実行した際に、0.25.xまでは値にnanが含まれていればnanが返却されていましたが、1.0.0からはnan以外で返却されるようです。
>>> categorical = pd.Categorical(values=[1, 2, np.nan], ordered=True)
>>> categorical
[1, 2, NaN]
Categories (2, int64): [1 < 2]
>>> categorical.min()
1
Seriesの初期化でdtypeを指定しないとDeprecationWarningになるようになった
将来のリリースで、dtypeを指定しない場合のSeriesの初期化はfloat64ではなくobjectの型になるそうです。
予期せぬ挙動を避けるため、今後は型を明示してくださいね、という感じでしょうか・・・。
この点は以外と実務でやっている気がする・・・
>>> sr = pd.Series()
C:\\...\Anaconda3\envs\pandas1.0.0rctest\lib\site-packages\ipykernel_launcher.py:1:
DeprecationWarning: The default dtype for empty Series will be 'object' instead of 'float64' in a future version. Specify a dtype explicitly to silence this warning.
Pythonのサポートしている最低バージョンと依存ライブラリのバージョンが上がった
Pythonは3.6.1以降のサポートとなり、NumPyは1.13.3以降、matplotlibは2.2.2以降、その他諸々の依存ライブラリバージョンが上がっています。
詳細はIncreased minimum versions for dependenciesの節などをご確認ください。
パフォーマンスの改善
数値計算や比較、range使用時のデータフレームの初期化、特定条件のreplaceなど、さまざまな点でパフォーマンスが良くなったそうです。
詳細はPerformance improvementsの節をご確認ください。
その他
バグのissueも170件くらい修正分がマージされているようです。
バグの節が果てしなく長い・・・感じですが、よりバグに遭遇しにくくなるのは素晴らしいことなので、Contributorの皆様にはとても感謝・・・という印象です。
大きなところは大体触れたと思いますが、全ては触れておらず一部割愛した内容もあるため、残りは必要な方は公式の資料をご確認ください。