はじめに
ベイジアンネットワーク分析の代表的なライブラリに R の bnlearn がありますが、その描画部分の不明点を製作者の Marco Scutari 先生に教えてもらいました。とても有難かったのでシェアするために記事を書いています。
TL;DR
bnlearn で作成したネットワーク図のうち、一部分を描画したいときの tips です。大きなグラフのサブグラフでも可能ですし、他のノードと接続されていない孤立ノードの削除にも使えます。
結論としていいメソッドは存在しないので手動で行う必要があり、簡単な関数化してみました。
準備
(自分の備忘録のために残していますが、この記事を読むような人には不要なセクションです)
install.packages("bnlearn");
#devtools::install_github("vspinu/bnlearn") #こちらでもOK
install.packages("BiocManager");
BiocManager::install("Rgraphviz");
library(bnlearn);
library(Rgraphviz);
set.seed(0);
N <- 100;
x1 <- round(runif(N), 0);
x2 <- round(x1 + rnorm(N) * 0.2, 0);
x3 <- round(runif(N), 0);
x4 <- round(x1 / 2 + x3 / 2 + rnorm(N) * 0.2, 0);
x5 <- round(runif(N), 0);
dat <- data.frame(x1, x2, x3, x4, x5);
dat[dat > 1] <- 1;
dat[dat < 0] <- 0;
dat <- data.frame(lapply(dat, as.factor));
データは x1 -> x2、{x1, x3} -> x4 で x5 が独立しています。
今回は離散なので factor 型に変換。
解析
ここも本題ではないので必要に応じて読み飛ばしてください。
※@hrkz_szk 様の bnlearnを使ってベイジアンネットワーク分析をやってみた ほとんどそのままです。そちらの方が勉強になります。
g <- hc(dat, score = "bic");
graphviz.plot(g, shape = "ellipse");
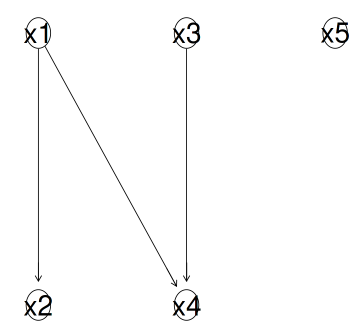
元の構造を正確に捉えています。
このデータなら必要ないのですが、より複雑なデータではブートストラップを行うと構造推定の精度が向上します。
str_g <- boot.strength(dat, R = 200, algorithm = "hc",
algorithm.args = list(score = "bic"));
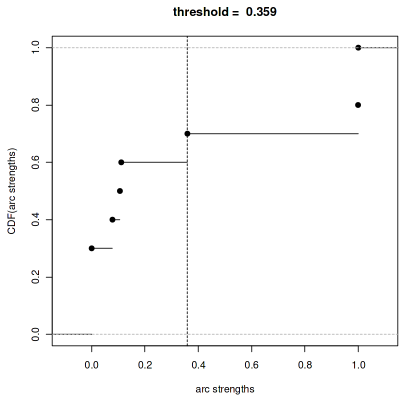
plot(str_g);
avg_g <- averaged.network(str_g);
strength.plot(avg_g, str_g, shape = "ellipse");

今回は変わらないですね。
ちなみに閾値を変えると別の結果になります
g_lower <- averaged.network(str_g);
strength.plot(g_lower, str_g, shape = "ellipse");

無関係なはずだった x3とx5 にエッジが引かれてしまっています。
本題:部分的なグラフ描画
少しだけ中身に触れます
strength.plot 関数のうち、第1引数は bn クラス、第2引数は bn.strength クラスです。このうち bn クラスは subgraph 関数を用いて操作できるのですが、bn.strength クラスにはいいメソッドがないのが問題でした。Marco Scutari 先生曰く
サブセットを作ろうとすると R が attribute を消してしまうのが問題です
ということです。解決策としては、手動で attribute を付け加えるという形になります。
コード
set_bn_attr <- function(bn_strength, subset_names){
subset_s <- bn_strength[(bn_strength$from %in% subset_names) &
(bn_strength$to %in% subset_names), ];
attr(subset_s, "threshold") <- attr(bn_strength, "threshold");
attr(subset_s, "method") <- attr(bn_strength, "method");
attr(subset_s, "nodes") <- subset_names;
attr(subset_s, "class") <- attr(bn_strength, "class");
return(subset_s)
}
ss <- set_bn_attr(str_g, c("x1", "x2", "x3", "x4"));
strength.plot(subgraph(avg_g, nnames), ss, shape = "ellipse");
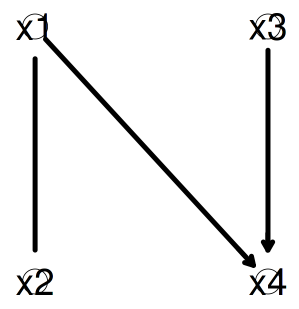
という訳であっさり解決しました。Marco Scutari 先生に重ねて感謝です。
なおネットワークの中身に関しては、bn オブジェクトを見るといろいろ分かりそうです。
> names(avg_g)
[1] "learning" "nodes" "arcs"
> avg_g$nodes
$x1
$x1$mb
[1] "x2" "x3" "x4"
$x1$nbr
[1] "x2" "x4"
$x1$parents
character(0)
$x1$children
[1] "x2" "x4"
$x2
$x2$mb
[1] "x1"
$x2$nbr
[1] "x1"
$x2$parents
[1] "x1"
$x2$children
character(0)
$x3
$x3$mb
[1] "x1" "x4"
$x3$nbr
[1] "x4"
$x3$parents
character(0)
$x3$children
[1] "x4"
$x4
$x4$mb
[1] "x1" "x3"
$x4$nbr
[1] "x1" "x3"
$x4$parents
[1] "x1" "x3"
$x4$children
character(0)
$x5
$x5$mb
character(0)
$x5$nbr
character(0)
$x5$parents
character(0)
$x5$children
character(0)