0. 事前に行ったこと
「AIの医療応用研究事例について一つ調べ、メリットと課題をまとめよ」というレポート課題をgoogle classroom上で課し、gdocで提出してもらったレポートを前回の記事に記載した方法で抽出した。
続いて、類似度評価のために、4種類のよく似たレポートを自作した。表現の改変(改悪)は、エキサイト日英翻訳→google英日再翻訳をした文章をベースに、提出されたレポートを参考にして読みづらい日本語表現アレンジを加えた。
**ダミーレポート1 原本 学籍番号:99999**
*・人工知能の医療応用事例 ゲノム情報と電子カルテ情報から腹部大動脈瘤を予測する研究結果が2018年にスタンフォード大学の研究グループから発表されている。 腹部大動脈瘤は頻度が最も高い大動脈瘤であり、動脈硬化などの原因によって通常直径約2cmの腹部大動脈が3cm以上に膨張した状態を指す。破裂するまで自覚症状がないこと、破裂した場合出血性ショックによって高確率で生命が奪われてしまうことから、超音波エコー検査による早期発見および迅速な外科的治療が必要である。 スタンフォード大学のJingjing Liらは、一塩基多型(Single nucleotide variants, SNVs)の情報と電子カルテ情報を組み合わせて腹部大動脈瘤の有無を予測する機械学習モデルHEALを構築した[1, 2]。Liらは、401名のデータ利用してロジスティック回帰を基本とするモデルを作成し、腹部大動脈瘤の有無を予測した。その結果、SNVs情報のみ利用したモデルの受信者動作特性(ROC)曲線のArea Under the Curveは0.690、電子カルテから抽出した年齢・性別・喫煙歴など14種類の情報を利用したモデルでは0.775に留まったが、両者を組み合わせたモデルは0.803となり、精度の向上が認められた。 ・メリット 腹部大動脈瘤の有無の予測にとどまらず、疾患の本態解明にも役立つ情報が得られる点が最大のメリットである。変数重要度の高いSNVsに着目したパスウェイ解析により、発症に寄与する分子機序の予測や新規創薬標的分子の提案が可能となる。 ・課題 SNVs情報を電子カルテ情報に上乗せした際の性能向上率は、全ゲノムシークエンスのコストに対して到底割に合っているとは言い難い。Liらは、SNVsに加えてゲノムへの塩基の挿入や欠失(Indel)、コピー数多型(CNVs)の情報を加味することで性能が向上する可能性について言及している[1]。また、疾患ゲノムAI研究に共通する新NP問題も課題である。新NP問題とは、解析サンプル数(Number, N)に対してSNVsなどのパラメタ数(P)が非常に多いために、従来の統計学では解析が困難になる問題である。予測性能向上のためには、新たな統計的推論方法の構築が必要である。参考文献 [1] Jingjing Li, et. al., Decoding the Genomics of Abdominal Aortic Aneurysm, Cell, 6;174(6):1361-1372.e10. (2018) [2] 第2回日本メディカルAI学会公認資格 メディカルAI専門コース講義テキスト p59-62*
**ダミーレポート1の表現改悪 学籍番号:88888**
*人工知能の医療応用例 2018年、スタンフォード大学の研究会から、ゲノム情報と患者の電子カルテ情報から腹部大動脈瘤を予測する研究結果が発表されました。 腹部大動脈瘤とは、頻度が最も高い大動脈瘤であり、通常、直径約2cmの腹部大動脈は、3cm以上腫れた状態を示します。発症原因の90%以上が動脈硬化であり、リスクファクターとして老化、喫煙、高血圧などがあります。自覚症状のない腹部大動脈瘤が破裂した場合、出血性ショックを起こして高率に命を奪われるということになるので、超音波エコー検査による早期発見と手術のような迅速な治療が必要になると思います。 この腹部大動脈瘤を、ゲノムの一塩基多型に関する情報と電子カルテ情報を組み合わせて予測するHEALという人工知能が開発されました。 このHEALというAIは、電子カルテやSNV情報だけをそれぞれ単独で使うのではなく、両方を使うことによって予測の精度を高めることができます。 メリット 腹部大動脈瘤の存在の予測に加えて、疾患の実態解明にも役立つ情報が得られるということもメリットです。遺伝子解析を詳細に行うことで、腹部大動脈瘤のリスクを高める生活習慣と遺伝子変異を解明できたり、新しい革新的な医薬品の開発が期待されます。 課題 HEALの予測精度は決して高いわけではなく誤診をしてしまう可能性があり、超音波検査は簡単な検査なのでAI導入の必要がないことが課題です。また、プログラムのバグのせいで誤診が起きたときに誰が責任を取るのか、という問題もあります。***ダミーレポート2(1の短縮ver) 学籍番号:99998**
*ゲノム情報と電子カルテ情報から腹部大動脈瘤を予測する研究結果が2018年に発表されている。スタンフォード大学のJingjing Liらは、一塩基多型(Single nucleotide variants, SNVs)情報と電子カルテから抽出した年齢・性別・喫煙歴など14種類の情報から腹部大動脈瘤の有無を予測する機械学習モデルHEALを構築した[1, 2]。Liらは、受信者動作特性(ROC)曲線のArea Under the Curve (AUC)を用いて複数の学習条件におけるモデル性能を評価した。その結果、SNVs情報のみを学習させたモデルのAUCは0.690、電子カルテ情報のみを学習させたモデルのAUCは0.775に留まったが、両者を組み合わせたモデルのAUCは0.803となり、精度の向上が認められた。 HEALの最大のメリットは、疾患の有無が予測可能なだけでなく、腹部大動脈瘤の本態解明にも役立つ点である。モデル中で重要度の高いSNVsに着目したパスウェイ解析を行うことで、発症に寄与する分子機序の予測や新規創薬標的分子の提案が可能となる。 一方で、予測器としてのHEALは様々な課題を抱えている。最大の課題は、ゲノム配列検査のコストに対して予測性能の向上率が割に合っていない点である。Liらの報告では、ゲノムへの塩基の挿入や欠失(Indel)、コピー数多型(CNVs)の情報とSNVsを組み合わせることで改善される可能性が考察されている[1]。また、疾患ゲノムAI研究に共通する新NP問題も課題である。新NP問題とは、サンプル数(Number, N)に対してパラメタ数(P)が非常に多いために従来の統計学では解析が困難になる問題であり、新たな解析手法の登場が待ち望まれている。腹部大動脈瘤が簡便かつ非侵襲的な超音波検査でスクリーニング可能な疾患であることも合わせると、予測のためにHEALを臨床現場に導入する意義は乏しいといえる。参考文献 [1] Jingjing Li, et. al., Decoding the Genomics of Abdominal Aortic Aneurysm, Cell, 6;174(6):1361-1372.e10. (2018) [2] 第2回日本メディカルAI学会公認資格 メディカルAI専門コース講義テキスト, 日本メディカルAI学会, p59-62*
**ダミーレポート2の表現改悪 学籍番号:88887**
* ゲノム情報と電子患者の症例記録情報から腹部大動脈瘤を予測する研究結果が2018年に発表されました。この研究結果はスタンフォード大学から発表されたものであり、電子カルテ情報とSNVs情報から、腹部大動脈瘤の存在を予測します。 このモデルはHEALと言われ、SNVs情報のみを学習するとAUC 0.690、電子カルテ情報のみを学習すると0.775という予測性能でしたが、両方を組み合わせたモデルは、0.803となり、精度が向上します。 HEALの最大のメリットとして、疾患の実態解明にも役立つ情報が得られ、遺伝子解析を詳細に行うことで、腹部大動脈瘤のリスクを高める生活習慣と遺伝子変異を解明できたり、新しい革新的な医薬品の開発が期待されます。 一方、HEALにはさまざまな課題があります。一番問題なのは、予測精度は決して高いわけではなく誤診をしてしまう可能性があることです。SNVsに加えて、IndelやCNVsなどの変異情報を組み合わせると、精度が改善できると思われます。新NP問題も解決すべき課題だと考えられます。 また、超音波検査は簡単な検査なのでわざわざHEALを導入する必要がないことも課題です。また、プログラムのバグのせいで誤診が起きたときに誰が責任を取るのか、という問題もあります。参考文献 https://www.sciencedirect.com/science/article/pii/S0092867418309164?via%3Dihub*
1. 目的
提出レポートの類似性評価および簡単な内容のグルーピングを(なるべく学習コストの少ない方法で)実施する。
具体的には、以下の順に実施した。
a. Google colab上でDoc2Vecを利用して、レポートをベクトル化する。
b. 各レポートのコサイン類似度を評価する。
c. ベクトルの次元削減→クラスタリングを行い、ワードクラウドで内容の可視化を行う。
なお、類似性評価のコントロール指標して、上述のダミーレポートを複数用意し、学生から回収した回答と混ぜて解析を行った。
本記事の目的は、自然言語処理の素人が簡単に提出レポートの評価を行う方法の記録を公開し、共有することです。
解析手法の原理解説は行いませんので、必要に応じて別途文献をあたって下さい。
対象読者:提出レポートの類似性評価をサクッと行いたい教員・評価者
非対象者:自然言語処理の原理手法の解説を求めている人
1.5 【結果概略】
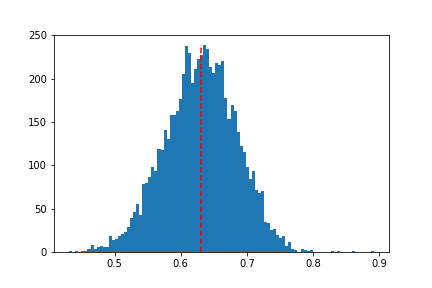
まともな表現のダミーレポート2種(擬似学籍番号99999と99998)のコサイン類似度は0.891と一番高い結果となった。次いで類似度が高かったのは表現を悪化させたダミーレポート2種(88888と88887)で、コサイン類似度は0.863となった。
一方で、日本語表現力に差があるが文章量や内容を類似させたレポートのペアの類似度は0.655(99999と88888のペア)、および0.745(99998と88887)となった。
この結果は、日本語wikipedia学習済みdoc2vecを利用したレポート類似度評価モデルが、文章内容よりもレポートの体裁(日本語表現や数値結果記載の有無、参考文献の書き方など)により敏感なことを示唆している。
参考文献の情報まで評価文章として扱った方が良いかは、モデルをどのような目的で利用するかによるだろう。
2. ライブラリインストール・インポート
google colabに入っていないライブラリをインストールする。
・自然言語処理用ライブラリgensimのインストール
!pip install gensim
・形態素解析用のライブラリmecabのインストール
インストール方法は↓を参考にした。
ColaboratoryでMeCabを使えようにする。
!apt install aptitude
!aptitude install mecab libmecab-dev mecab-ipadic-utf8 git make curl xz-utils file -y
!pip install mecab
UMAP後のクラスタリングに使うhdbscanのインストール
!pip install hdbscan
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
from sklearn.metrics.pairwise import cosine_similarity
import re
import pickle
import string
import unicodedata
%matplotlib inline
3. 学習済み公開モデルのロード
日本語wikipediaで学習したdoc2vecモデル(CC-BY-SA)が公開されているので、こちらを利用させて頂いた。
今回は、dmpv300dモデルを利用。9GB近いので、ロードに数分を要する。
from gensim.models.doc2vec import Doc2Vec
model = Doc2Vec.load("/モデルが入ったディレクトリ/jawiki.doc2vec.dmpv300d.model")
例えば、「生理学」と類似するwikipediaのドキュメントを表示すると、
model.docvecs.most_similar("生理学", topn=5)
---------------------------------------------------------------------------
/usr/local/lib/python3.6/dist-packages/gensim/matutils.py:737: FutureWarning: Conversion of the second argument of issubdtype from `int` to `np.signedinteger` is deprecated. In future, it will be treated as `np.int64 == np.dtype(int).type`.
if np.issubdtype(vec.dtype, np.int):
[('病態生理学', 0.6333913803100586),
('口腔生理学', 0.6323986649513245),
('体細胞', 0.6239209175109863),
('病態生化学', 0.6197534799575806),
('血管内皮', 0.6145813465118408)]
とこのように似ている分野の記事が抽出される。
4. レポート文章のインポート
前回の記事 に記載した方法で、gdocファイルで提出されたレポートから学籍番号と本文を抽出したテキストファイルを読み込む。
DIR = "/テキスト抽出ファイルのあるディレクトリ/"
dat = pd.read_csv(DIR + "Report_from_gdoc.txt", encoding = "utf-8", sep='\t', index_col=0) #タブ区切りで出力したもの
google classroomには学生が途中で保存した中身の無いファイルなどが含まれている場合がある。
後の解析のために、Nanが入っている行を削除する。
if type(dat.index) != "str":
dat.index = dat.index.fillna(-1)
dat = dat.dropna(how='any', axis=0)
dat.index = dat.index.astype(int).astype(str)
# 学籍番号が5桁の行のみ抽出
dat2 = dat.filter(regex='^\d{5}$', axis=0)
dat2.index = dat2.index.astype(int)
dat2 = dat2.dropna(axis = 0, how = 'any')
# ソート
dat2 = dat2.sort_index()
# 中身確認
dat2.tail(4)
| 学籍番号 | 本文 |
|---|---|
| 88887 | ゲノム情報と電子患者の症例記録情報から腹部大動脈瘤を予測する研究結果が2018年に発表されま... |
| 88888 | 人工知能の医療応用例\n 2018年、スタンフォード大学の研究会から、ゲノム情報と患者の電子... |
| 99998 | ゲノム情報と電子カルテ情報から腹部大動脈瘤を予測する研究結果が2018年に発表されている。ス... |
| 99999 | ・人工知能の医療応用事例\n ゲノム情報と電子カルテ情報から腹部大動脈瘤を予測する研究結果が... |
5. 分かち書き・前処理
model.infer_vector()で文章を300次元ベクトルに変換するのだが、このメソッドには「分かち書き済みの単語リスト」を入力する必要がある。
そのため、はじめにMeCabを用いた分かち書き用の関数tokenize()を定義する(wikipediaモデルの使い方より)。
import MeCab
def tokenize(text):
wakati = MeCab.Tagger("-O wakati")
wakati.parse("")
return wakati.parse(text).strip().split()
続いて、文章の前処理を行う関数を定義する。
なお中身はこちらの記事の内容を殆ど改変せずに利用している。
def preprocess(text):
### 改行コードなど空白で置換
#new=' '.join(text.splitlines())
new=' '.join(text.split("\\n"))
### 全角記号を一度半角に変えてから削除
new=unicodedata.normalize("NFKC", new).translate(str.maketrans("", "", string.punctuation + "「」、。・"))
return new
最後に、分かち書き後の処理を行う関数を定義する。
同じくこちらの記事の内容をほぼ丸ごと利用している。
def delete_words(words):
# ひらがな1文字削除
newWords = [w for w in words if re.compile('[\u3041-\u309F]').fullmatch(w) == None]
# カタカナ1文字削除
newWords = [w for w in newWords if re.compile('[\u30A1-\u30FF]').fullmatch(w) == None]
# 数字1文字を削除
newWords = [w for w in newWords if re.compile('[0-9]{1}').fullmatch(w) == None]
# 特定の単語削除
delwords=["年","月","日", "AI", "人工知能", "人工", "知能",
# "メリット", "デメリット", "課題", "利点", "欠点", "応用", "研究", "事例",
# 課題に関連した頻出ワードを削除 出題文の分かち書きを指定しても良いかもしれない。
"文献", "参考", "参考文献", "引用", "引用文献"]
newWords = [w for w in newWords if not(w in delwords)]
return newWords
以上の工程をまとめて、infer_vectorに放り込むデータを作成する関数
(前処理ー分かち書きー後処理)は、
def generate_dataset(text):
preprocess_text = preprocess(text)
split_words = tokenize(preprocess_text)
return delete_words(split_words)
となる。自前で用意したレポート(学籍番号99998)から作成された分かち書きデータを確認すると、
['ゲノム', '情報', '電子', 'カルテ', '情報', 'から', '腹部', '大動脈', '瘤', '予測']
の様に品詞分解されている。
6. 全レポートのベクトル抽出
generate_datasetを使って、
Vecall: 全レポートのベクトル化情報、np.array
Wordall: 全レポートの分かち書き情報、dict
を作成する。
Vecall = np.zeros(shape=[len(dat2.index), 300])
Wordall = dict()
for i in range(len(dat2.index)):
tmp_sentence = dat2.iloc[i].values.tolist()[0]
tmp_doc = generate_dataset(tmp_sentence)
tmp_vec = model.infer_vector(tmp_doc)
Wordall[int(dat2.iloc[i].name)] = tmp_doc
Vecall[i,:] = tmp_vec
計算結果保存
Vecall_df = pd.DataFrame(Vecall)
Vecall_df.index = dat2.index
Vecall_df.to_csv(DIR + "Doc2Vec.csv")
with open(DIR + 'Wordall.pkl', 'wb') as handle:
pickle.dump(Wordall, handle)
7. コサイン類似度を用いた全レポートの類似度計算
mat = np.round(cosine_similarity(Vecall),4)
mat_df = pd.DataFrame(mat)
mat_df.index = dat2.index
mat_df.columns = dat2.index
mat_df.to_csv(DIR + "cosine_similarity.csv")
fig, ax = plt.subplots(figsize=(20,20)) # Sample figsize in inches
sns.heatmap(mat_df, square=True, annot=False, cmap="cividis", ax=ax)
plt.title('cosine distance')
plt.savefig(DIR + 'heatmap.pdf')
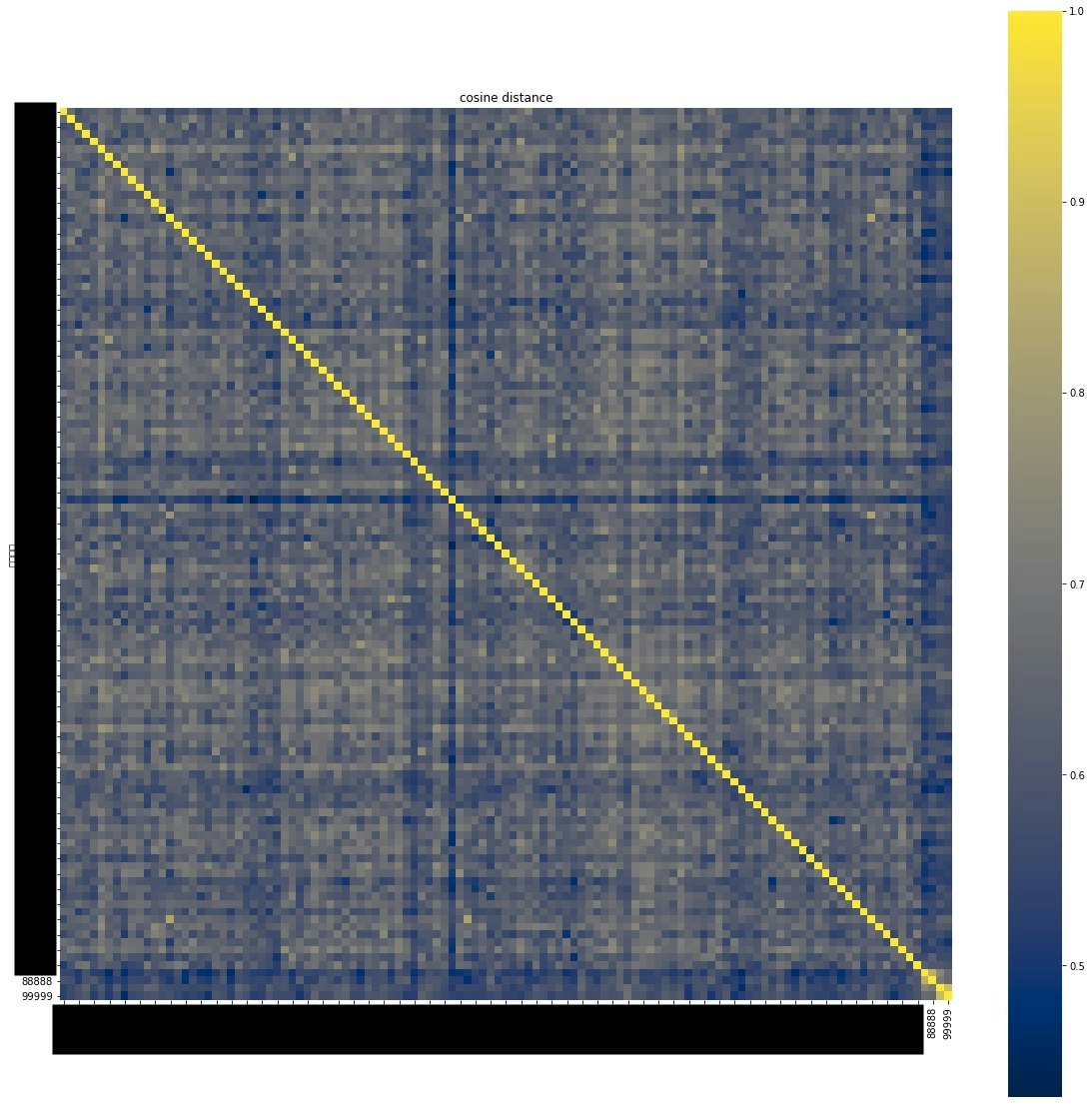
このヒートマップでは、縦横に学籍番号が並んでおり、1マスが該当のレポートのペアの類似度を表している。
対角成分は同一レポートの比較になるので、類似度は1となる。
実際にヒートマップで目立つ点のペアを見比べてみると、学会発表要旨のほぼコピペや、誤字まで同じ文章を含むレポートを検出することが出来た。今回の場合、0.8を越えるペアは類似した内容となっていた。
その他結果の概略は、記事上部を参考のこと。
**類似度が高い上位top n位のペアを出力**
# 下三角成分のみ取り出す
mat_de = mat.copy()
for i in range(mat_de.shape[0]):
for j in range(i, mat_de.shape[0]):
mat_de[i, j] = 0
n=10
flat_indices = np.argpartition(mat_de.ravel(),-n)[-n:]
row_indices, col_indices = np.unravel_index(flat_indices, mat_de.shape)
cols = ["Student1", "Student2", "Similarity", "Docs(Student1)", "Docs(Student2)"]
similar_topn = pd.DataFrame(index=[], columns=cols)
for i in range(len(row_indices)):
stu1 = dat2.index[[row_indices[i]]][0]
stu2 = dat2.index[[col_indices[i]]][0]
sim = mat_de[row_indices[i], col_indices[i]]
doc1 = dat2.iloc[row_indices[i]].values.tolist()[0]
doc2 = dat2.iloc[col_indices[i]].values.tolist()[0]
record = pd.Series([stu1, stu2, sim, doc1, doc2], index=similar_topn.columns)
similar_topn = similar_topn.append(record, ignore_index=True)
similar_topn = similar_topn.sort_values("Similarity", ascending = False)
similar_topn.to_excel(DIR + "top_" + str(n) + "similar.xlsx")
**全レポートの類似度平均およびヒストグラム**
8. UMAPによる次元削減、分類
使用ライブラリの呼び出し
# Dimension reduction and clustering libraries
import umap
import hdbscan
import sklearn.cluster as cluster
from sklearn.metrics import adjusted_rand_score, adjusted_mutual_info_score
次元削減・クラスタリング
# クラスタリングのハイパーパラメタは微調整が必要
clusterable_embedding = umap.UMAP(
n_neighbors=15,
spread = 1,
min_dist=0.0,
n_components=2,
random_state=42,
metric = 'cosine',
).fit_transform(Vecall_df.drop('学籍番号', axis=1))
labels = hdbscan.HDBSCAN(
#min_samples=3,
gen_min_span_tree=True,
min_cluster_size=3,
).fit_predict(clusterable_embedding)
作図
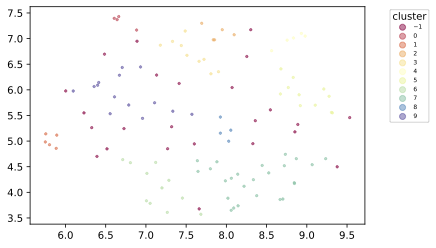
scatter = plt.scatter(clusterable_embedding[:, 0], clusterable_embedding[:, 1],
c=labels, s=5, cmap='Spectral', marker="o", alpha = 0.5)
lg = plt.legend(*scatter.legend_elements(),
loc="upper right", title="cluster", prop={'size': 10}, bbox_to_anchor=(1.2, 1))
plt.savefig(DIR + "UMAP_cluster.pdf", bbox_extra_artists=(lg,), bbox_inches='tight')
Vecall_df_cluster.to_csv(DIR + "Doc2Vec_cluster.csv")
9. クラスターごとにワードクラウドを作る
以下のブログ・記事を参考に、クラスタごとの特徴をワードクラウドで可視化した。
Pythonでワードクラウド作り②日本語版
【備忘録】日本語のワードクラウドを作る
google colabでフォントを入れる方法
せっかく文章をベクトル化したのに、可視化にはベクトル化前の情報しか使っていない点が個人的にもやっとしている。改善の余地があるか。
使用ライブラリのインポート
from wordcloud import WordCloud
ワードクラウド作成。
google colabで動かすには、上記の記事を参考に日本語フォントを別途読み込ませる必要がある。
for x in np.unique(Vecall_df_cluster["Cluster"]):
cond = Vecall_df_cluster["Cluster"] == x
Num = Vecall_df_cluster["学籍番号"][cond]
WD = []
for i in list(Num):
WD.extend(Wordall[i])
#分割テキストからwordcloudを生成
delwordsWC = ["ある", "する", "いる", "こと", "応用", "研究", "事例", "医師", "医療", "医者", "患者"] #word cloudに邪魔な単語を削除。お好みで
WD = [w for w in WD if not(w in delwordsWC)]
splitted = ' '.join(WD)
wordc = WordCloud(background_color='white', font_path= "/日本語フォントのディレクトリ/NotoSansCJKjp-DemiLight.otf",
width=800, height=600).generate(splitted)
#画像ファイルとして保存
wordc.to_file(DIR + 'wordcloud_Clu' + str(x) + '.png')
自分が作ったダミーレポート群は同じクラスタに入り、内容の特徴が可視化できた。
10. 考察
提出レポートを追学習することによって、分類精度が向上できるかもしれない。
次年度以降同様の課題を出せば、過去の提出レポートとの類似度計算も行うことが出来るのも良い。
もう少し使用感を見てから関数化する。
レポートや記述問題採点の労力削減に自然言語処理がどれだけ役立つのか検討していきたい。