ggplot2③
さて、ggplot2 の第3回です。今回は度数分布の表現が主なテーマです。ヒストグラム、棒グラフ、箱ひげ図、バイオリンプロットを説明します。各種のグラフを描きつつ、Rの組み込み(built-in)データもどんどん扱っていきたいと思います。フィルタリングについても触れます。今回もDr.Greg Martinの動画で勉強したことが中心です。
ヒストグラムと棒グラフ
ヒストグラムと棒グラフ、見た目は似ています。ヒストグラムはビンとビンの間にすきまがありませんが、棒グラフは棒どうしがお互い離れています。見た目の違いは以上ですが、後ほどもう少し突っ込んで違いを述べます。
ヒストグラム
まず、データセットmsleep を使ってヒストグラムを描こうと思います。msleep はRの組み込みのデータセットなので、ダウンロードすることなく利用することができます。msleep とはdata()の説明によると"An updated and expanded version of the mammals sleep dataset"、つまり哺乳類の睡眠に関するデータです。
以下のような列があります。
> head(msleep)
# A tibble: 6 × 11
name genus vore order conservation sleep_total sleep_rem sleep_cycle awake brainwt bodywt
<chr> <chr> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Cheetah Acin… carni Carn… lc 12.1 NA NA 11.9 NA 50
2 Owl monkey Aotus omni Prim… <NA> 17 1.8 NA 7 0.0155 0.48
3 Mountain beaver Aplo… herbi Rode… nt 14.4 2.4 NA 9.6 NA 1.35
4 Greater short-tailed shrew Blar… omni Sori… lc 14.9 2.3 0.133 9.1 0.00029 0.019
5 Cow Bos herbi Arti… domesticated 4 0.7 0.667 20 0.423 600
6 Three-toed sloth Brad… herbi Pilo… <NA> 14.4 2.2 0.767 9.6 NA 3.85
欠損データ(NAの部分)が多いのが気になります。
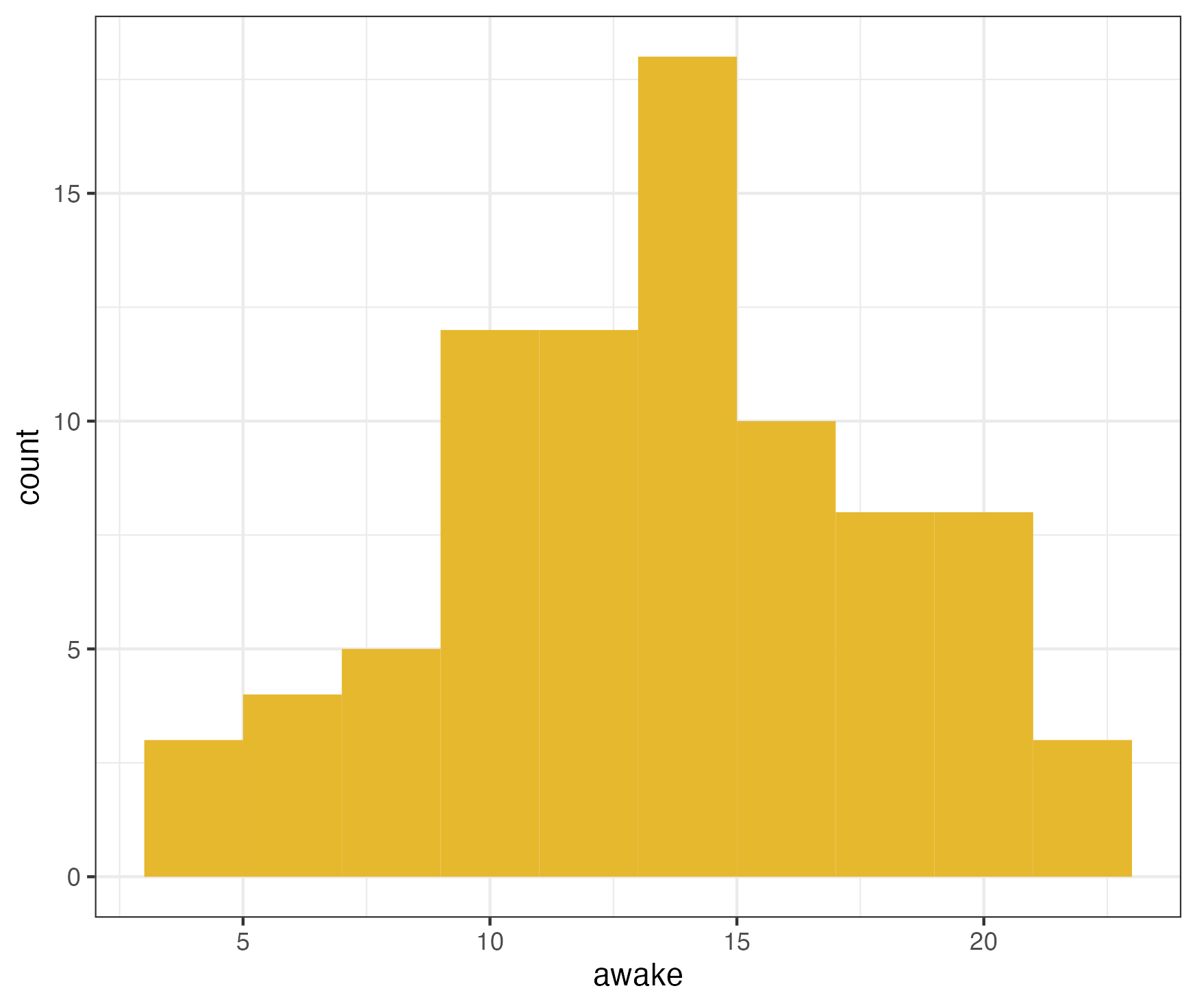
このうち、awake の度数分布をヒストグラムで描きます。awake は起きている時間を表すnumerical変数です。
msleep %>%
ggplot(aes(awake)) +
geom_histogram(binwidth = 2, fill="#e6b82e") +
theme_bw()
棒グラフ
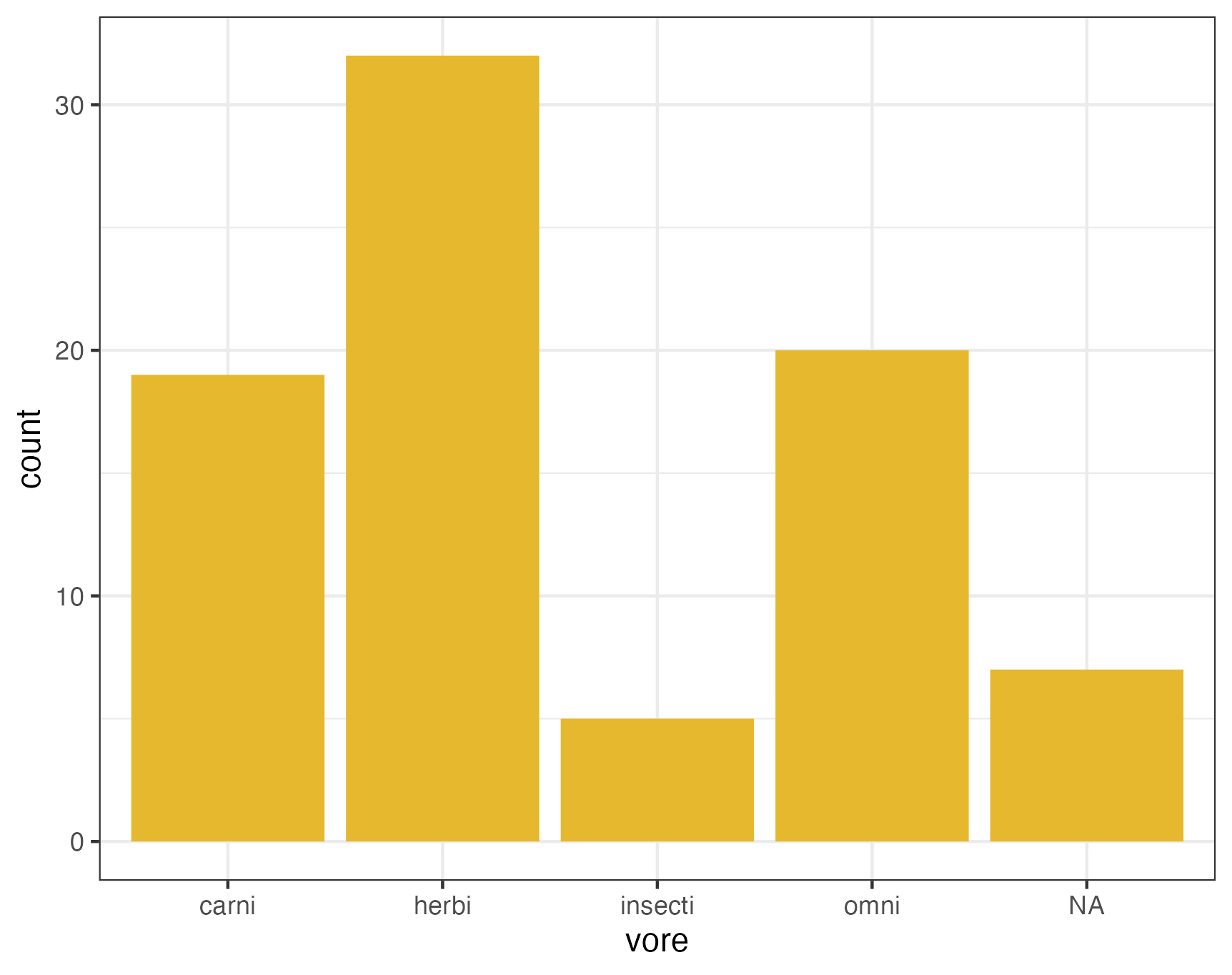
awake はnumerical 変数(つまり数値)でしたが、次はcategorical 変数(種類を表す変数)であるvore を描画します。ちなみにvore とは草食(herbi)、雑食(omni)、肉食(carni)、insecti(昆虫食)のいずれかを表すcategorical 変数です。
msleep %>%
ggplot(aes(vore)) +
geom_bar(fill="#e6b82e") +
theme_bw()
ヒストグラムと棒グラフの違い
ヒストグラムと棒グラフの違いですが、決定的に異なるのは説明変数の性質です。説明変数がnumerical のときはヒストグラム、categorical のときは棒グラフを描くことになります。awake は連続的な変量(順序や距離の概念が存在する)なので、グラフもビンとビンが隣接している、一方でvore は互いに無関係な4つ(順序や距離の概念が存在しない。NAを含めれば5つに見えますが、このことは次項で説明します)のカテゴリの度数を示すので、棒と棒は離れると理解しておくとよいと思います。
順序や距離の概念が存在しない、の意味はお分かりでしょうか。例えば上記の棒グラフでは左から"carni"、"herbi"...の順に並んでいますが、"herbi"、"carni"...と入れ替えてもグラフとしては成立するわけです。もちろん筆者に何らかの意図がある場合には入れ替わってもらったら困ることもあるでしょうが、グラフとしておかしいということはありません。一方ヒストグラムでは「10が5より左にあるけど15は5より右にある」なんていう状態は理解不能ですよね。
フィルタリング
さて、上記のヒストグラムにはご覧の通り、カテゴリの1つに"NA"(欠損値)が入ってしまっています。欠損値とは文字通りデータが欠落している箇所であり、本当は"carni"かも知れないし"herbi"かも知れないけど「データがないから判断できない」という意味です。それを1つのカテゴリとして扱うのはちょっと違いますね。
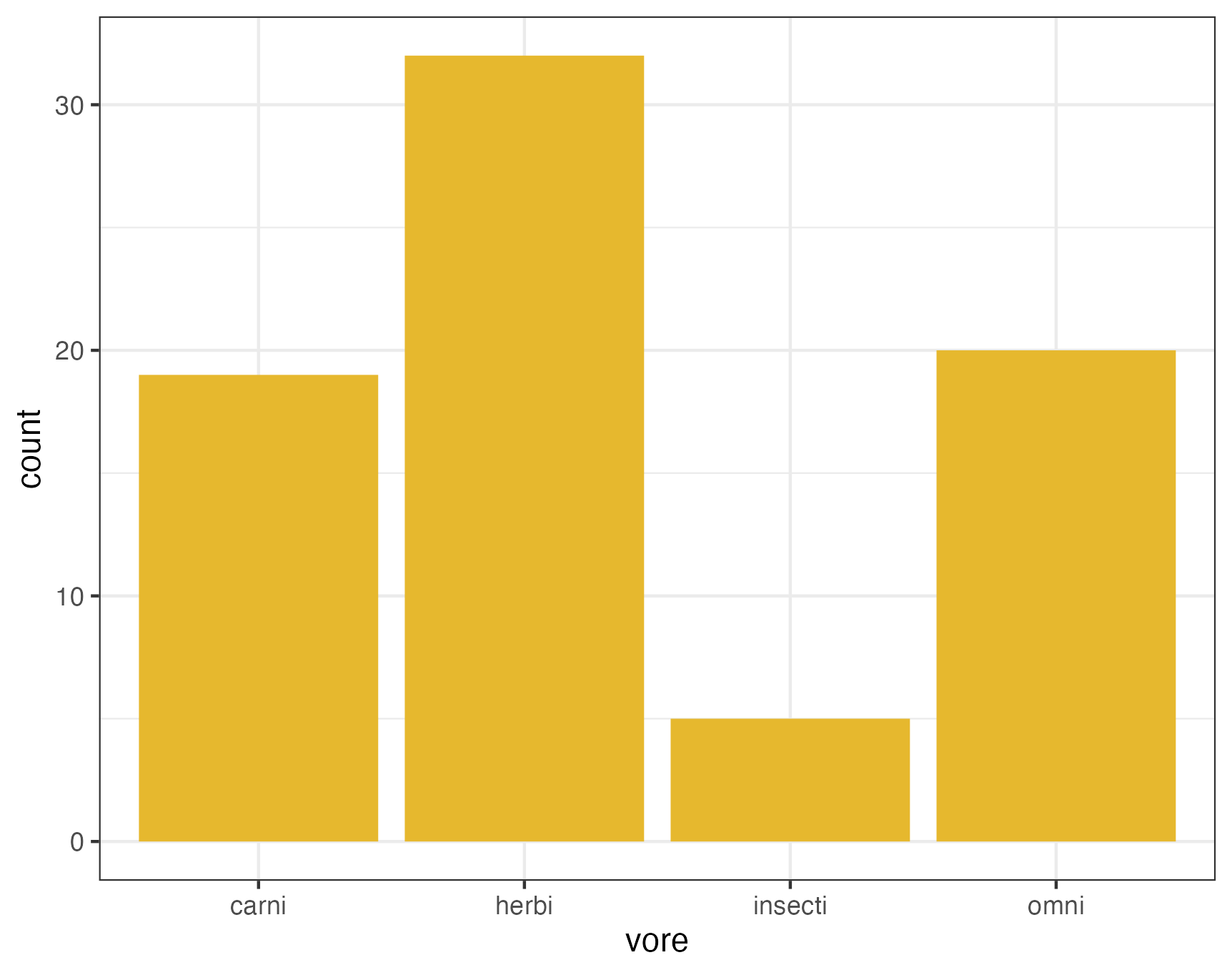
そこで、欠損値をどう扱えばいいか、といことですが、ここでは単純に欠損値を無視して描画することにします。データから"NA"を無視するにはdrop_na()という関数をフィルタとして使います。
# example A
msleep %>%
drop_na(vore) %>% # 欠損値を無視するフィルタ
ggplot(aes(vore)) +
geom_bar(fill="#e6b82e") +
theme_bw()
"NA"という"カテゴリ"(本来はカテゴリとして扱ってはいけない)がなくなりました!
drop_na(vore) というフィルタによってvoreが"NA"である行をすべて削除した上でggplot()に代入しています。
蛇足ですが、別の観点でもう一言述べておきます。これまではmsleep というdata.frame をそのまま代入するだけだったので、パイプオペレータ"%>%"を使う利点がよくわかりませんでした。しかし、この例ではmsleep からvore=NA である行をすべて取り除くという少し複雑な処理を行いましたので、もしパイプオペレータを用いないとすると、以下のようになります。
# example B
ggplot(drop_na(msleep, vore), aes(vore)) +
geom_bar(fill="#e6b82e") +
theme_bw()
いかがですか?だんだん読みにくくなってきたのではないでしょうか。 example Bに比べ、example Aは「まずmsleepを使います。そこからvore=NA なる行をすべて削除します。その出力をggplot()に代入します。」という流れが明確で可読性がグッと上がっています。
箱ひげ図とバイオリンプロット
箱ひげ図
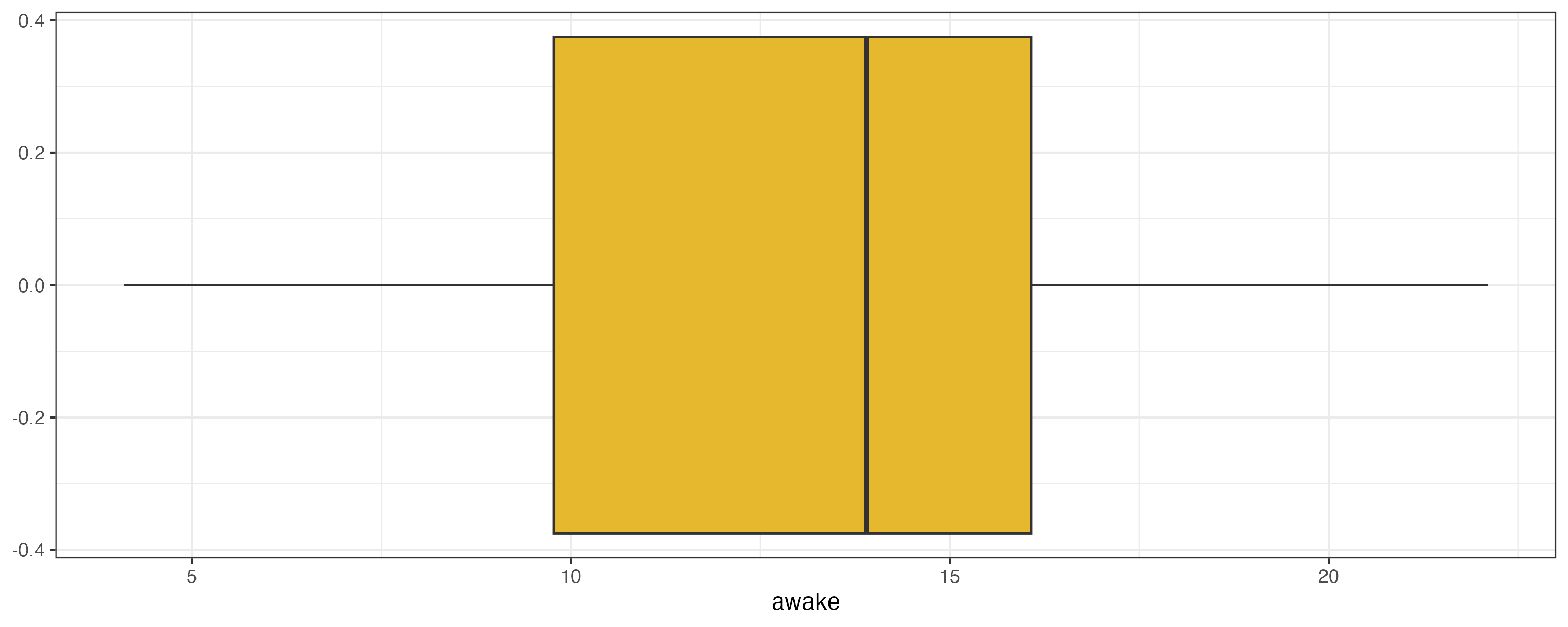
さて、今度はawakeの度数分布の可視化を、ちょっと視点を変えて、箱ひげ図を描いて考察してみます。
msleep %>%
drop_na(vore) %>%
ggplot(aes(awake)) +
geom_boxplot()
theme_bw()
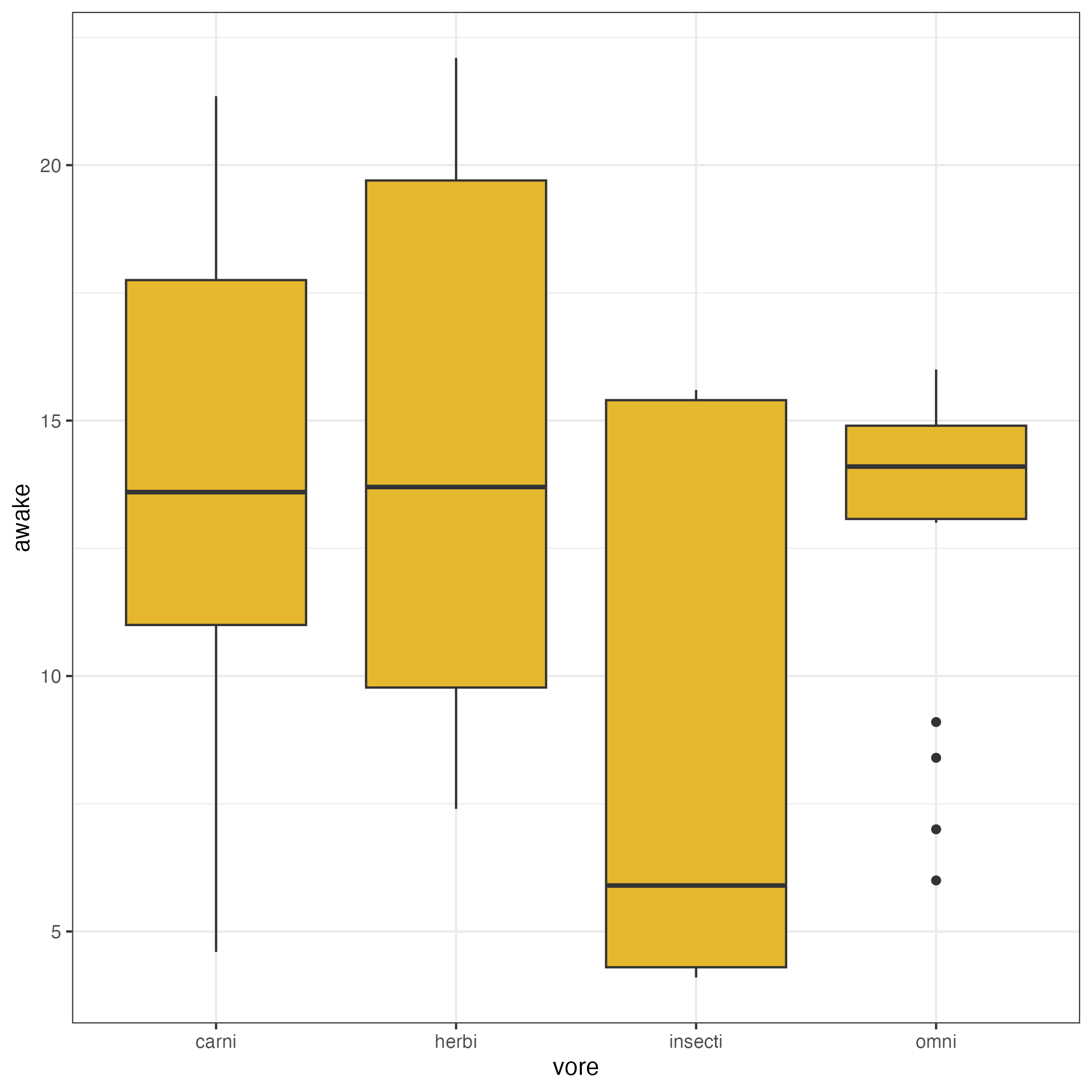
中央値が14付近、第1四分位数が10くらい、第3四分位数が16くらいであることは読み取れます。今度はvoreのカテゴリ別に箱ひげ図を描いてみます。具体的にはggplot() の引数をaes(awake) からaes(vore, awake) に書き換えればOKです。
msleep %>%
drop_na(vore) %>%
ggplot(aes(vore, awake)) +
geom_boxplot() +
theme_bw()
昆虫食(insecti)の動物は顕著に起きている時間が短いようです。
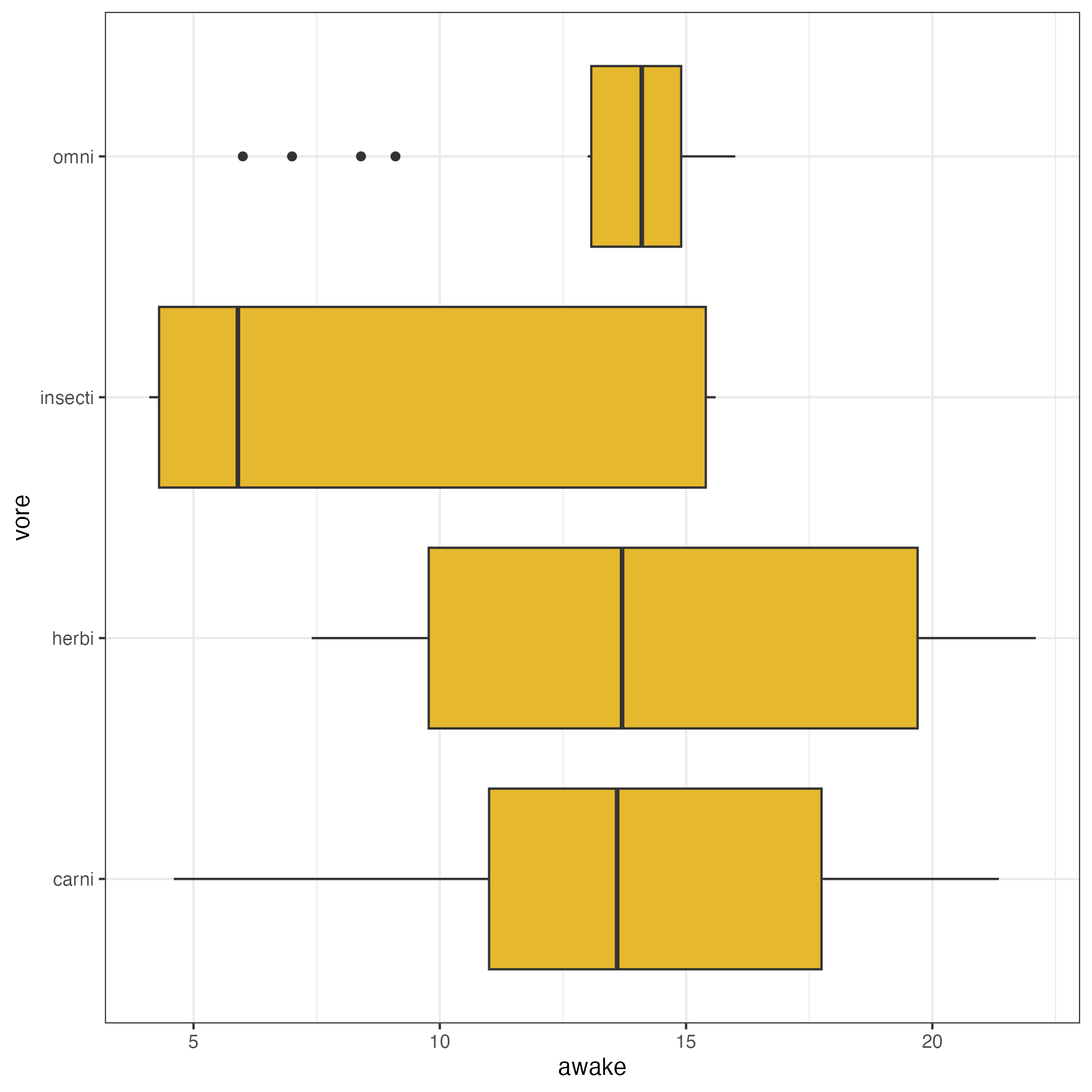
この形状でもよいですが、横向きにした方が可読性が上がるかも知れません。
msleep %>%
drop_na(vore) %>%
ggplot(aes(vore, awake)) +
geom_boxplot() +
coord_flip() # 縦横を入れ替える
theme_bw()
ここで追加したcoord_flip() レイヤは縦横を入れ替える、という役割をはたします。
バイオリンプロット
awake の分布を可視化するにはバイオリンプロットという方法もあります。
msleep %>%
drop_na(vore) %>%
ggplot(aes(vore, awake)) +
geom_violin(fill="#e6b82e") +
coord_flip() +
theme_bw()
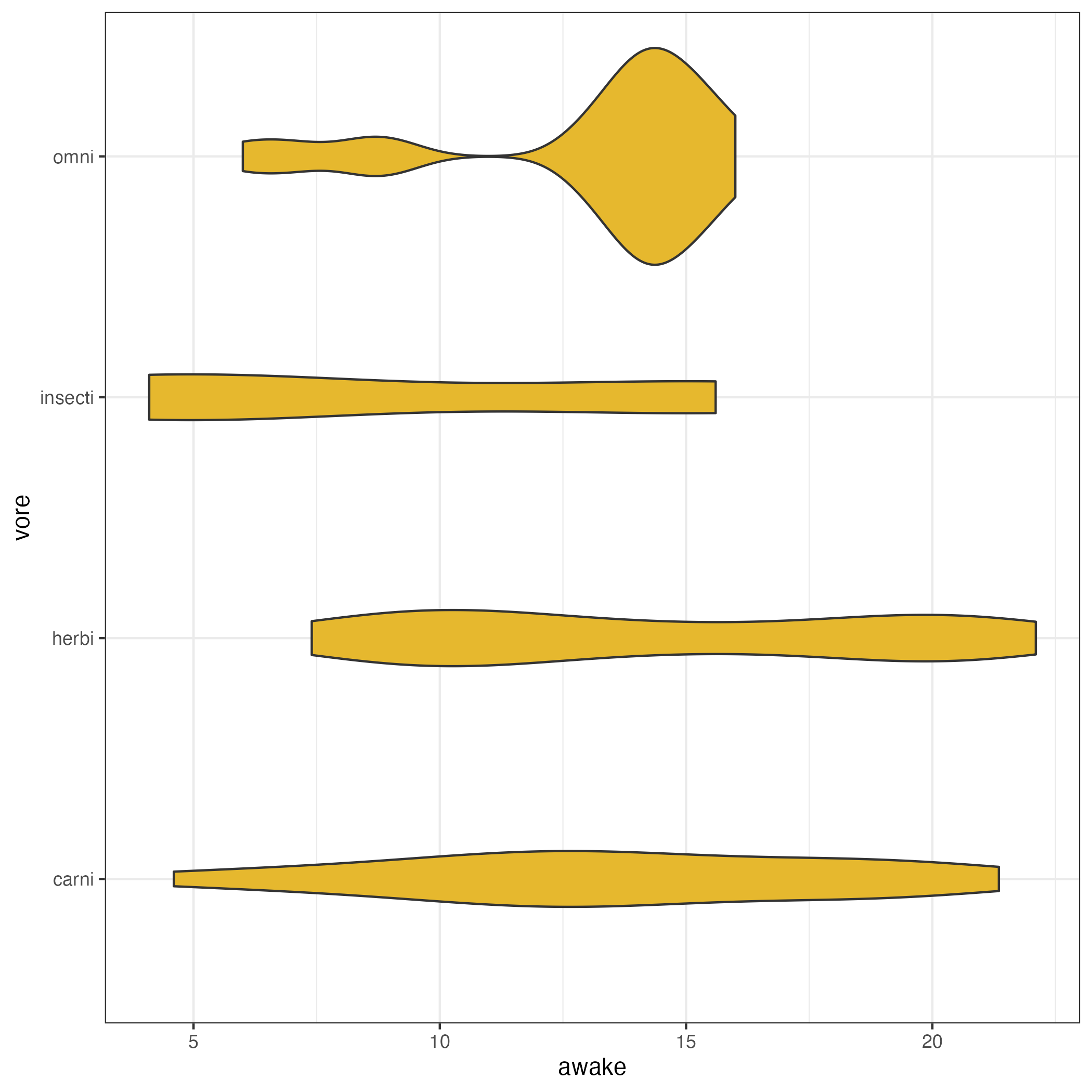
これを見ると、その他3カテゴリは最小値から最大値まで満遍なく分布しているのに対し、omni だけは14時間付近に分厚く分布している様子がよくわかります。同じ度数分布の描画でもグラフの種類を変えれば、違った特徴が見えてきます。
facetting
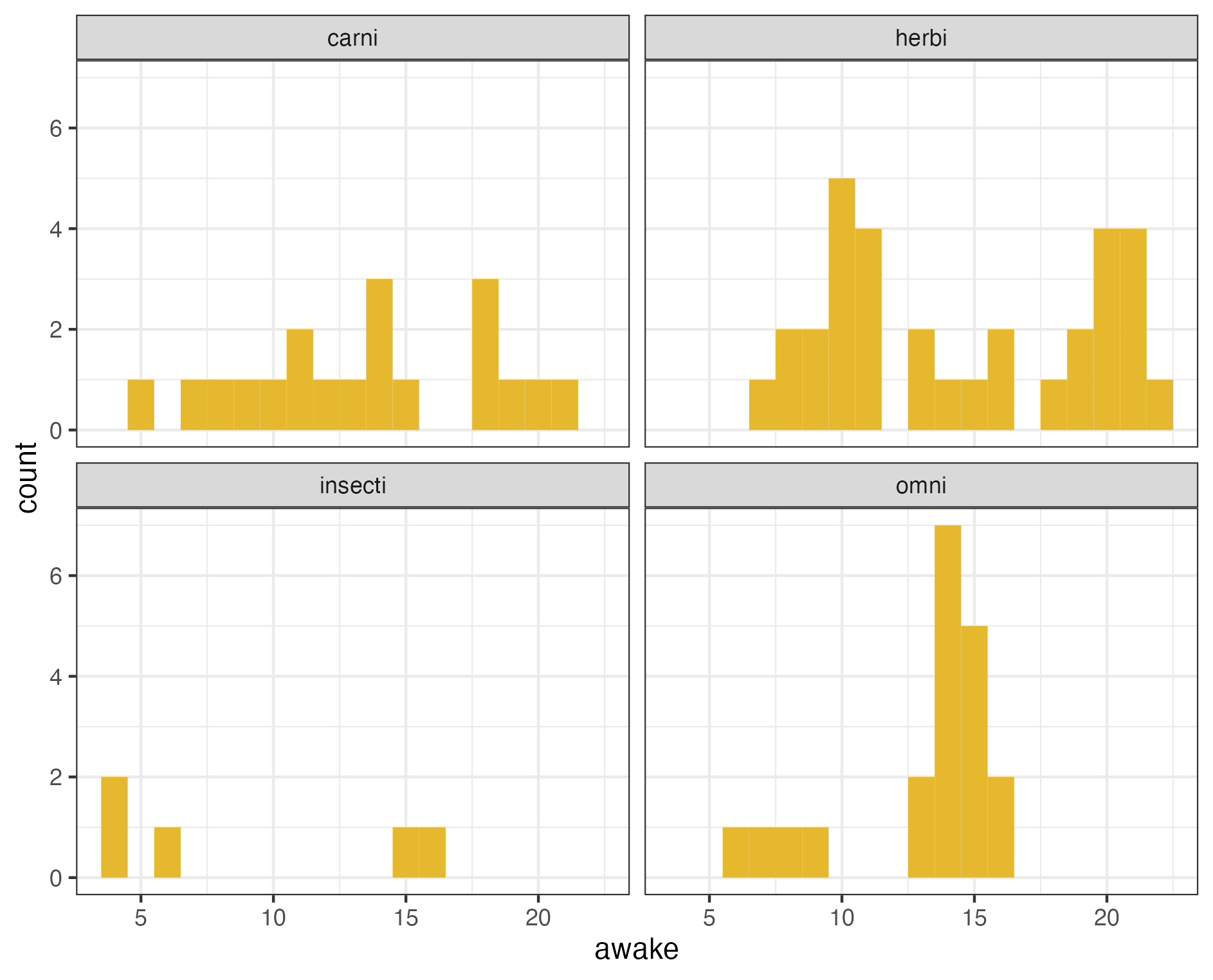
omni のこの特徴を可視化する他の方法はないでしょうか。
ここではhistogram + facetting で解決を試みたいと思います。facettingとは多面表示、つまりvore の各カテゴリごとに分けた上で別々にヒストグラムを描画するということです。
msleep %>%
drop_na(vore) %>%
ggplot(aes(awake)) +
geom_histogram(binwidth=1, fill="#e6b82e") +
facet_wrap(~vore) +
theme_bw()
「omni だけは14時間付近に分厚く分布している」という特徴についてはバイオリンプロットがより明確に示しています。しかし、facetting を用いたグラフではまた新たな視点が生まれるかも知れませんので、どちらが優れているということではありません。各グラフにはそれぞれ得手不得手がありますので、筆者が主張したいことに応じて適切なものを選択することが大事になってきます。
参考文献
Data visualisation using ggplot with R Programming : Dr.Greg Martin の動画
Cheatsheet : (公式)チートシートです。
An updated and expanded version of the mammals sleep dataset
トップページはこちら