Python MNEはオープンソースの脳磁図(MEG),脳波(EEG)の解析や可視化のツールです.多くのデバイスのデータフォーマットに適用できるため,汎用性が高いと言えるでしょう.この記事では,最もベーシックなチュートリアルに沿って,MEGとEEGのMNEによる解析手順を説明します.
実行環境
- Mac OS 10.15.3
- Python 3.6.5
- mne 0.20.4
インストール
Anacondaでもpipでもインストールできます.公式ページではAnacondaの方がおすすめだそうです.
- Anacondaの場合
conda install -c conda-forge mne
- pipの場合
pip install -U mne
また,GPUの設定もできるようなので,フィルタリングやサンプリングで時間がかかるようであれば設定すると良いかもしれません.
$ conda install cupy
$ MNE_USE_CUDA=true python -c "import mne; mne.cuda.init_cuda(verbose=True)"
Enabling CUDA with 1.55 GB available memory
>>> mne.utils.set_config('MNE_USE_CUDA', 'true')
# mne.io.Raw.filter()などにn_jobs='cuda'を渡す
MEG/EEG解析のチュートリアル
こちらのチュートリアルに沿って,脳波解析の典型的な処理とMNEの基本的なデータ構造について説明します.適当に簡潔にかいつまんで説明するので,英語と脳波解析の知識がある方は元のページを読んでもらったほうがよろしいかと思います.
解析手順
モジュールのインポート
import os
import numpy as np
import mne
データのロード
mneにサンプルとして用意されているデータをロードします(全データセットの一覧).ちなみに,MEGやEEGにはデバイスごとにいくつかファイルフォーマットがあるのですが,それらのデータをロードする関数が用意されています(サポートされているデータフォーマット)
sample_data_folder = mne.datasets.sample.data_path()
sample_data_raw_file = os.path.join(sample_data_folder, 'MEG', 'sample',
'sample_audvis_filt-0-40_raw.fif')
raw = mne.io.read_raw_fif(sample_data_raw_file)
今回はsample_audvis_filt-0-40_raw.fifという40Hzでローパスフィルタ済みのデータを使用しています.データ(1.54GB)のダウンロードには1分ほど要しました.
mne.io.read_raw_fif()という関数により,fifフォーマットのファイルからRawオブジェクトと呼ばれるMNEのデータ構造のデータを読み込んでいます.
ちなみにこのデータの実験では,被験者が視覚刺激と聴覚刺激を左右ランダムに750msごとに呈示されます.また時々画面に笑った顔が表示され,被験者はそれを見たときにボタンを押すというものだそうです.これらの刺激の呈示のことをイベントと呼びます.
ロードしたオブジェクトについて表示します.
print(raw)
print(raw.info)
# output
<Raw | sample_audvis_filt-0-40_raw.fif, 376 x 41700 (277.7 s), ~3.6 MB, data not loaded>
<Info | 15 non-empty values
bads: 2 items (MEG 2443, EEG 053)
ch_names: MEG 0113, MEG 0112, MEG 0111, MEG 0122, MEG 0123, MEG 0121, MEG ...
chs: 204 GRAD, 102 MAG, 9 STIM, 60 EEG, 1 EOG
custom_ref_applied: False
dev_head_t: MEG device -> head transform
dig: 146 items (3 Cardinal, 4 HPI, 61 EEG, 78 Extra)
file_id: 4 items (dict)
highpass: 0.1 Hz
hpi_meas: 1 item (list)
hpi_results: 1 item (list)
lowpass: 40.0 Hz
meas_date: 2002-12-03 19:01:10 UTC
meas_id: 4 items (dict)
nchan: 376
projs: PCA-v1: off, PCA-v2: off, PCA-v3: off, Average EEG reference: off
sfreq: 150.2 Hz
>
MNEには,Raw, Epochs, Evokedオブジェクトと呼ばれるオブジェクトによってデータが管理されています.データの時間的な区切られ方によってこれらは区別されており,Rawでは脳波の収録開始から停止までのすべての時間のデータを含んでおり,イベントにより区切られる前の状態だと考えてもらうと良いと思います.raw.infoではrawのチャンネル数や名前,サンプリングレートなどの情報が保持されています.
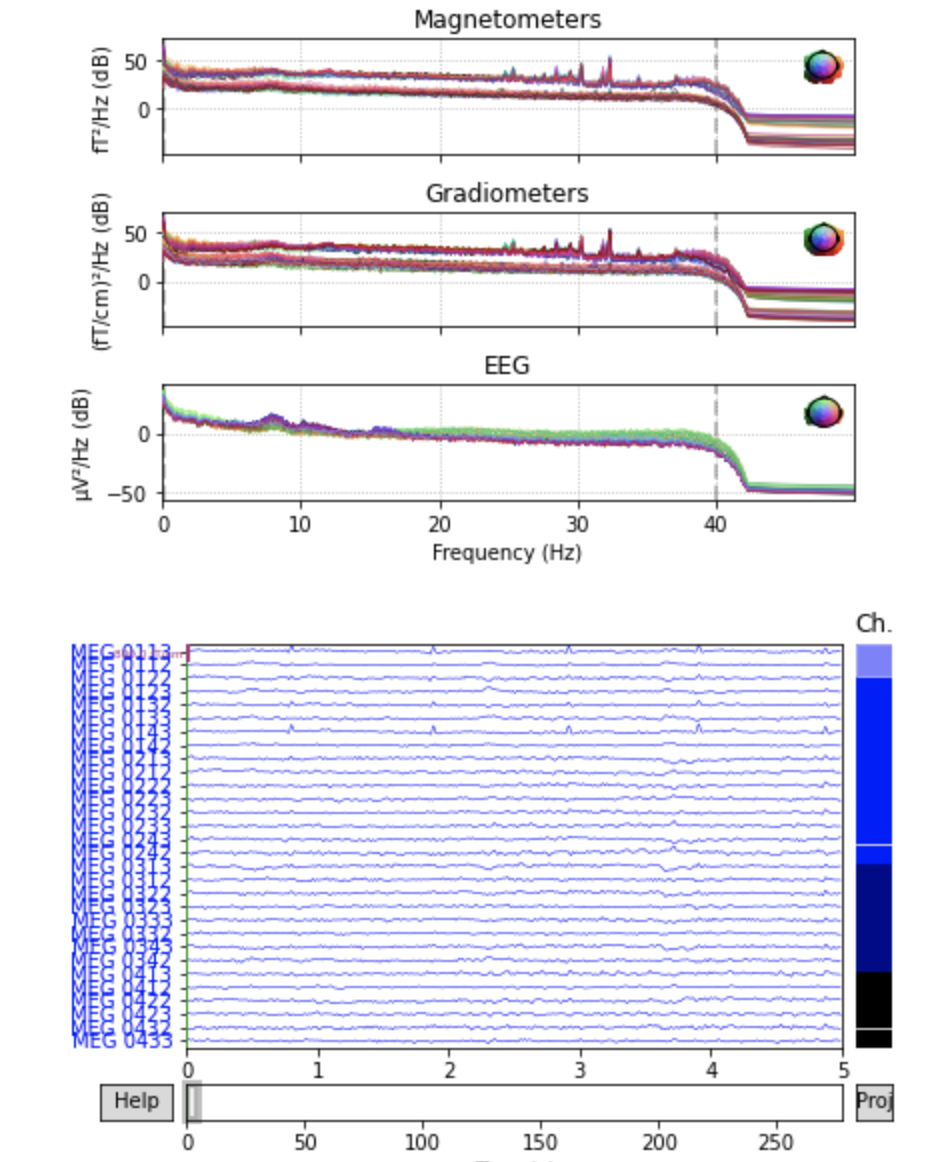
このRawオブジェクトのpower spectral density(PSD)とセンサーごとの波形を表示します.plot()はインタラクティブシェルではスクロールやスケーリングが可能です.
raw.plot_psd(fmax=50)
raw.plot(duration=5, n_channels=30)
前処理
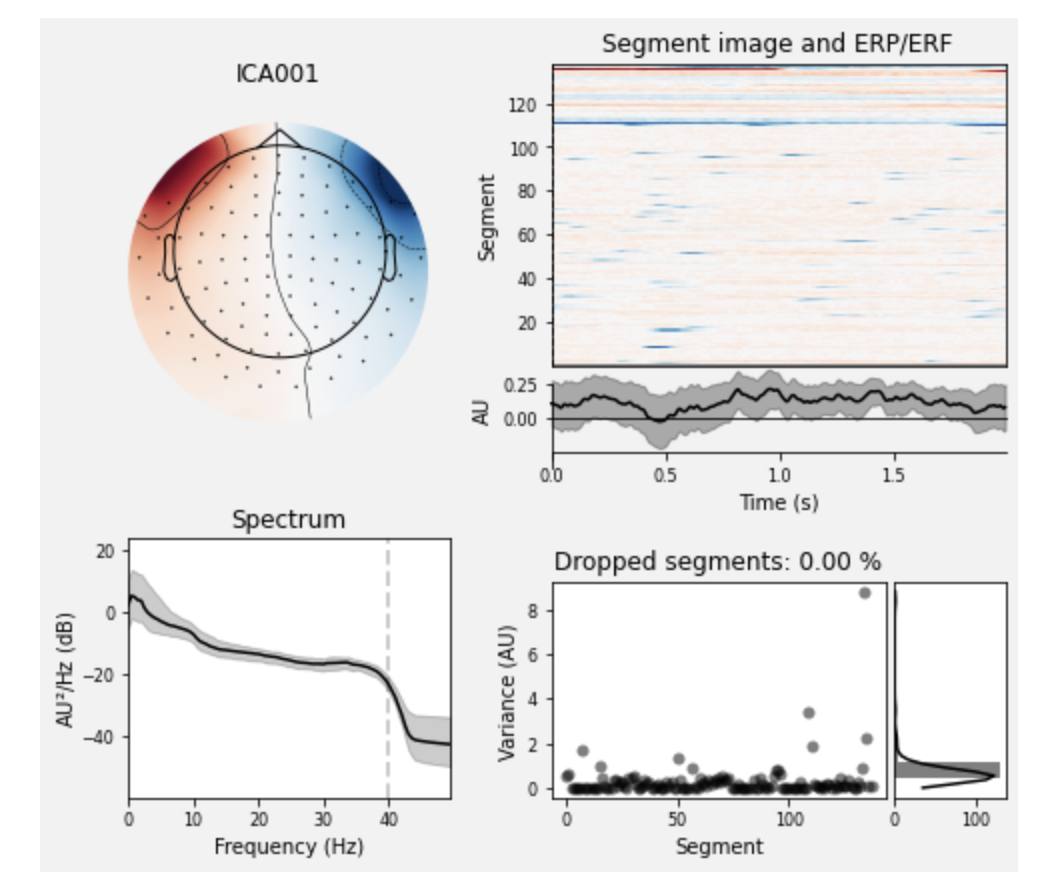
EEG/MEG解析のノイズ処理などでは定番の独立成分分析(ICA)を行います.簡単に言うと,多チャンネルの信号を独立した成分に分解することで,電源ノイズや瞬きなど脳活動に独立なものを取り除く処理を行います.
ここでは,364チャンネルの信号に対して,20個の独立成分に分解した後,[1, 2]と取り除く成分のインデックスを指定しています(実際の除去は,次のica.apply(raw)によって行われます).ica_properties()で,独立成分の情報を表示します.
# set up and fit the ICA
ica = mne.preprocessing.ICA(n_components=20, random_state=97, max_iter=800)
ica.fit(raw)
ica.exclude = [1, 2] # details on how we picked these are omitted here
ica.plot_properties(raw, picks=ica.exclude)
# output
Fitting ICA to data using 364 channels (please be patient, this may take a while)
Inferring max_pca_components from picks
Selecting by number: 20 components
Fitting ICA took 2.4s.
Using multitaper spectrum estimation with 7 DPSS windows
138 matching events found
No baseline correction applied
Not setting metadata
0 projection items activated
0 bad epochs dropped
138 matching events found
No baseline correction applied
Not setting metadata
0 projection items activated
0 bad epochs dropped

指定した独立成分を除去した信号を手に入れるため,先ほどのICAオブジェクトをRawオブジェクトに適用します.
orig_raw = raw.copy()
raw.load_data()
ica.apply(raw)
前頭のチャンネルを表示してみると次の2つの図のようにノイズが除去されています.
# show some frontal channels to clearly illustrate the artifact removal
chs = ['MEG 0111', 'MEG 0121', 'MEG 0131', 'MEG 0211', 'MEG 0221', 'MEG 0231',
'MEG 0311', 'MEG 0321', 'MEG 0331', 'MEG 1511', 'MEG 1521', 'MEG 1531',
'EEG 001', 'EEG 002', 'EEG 003', 'EEG 004', 'EEG 005', 'EEG 006',
'EEG 007', 'EEG 008']
chan_idxs = [raw.ch_names.index(ch) for ch in chs]
orig_raw.plot(order=chan_idxs, start=12, duration=4)
raw.plot(order=chan_idxs, start=12, duration=4)
# output
Reading 0 ... 41699 = 0.000 ... 277.709 secs...
Transforming to ICA space (20 components)
Zeroing out 2 ICA components


イベント検出
このサンプルデータにはSTIMチャンネルと呼ばれるものが含まれており,そのチャンネルにイベント種類とその時刻のデータがあります.今回はSTI 014という名前のチャンネルが該当するため,それを指定して実験のイベントを取り出します.
events = mne.find_events(raw, stim_channel='STI 014')
print(events[:5]) # show the first 5
# output
319 events found
Event IDs: [ 1 2 3 4 5 32]
[[6994 0 2]
[7086 0 3]
[7192 0 1]
[7304 0 4]
[7413 0 2]]
eventsはnumpy arrayであり,最初の列がサンプリングの時刻,最後の列がイベントの種類(ID)を表しています(真ん中の0は無視して良いです).ここでは,IDが[ 1 2 3 4 5 32]のイベントがあるそうです.
| Event ID | Condition |
|---|---|
| 1 | auditory stimulus (tone) to the left ear |
| 2 | auditory stimulus (tone) to the right ear |
| 3 | visual stimulus (checkerboard) to the left visual field |
| 4 | visual stimulus (checkerboard) to the right visual field |
| 5 | smiley face (catch trial) |
| 32 | subject button press |
Event IDとそのイベントの説明を辞書型で格納します.ちなみにここでのkeyは任意の文字列で構いません.
event_dict = {'auditory/left': 1, 'auditory/right': 2, 'visual/left': 3,
'visual/right': 4, 'smiley': 5, 'buttonpress': 32}
時間軸上にそれぞれのイベントの呈示タイミングをプロットしてみます.event_id=evend_dictによって先ほど定義したIDとイベントの名前がプロット上で対応づいています.
fig = mne.viz.plot_events(events, event_id=event_dict, sfreq=raw.info['sfreq'],
first_samp=raw.first_samp)

エポックに切り出す
MEG/EEG解析で,特にある刺激に対する反応について調べる際に,信号をエポックと呼ばれる単位に分けて,一つのデータとして扱います.刺激に対して前後の一定の区間を切り出すことで,それを一つのデータ単位とします.この切り出し処理のことをエポッキングと呼びます.Rawオブジェクトは収録中の全時間がつながっているものですが,これらを刺激に対する反応の部分のみ切り出したものが,Epochsオブジェクトというデータ型で保持されます.
エポッキングにおいて,しばしばスレッショルドリジェクションと呼ばれる処理が行われます.これは該当の時間中に大きすぎる信号が含まれている場合,その部分は大きなノイズが入り込んでしまったであろうということで,データから除外するというものです.その除外の基準を決めます.
reject_criteria = dict(mag=4000e-15, # 4000 fT
grad=4000e-13, # 4000 fT/cm
eeg=150e-6, # 150 µV
eog=250e-6) # 250 µV
次に,RawオブジェクトからEpochsオブジェクトを作成します.ここでのmne.Epochsの引数は以下の通りです.
-
raw:信号のデータを含んだRawオブジェクト -
events:時間とイベントIDの情報を持ったnumpy array -
event_id:イベントIDとその名称を対応付ける辞書型 -
tmin:刺激呈示の何秒からのデータを切り出すか(刺激呈示前の場合マイナスを付ける) -
tmax:刺激呈示後何秒までのデータを切り出すか -
reject:データ除外の基準
epochs = mne.Epochs(raw, events, event_id=event_dict, tmin=-0.2, tmax=0.5,
reject=reject_criteria, preload=True)
# output
319 matching events found
Applying baseline correction (mode: mean)
Not setting metadata
Created an SSP operator (subspace dimension = 4)
4 projection items activated
Loading data for 319 events and 106 original time points ...
Rejecting epoch based on EOG : ['EOG 061']
Rejecting epoch based on EOG : ['EOG 061']
Rejecting epoch based on MAG : ['MEG 1711']
Rejecting epoch based on EOG : ['EOG 061']
Rejecting epoch based on EOG : ['EOG 061']
Rejecting epoch based on MAG : ['MEG 1711']
Rejecting epoch based on EEG : ['EEG 008']
Rejecting epoch based on EOG : ['EOG 061']
Rejecting epoch based on EOG : ['EOG 061']
Rejecting epoch based on EOG : ['EOG 061']
10 bad epochs dropped
今回は10個のエポックが除外されたようです.
被験者のボタンを押したタイミングは今回扱わないので,視聴覚刺激イベントのみを対象にします.また,条件間でのデータ数に偏りが生じることを防ぐため,epochs.equalize_event_counts()で,数が多い条件のイベントを消すことで,各条件の数を揃えています.
conds_we_care_about = ['auditory/left', 'auditory/right',
'visual/left', 'visual/right']
epochs.equalize_event_counts(conds_we_care_about) # this operates in-place
event_dictで'auditory/left'とauditory/rightのように指定していましたが,Epochsオブジェクトに渡すことで,auditoryを指定するとこれら両方を指すことになります.ここで聴覚刺激に対するデータと視覚刺激データに分けます.
aud_epochs = epochs['auditory']
vis_epochs = epochs['visual']
del raw, epochs # free up memory
聴覚データでチャンネルを指定して,刺激呈示時間を揃えた加算平均波形を表示します.
aud_epochs.plot_image(picks=['MEG 1332', 'EEG 021'])
# output
136 matching events found
No baseline correction applied
Not setting metadata
0 projection items activated
0 bad epochs dropped
136 matching events found
No baseline correction applied
Not setting metadata
0 projection items activated
0 bad epochs dropped


補足ですが,Epochsオブジェクトのget_data()メソッドによって,numpy arrayの形で時系列波形データを取得できるので,条件ごとにarrayを取ることで,機械学習のモデルに入力することなどが可能です.
aud_epochs.get_data().shape
(136, 376, 106) # (エポック,チャンネル,時刻)
以下の周波数処理などもnumpy arrayを返すものも同様です.
時間周波数解析
MEG/EEG波形における,刺激呈示前後の時間と周波数の関係について調べるために,時間周波数解析を行います.ここでは,聴覚刺激に対する反応を,wavelet解析によって計算し,それをプロットします.カラーバーに表示されているように,赤が濃くなるほど,その時間のその周波数の振幅が大きいということを示しています.(もっと詳しいExample)
frequencies = np.arange(7, 30, 3)
power = mne.time_frequency.tfr_morlet(aud_epochs, n_cycles=2, return_itc=False,
freqs=frequencies, decim=3)
power.plot(['MEG 1332'])
# output
No baseline correction applied

誘発反応の推定
MNEのデータ型として,Epochsオブジェクトの加算平均を行ったものがEvokedオブジェクトになります.MEG/EEGデータは脳活動以外のノイズが多く,加算平均を行うことで条件間の差を調べることが多くあります.
ここでは,聴覚データと視覚データの加算波形の違いをプロットします.
aud_evoked = aud_epochs.average()
vis_evoked = vis_epochs.average()
mne.viz.plot_compare_evokeds(dict(auditory=aud_evoked, visual=vis_evoked),
legend='upper left', show_sensors='upper right')

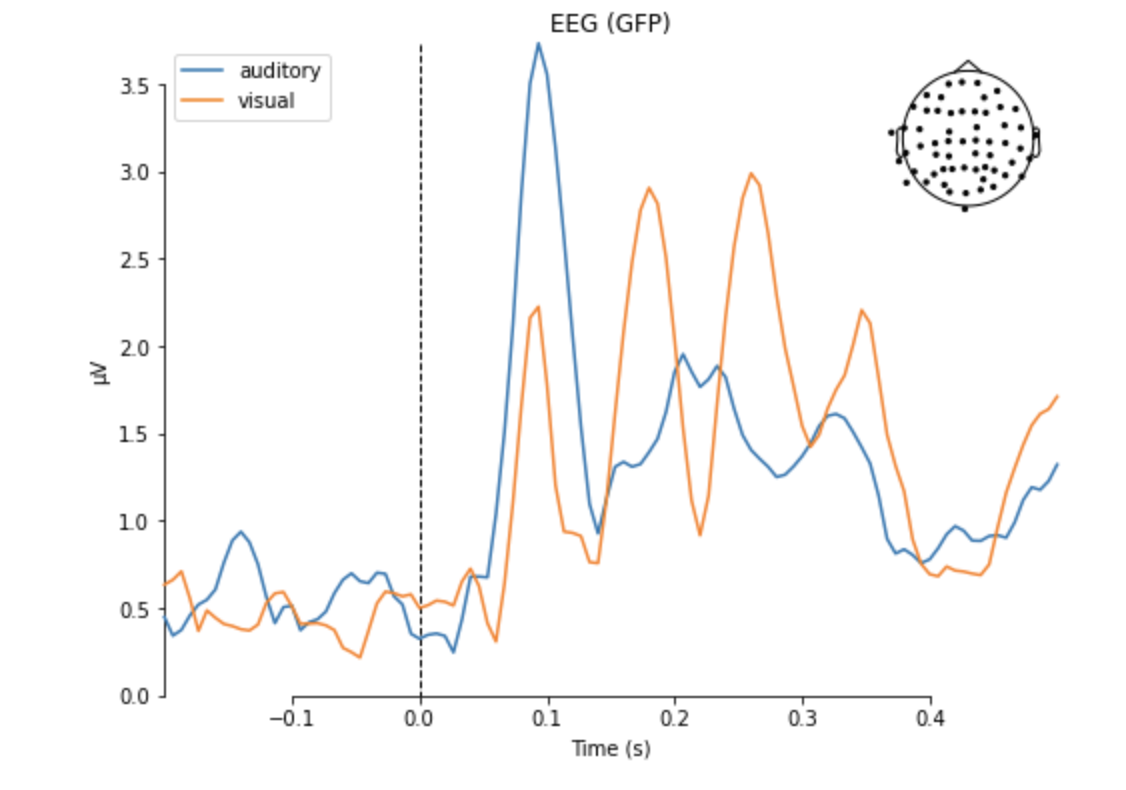

Evokedオブジェクトはさらに詳しい解析を行うメソッドを備えています.各チャンネルごとの波形を表示すると,前頭のチャンネルで100msで負になり(N100),そこから200msあたりで正のピークを持つ振れ(P200)を確認できます.
aud_evoked.plot_joint(picks='eeg')

また,トポグラフィーと呼ばれる頭皮上の分布を表示することもできます.ここでは,赤いほど正の,青いほど負の電位を持った脳波が以下のように頭皮上で観測されていることが分かります.
aud_evoked.plot_topomap(times=[0., 0.08, 0.1, 0.12, 0.2], ch_type='eeg')

また,条件間の差を見るためにmne.combine_evoked()によって,2つの条件差のデータを持ったEvokedオブジェクトが生成されます(このとき片方のオブジェクトにマイナス符号をつけます).このオブジェクトの波形を表示すると,2つの条件間の差の波形を見ることができます.
evoked_diff = mne.combine_evoked([aud_evoked, -vis_evoked], weights='equal')
evoked_diff.pick_types('mag').plot_topo(color='r', legend=False)

おわりに
ここでは基礎的な機能のみ紹介しましたが,MNE公式ページには多くのTutorialとExampleが用意されているので,さらに詳しい分析を行うための最初のステップとして参考にしていただけたらと思います.
また,Pythonには統計や機械学習のライブラリが多くあるので,MEG/EEGの機械学習などによる分析が容易になると思います.