はじめに
次世代シークエンサー(NGS)のデータ解析はLinuxで行うことが一般的で、どの入門書を読んでもLinux、もしくはそれをベースにしたOSであるMacで行うことが当然とされています。しかし、そもそも、WindowsではNGSデータの解析は難しいのでしょうか?本記事ではそのことを検証し、WindowsユーザーがNGS解析を実施するための環境構築の方法を考えます。
Windows用のNGS解析アプリは非常に少ない
いきなり結論ですが、残念ながらNGS解析アプリはほとんどWindowsには対応していません。
Javaで書かれたゲノムブラウザならWindowsでも動きますが、基本的な相同性検索ツールであるBLASTやマッピングツールのBowtieなどがLinuxもしくはMacでしか動かないので、Windowsによる解析はほぼ不可能な状況です。
そうだ!WSLがある!
以前であればここで手詰まりで、VMwareなどの仮想環境を構築する以外に方法はなかったのですが、Windowsも状況が変わりつつあります。Windows 10 version 1709以降は、WSL(Windows Subsystem for Linux)が利用可能になりました。これによりWindowsユーザーは簡単な設定をするだけで、Linux環境をコマンドプロンプトを扱うかのように、つまりWindows上の1つのアプリケーションかのように扱うことができます。
WSLの詳細については、 【WSL入門】第1回 Windows 10標準Linux環境WSLを始めよう などの記事をご覧ください。
本記事ではWSLを用いて、Windowsにバイオインフォマティクス環境を構築する方法を説明します。なお、自分自身がまだ初心者であるゆえに間違いなどあると思いますので、その点はご容赦ください。何かあればご指摘いただければ幸いです。
この記事は、バイオインフォマティクス分野の入門書のバイブルである「生命科学者のためのDr.Bonoデータ解析実践道場 (坊農秀雅先生)」の内容を参考にしています。詳しい内容についてはそちらをご覧ください。また、詳しいLinuxのコマンド等についても説明していません。
なお、WSLの後継となるWSL2の開発も進んでいるようです。WSLはLinuxのエミュレーターであり、Linuxのコマンドを内部でWindowsのコマンドに変換して実行していたのですが、WSL2ではWindowsの中に完全なLinuxカーネルが入るらしく、仮に今までのWSLでは実行できないことがあってもWSL2では改善されているかもしれません。この辺はWSL2の正式版がリリースされたら検証してみたいと思っています。
Linuxのインストール・設定
WSL(Windows Subsystem for Linux)を有効にする
Windowsの設定アプリで、検索欄に「Windowsの機能の有効化または無効化」と入力し、出てきた項目をクリックします。(→ 「Windowsの機能」ダイアログが出てくる)
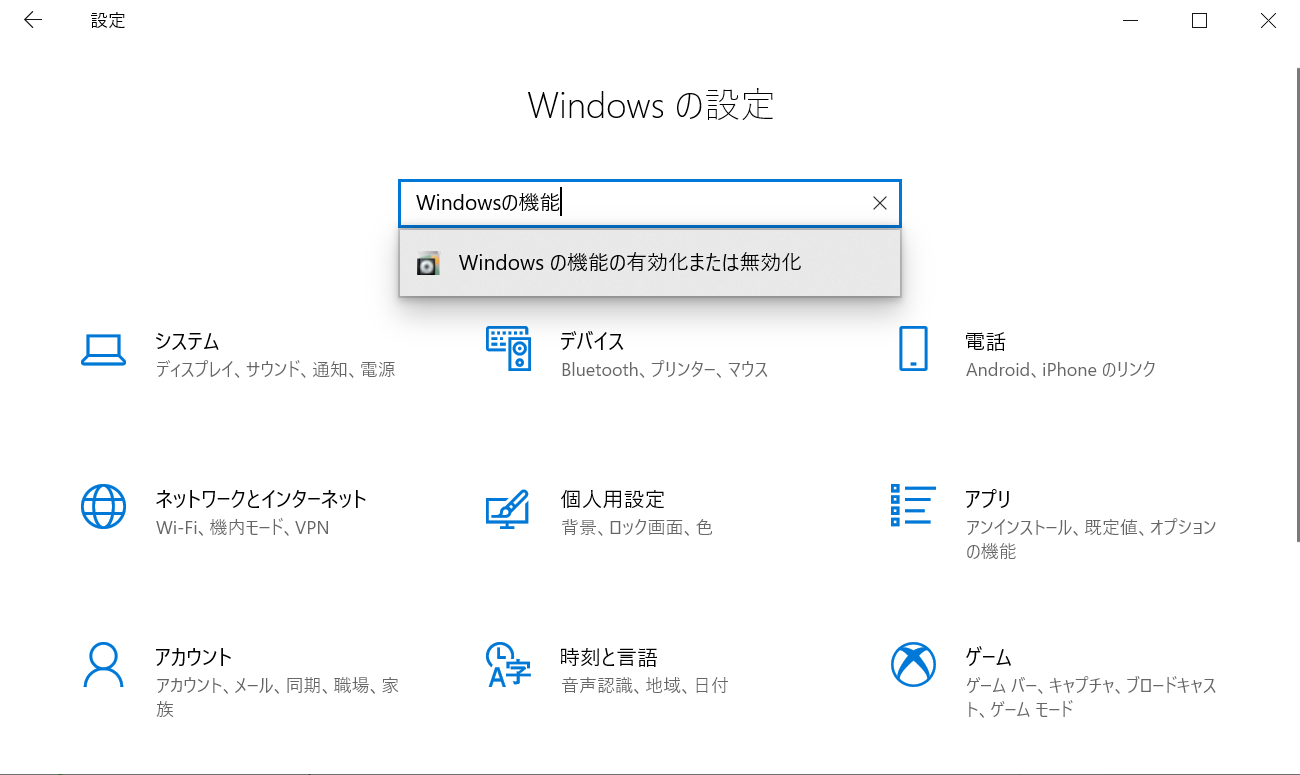
Windows Subsystem for Linux のチェックを入れて、OKをクリックする
再起動が要求されるので、再起動します。
Microsoft StoreからLinuxをインストール
続いて、Linuxをインストールします。インストールするLinuxのバージョンは任意ですが、ここでは最も一般的なUbuntuをインストールします。
Microsoft Storeで検索すると、「Ubuntu」「Ubuntu 18.04 LTS」「Ubuntu 16.04 LTS」の3種類が出てきますが、それぞれ以下のような違いがあります。
- Ubuntu:インストール時点でのUbuntuの最新バージョンをインストールする (現在はUbuntu 20.04 LTS)
- Ubuntu 18.04 LTS:Ubuntu 18.04 LTS とバージョンを指定してインストールする
- Ubuntu 20.04 LTS:Ubuntu 20.04 LTS とバージョンを指定してインストールする

初回起動時に、ユーザー名とパスワードを設定
インストールされたUbuntuを起動します。ここで出てくる黒い画面にキーボードで文字(コマンド)を打ち込んで、Enterを押すことで以降の操作をしていきます。
初回起動時にユーザー名とパスワードを設定するように要求されるので、設定してください。
(パスワードの設定の際に、キーボードで入力してもWindowsのように「●●●●」などの文字列は表示されませんが、ちゃんと入力できているのでそのまま続けてください)
Ubuntuを最新の状態にアップデートする
以下のコマンドを入力して、インストールしたUbuntuを最新の状態にアップデートします。
sudo apt update
データの保存先
Windows側からUbuntuの本体にアクセスすることは推奨されていませんが、Ubuntu側からWindowsのフォルダにアクセスすることは可能です。
例えば D:\UbuntuData というフォルダをWindowsで作成した場合は、以下のコマンドでUbuntuからそのフォルダに移動することができます。
cd /mnt/d/UbuntuData
NGS解析のデータをUbuntuからもWindowsからもアクセスできるようにするために、Windows側にデータを保存するフォルダを作成するとよいと思われます。
コマンドをコピー&ペーストで貼り付けできるようにする(おまけ)
そのままの初期設定では、別の場所でコピーしたコマンドをUbuntuにペーストして貼り付けることができません。そこで、以下のようにUbuntu上でのコピー&ペーストを有効化しましょう。
Ubuntuのウィンドウの左上のロゴマークをクリックし、プロパティをクリックします。
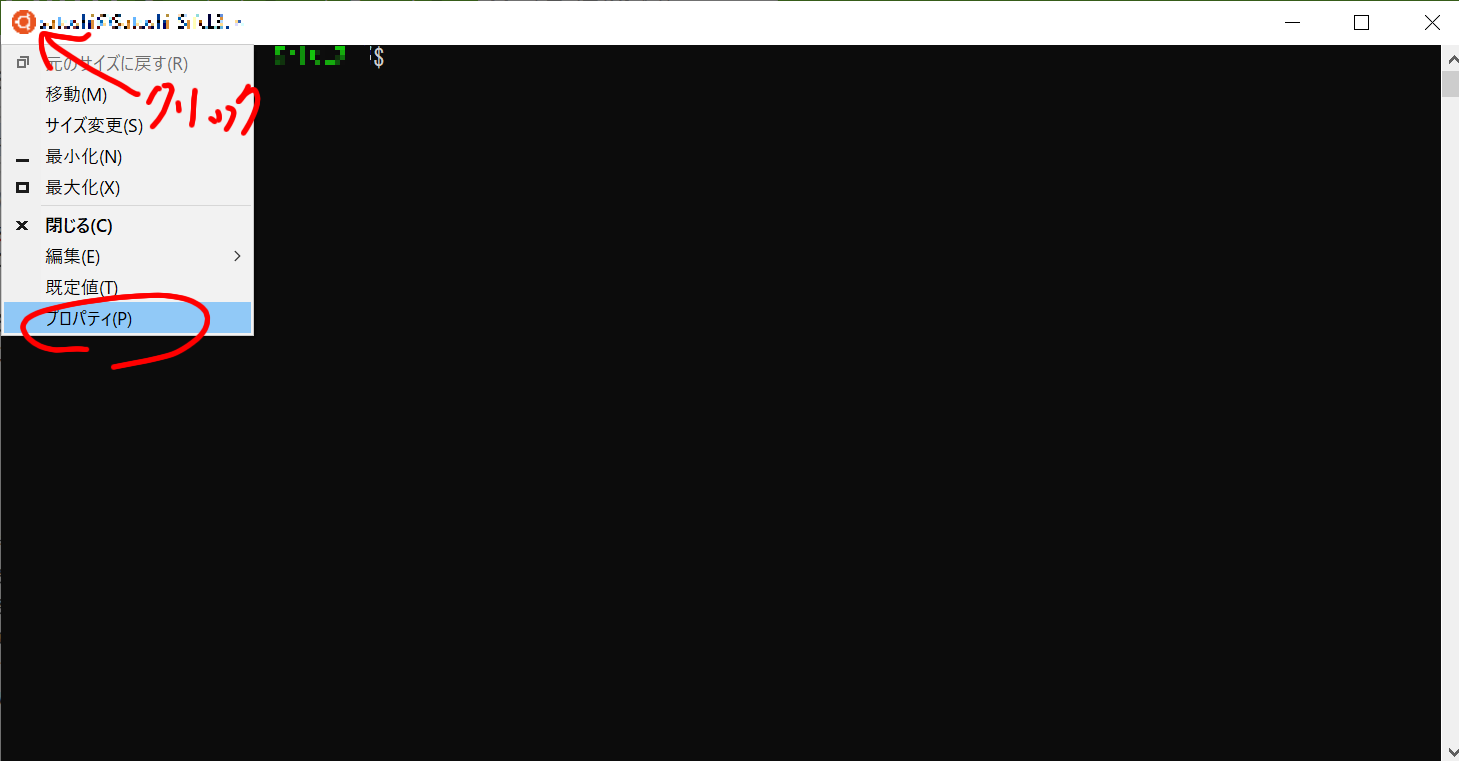
出てくるウィンドウの「Ctrl+Shift+C/Vをコピー/貼り付けとして使用する」にチェックを入れてOKをクリックします。
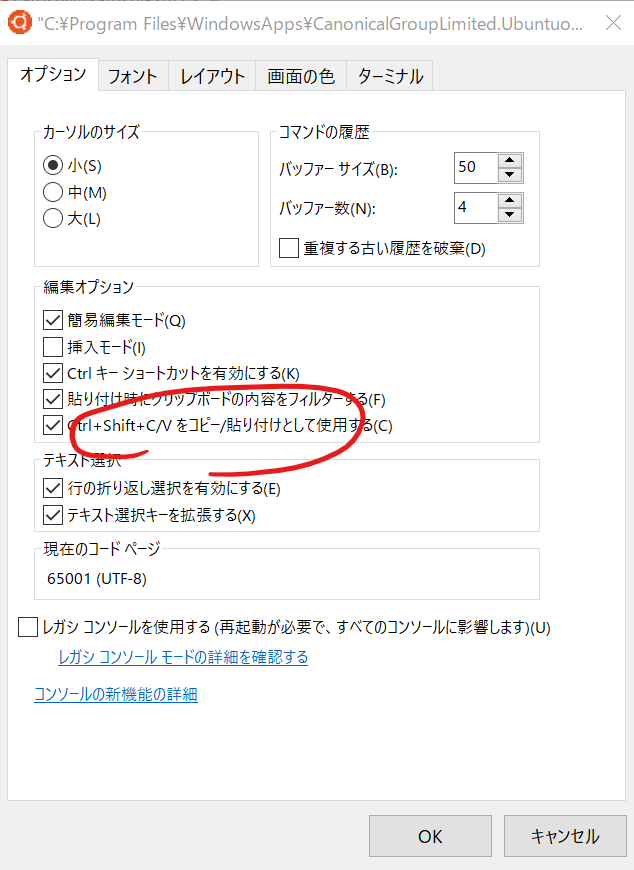
これでコピー&ペーストが有効になりました。
なお、ペーストは Ctrl+Shift+V で、Shiftを押す必要があることに注意してください。
NGS解析環境の構築
Anacondaをインストール
Ubuntuにインストールするアプリケーションを自分ですべて管理することは困難なので、何らかのパッケージマネージャを使用することをお勧めします。パッケージマネージャを使用することで、どのアプリのどのバージョンを、どこのフォルダにインストールしたかや、インストールしたアプリケーションの依存関係についても意識することなく、コマンド一つで簡単にインストールすることができるようになります。
ここでは、一般的に使われていて、バイオインフォマティクスツールも広くカバーしているAnacondaをインストールします。
Anacondaのうち最小限のパッケージで構成されているMinicondaのインストーラーをカレントディレクトリへダウンロード
curl -O https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh
ダウンロードしたインストーラーを実行する(カレントディレクトリへインストールされる)
sh Miniconda3-latest-Linux-x86_64.sh
Anaconda(Miniconda)がインストール出来たら、一度Ubuntuを再起動します。
アプリケーションの登録してあるchannelを追加
Anacondaのサーバーには様々なchannel(=アプリケーションを登録する場所)があり、その中からバイオインフォマティクスツールが多く登録されているchannelをAnacondaに登録しておきます。
conda config --add channels defaults
conda config --add channels conda-forge
conda config --add channels bioconda
代表的なツールをインストールする
以下のように conda install (アプリ名) でインストールができます。
conda install blast
conda install sra-tools
conda install samtools
インストールしたアプリケーションの一覧は以下のコマンドで確認できます。
conda list
(デフォルトでインストールされたアプリケーションもたくさん含まれています)
WSLを使用するにあたっての注意事項・不具合
まだ一部の機能しか使っていませんが、基本的なNGS解析関係のアプリケーションはWSLでも問題なく動いていそうでした。アプリケーションの実行速度にも大きな違いはなさそうです。
以下に、WSLを使っていて遭遇した不具合や注意について記載しておきます。
byobuがそのままでは使えない
大きなサイズのリファレンスデータのダウンロードや時間のかかる処理を行う際に、byobuという仮想端末管理ソフトを用いることで、途中で中断してしまっても続きから再開できるようになります。
byobuのインストールはAnacondaから簡単にできますが、WSLでは不具合がありそのままでは使えません。詳細は次の記事をご覧ください。
Windows Subsystem for Linuxでディストリビューションを区別するためにByobuを使う
UbuntuにはRubyがデフォルトではインストールされていない
macOSにはRubyがあらかじめインストールされているので、macOSを対象にした入門書ではRubyはインストール済みとして書かれていますが、UbuntuにはRubyはデフォルトでインストールされていないので、Rubyの機能がうようなときは追加する必要があります。
おまけ:"Windowsの中のLinuxで解析"ではなく、"Windowsで解析"したい!
ここでは、バイオインフォマティクス環境の構築のためにWindowsにWSLをインストールする方法を書きました。これでWindowsを用いたバイオインフォマティクスが可能になりますが、でも結局Windowsの中の"Linuxで解析"しているに過ぎませんよね?そうではなくて"Windowsで解析"することはできないのでしょうか?
結論から言うと、本当の意味でWindowsだけで解析することは無理なのですが、Windowsで解析している感覚にさせることは可能です。Visual Studio CodeからリモートでWSLに接続し、Windowsのアプリケーション内からLinux環境を操作することができます。もちろん、ファイルの管理はWindowsのシステム内で管理できます。そして、Linuxのシェルスクリプトの代わりにPythonを用います。こうすれば実質的にはWindowsで解析しているような感覚になると思います。(まあ、気持ちの問題かもしれませんが...)
詳しいやり方については、自分のブログの方に書かせていただきました。宣伝になってしまい申し訳ないですが、こちらの方もよろしければご覧ください。