概要
medibaアドベントカレンダーの7日目の記事です。
自身の学習も兼ねて、R言語のモダンな機械学習フレームワークである tidymodels を使った機械学習の基本的なワークフローをまとめていきます。
tidymodelsは、tidyverseの哲学に基づいたパッケージ群で、データの前処理からモデルの構築、評価、チューニングまでを一貫した文法で、整然と(Tidyに)実装することができます。
今回利用するデータセットについて
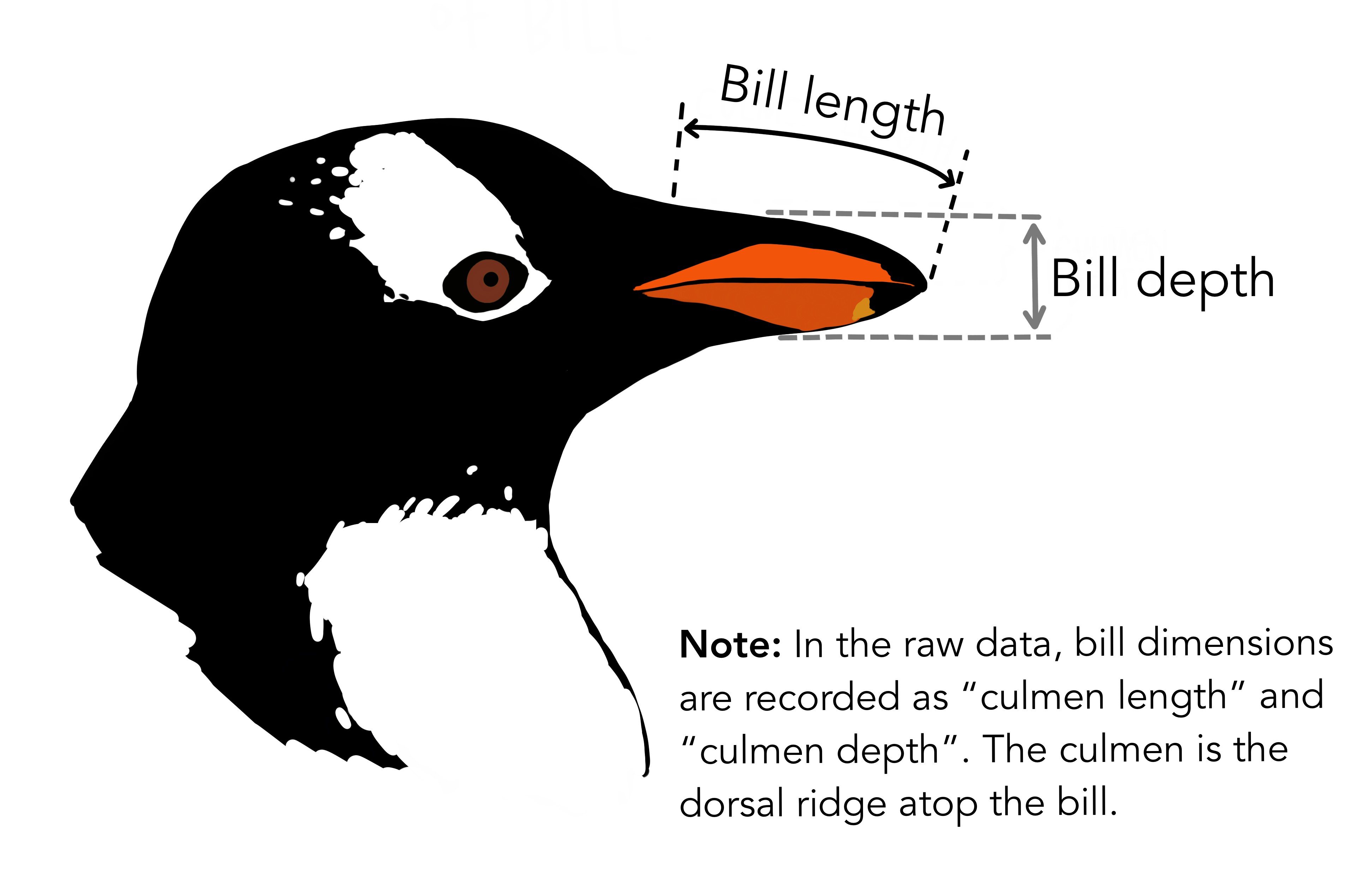
今回はpalmerpenguinsというデータセットを使用します。
これはRでおなじみのiris(アヤメ)データセットに代わるデータセットです。
irisと比べて、欠損値(NA)を含んでいたり変数の関係性が少し複雑だったりするため、より実践的なデータ前処理の練習になります。
このデータセットには、南極のパーマー諸島に生息するアデリー、ヒゲ、ジェンツーの3種類のペンギンの、くちばしの長さや深さ、フリッパー(翼)の長さ、体重、性別などの情報が含まれています。
この記事では、これらの特徴量からペンギンの種類(species)を予測する分類モデルを作っていきます。
ライブラリの読み込み
まずは必要なライブラリを読み込みます。
tidymodelsをロードすることで、recipesやparsnip、workflowsなど、機械学習に必要な中核パッケージが一括で読み込まれます。palmerpenguins(データセット)とtidyverse(データ操作・可視化)も併せてロードしておきます。
library(tidymodels)
library(palmerpenguins)
library(tidyverse)
データのロード
data(penguins) でデータをロードし、glimpse()関数でデータの構造を概観します。
# penguinsデータをロード(NAが含まれている状態)
data(penguins)
# データセットの中身を確認
glimpse(penguins)
Rows: 344
Columns: 8
$ species <fct> Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Ad…
$ island <fct> Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, Torgersen…
$ bill_length_mm <dbl> 39.1, 39.5, 40.3, NA, 36.7, 39.3, 38.9, 39.2, 34.1, 42.0, 37.8, 37.8, 41.1, 38.6, 34.6, 36.6, 38.7, 42.5, 34.4, 46.0, 37.8, 37.7, …
$ bill_depth_mm <dbl> 18.7, 17.4, 18.0, NA, 19.3, 20.6, 17.8, 19.6, 18.1, 20.2, 17.1, 17.3, 17.6, 21.2, 21.1, 17.8, 19.0, 20.7, 18.4, 21.5, 18.3, 18.7, …
$ flipper_length_mm <int> 181, 186, 195, NA, 193, 190, 181, 195, 193, 190, 186, 180, 182, 191, 198, 185, 195, 197, 184, 194, 174, 180, 189, 185, 180, 187, 1…
$ body_mass_g <int> 3750, 3800, 3250, NA, 3450, 3650, 3625, 4675, 3475, 4250, 3300, 3700, 3200, 3800, 4400, 3700, 3450, 4500, 3325, 4200, 3400, 3600, …
$ sex <fct> male, female, female, NA, female, male, female, male, NA, NA, NA, NA, female, male, male, female, female, male, female, male, fema…
$ year <int> 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007…
>
glimpse()の結果から、bill_length_mm(くちばしの長さ)やsex(性別)など、いくつかの列に欠損値(NA)が含まれていることがわかります。また、species(予測対象)やisland、sexが因子(fct)型であることも確認できます。
データの分割
モデルの性能を未知のデータで正しく評価するために、手元のデータを訓練用データ(Training data)とテスト用データ(Test data)に分割します。
tidymodelsではinitial_split関数を使います。
今回は、全データの80%(prop = 0.8)を訓練用に、残りをテスト用に分けます。 また、strata = speciesを指定することで、分割後の両方のデータセットで、目的変数であるペンギンの種類(species)の比率が元データと等しくなるように(=層化サンプリング)しています。これにより、特定の種類のペンギンが訓練データだけに偏る、といった事態を防ぎます。
set.seed(123)で乱数を固定し、誰でも同じ結果(分割)を再現できるようにしておきます。
set.seed(123)
penguins_split <- initial_split(penguins, prop = 0.8, strata = species)
# 設定に基づき、訓練用データとテスト用データを作成
penguins_train <- training(penguins_split)
penguins_test <- testing(penguins_split)
データ件数を確認
training()関数とtesting()関数で分割したデータを抽出し、それぞれの行数(件数)を確認します。set.seed(123)で乱数を固定した結果、今回は訓練用が274件、テスト用が70件となりました。
cat("訓練用:", nrow(penguins_train), "件、テスト用:", nrow(penguins_test), "件\n")
訓練用: 274 件、テスト用: 70 件
前処理レシピの作成
tidymodelsの強力な機能の一つが、このrecipe(レシピ)です。
これはデータの前処理の手順書のようなもので、モデルに投入する前のデータをどのように加工するかを定義します。
レシピを使うことで、訓練データに行った処理と全く同じ処理を後でテストデータや新しいデータにも情報漏洩(data leakage)の心配なく、簡単に適用できます。
今回のレシピでは、以下のステップを定義しました。
- 式の定義:
recipe(species ~ ., ...)で、species(種類)を予測したい目的変数、それ以外のすべての変数を説明変数(.で指定)として定義します。 - 欠損値の補完:
step_impute_knn(all_predictors())を使い、k-NN(k近傍法)で欠損値を補完します。 - ダミー変数化:
step_dummy(all_nominal_predictors())で、islandやsexといった名義変数(カテゴリ変数)をダミー変数(0か1の値を取る変数)に変換します。 - 正規化:
step_normalize(all_numeric_predictors())で、くちばしの長さなどのすべての数値変数を標準化(平均0、標準偏差1)します。
penguins_recipe <-
recipe(species ~ ., data = penguins_train) %>%
step_impute_knn(all_predictors()) %>%
step_dummy(all_nominal_predictors()) %>%
step_normalize(all_numeric_predictors())
レシピを作成した段階では、まだデータには適用されていません。あくまで「手順書」を作成しただけ、という点に注意してください。
レシピの確認
print(penguins_recipe)
── Recipe ────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────
── Inputs
Number of variables by role
outcome: 1
predictor: 7
── Operations
• K-nearest neighbor imputation for: all_predictors()
• Dummy variables from: all_nominal_predictors()
• Centering and scaling for: all_numeric_predictors()
モデルの指定
次に使用する機械学習モデルを指定します。
tidymodelsでは、parsnipパッケージがこの役割を担っており、モデルの種類に関わらず統一された記法でモデルを定義できます。
今回は、ランダムフォレスト(Random Forest)を選択しました。
- モデルのタイプ:
rand_forest()で、ランダムフォレストモデルを使用することを宣言します。 - タスクの種類:
set_mode("classification")で、今回のタスクが「分類(classification)」であることを明記します。 - 計算エンジン:
set_engine("ranger")で、モデルの計算にrangerパッケージを使用するよう指定します。
rf_model <-
rand_forest() %>%
set_mode("classification") %>%
set_engine("ranger")
モデルの指定内容を確認
これもレシピと同様、この段階ではまだモデルの「設計図」を指定しただけで学習は行われていません。
print(rf_model)
Random Forest Model Specification (classification)
Computational engine: ranger
ワークフローの作成
ワークフローオブジェクト(rf_workflow)を出力すると、設定した前処理(Preprocessor)とモデル(Model)が正しく格納されていることが確認できます。
ここで、tidymodelsの中核となるworkflow()が登場します。
ワークフローは、これまでに作成した「前処理レシピ(penguins_recipe)」と「モデルの設計図(rf_model)」を一つのオブジェクトにまとめる(パイプライン化する)ための仕組みです。
add_recipe()とadd_model()を使って、それぞれをワークフローに組み込みます。これにより、前処理からモデル学習までの一連の流れを一体として扱えるようになり管理がしやすくなります。
rf_workflow <-
workflow() %>%
add_recipe(penguins_recipe) %>%
add_model(rf_model)
ワークフローの中身を確認
print(rf_workflow)
══ Workflow ════════════════════════════════════════════════════════════════════════════════════════════════════════════════════════════════════════════════════════════════════
Preprocessor: Recipe
Model: rand_forest()
── Preprocessor ────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────
3 Recipe Steps
• step_impute_knn()
• step_dummy()
• step_normalize()
── Model ───────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────────
Random Forest Model Specification (classification)
Computational engine: ranger
モデルの学習(Fit)
いよいよモデルの学習です。
fit()関数に、作成したワークフロー(rf_workflow)と訓練データ(penguins_train)を渡すことで、学習が実行されます。
fit()関数内部では、まず訓練データがレシピに従って前処理され、そのデータを使ってランダムフォレストモデルが学習されます。
set.seed(456)
rf_fit <- fit(rf_workflow, data = penguins_train)
ここでもset.seed()で乱数を固定し、ランダムフォレストの挙動を再現可能にしています。
モデルの評価(Predict & Evaluate)
モデルが学習できたので、今度はその性能を評価します。
ここで重要なのは学習に使用していない「テスト用データ(penguins_test)」を使うことです。これにより、モデルが未知のデータに対してどれくらいの予測精度を持つか(=汎化性能)を測ることができます。
predict()関数に、学習済みワークフロー(rf_fit)とテストデータを渡して予測結果を取得します。その後、bind_cols()を使って、予測結果(.pred_class列)と、テストデータの正解ラベル(species列)を横に並べたデータフレーム(test_results)を作成し、評価の準備をします。
test_results <- predict(rf_fit, new_data = penguins_test) %>%
bind_cols(penguins_test %>% select(species)) # 正解ラベルを横に結合
混同行列 (Confusion Matrix) で評価
分類モデルの評価で最もよく使われるのが混同行列(Confusion Matrix)です。これは、モデルの予測(Prediction)と実際の正解(Truth)をクロス集計した表です。
conf_mat()関数で簡単に作成できます。
conf_mat(test_results, truth = species, estimate = .pred_class) %>%
print()
Truth
Prediction Adelie Chinstrap Gentoo
Adelie 30 1 0
Chinstrap 1 13 0
Gentoo 0 0 25
対角線(左上から右下)の数値が「正しく予測できた件数」です。
今回の結果を見ると、Adelieと予測したものが1件ChinstrapであったりChinstrapと予測したものが1件Adelieであったり、といった小さな誤分類が2件ありますが、それ以外はすべて正しく分類できています。
Gentoo(ジェンツーペンギン)は25件すべて正解していますね。
正解率 (Accuracy) で評価
最後に、より直感的な指標である正解率(Accuracy)を見てみましょう。
これはすべての予測のうち正しく予測できた割合を示す指標です。accuracy()関数で計算できます。
accuracy(test_results, truth = species, estimate = .pred_class) %>%
print()
# A tibble: 1 × 3
.metric .estimator .estimate
<chr> <chr> <dbl>
1 accuracy multiclass 0.971
結果は 0.971、つまり97.1% という非常に高い正解率が得られました。
おわりに
今回は、tidymodelsを使ってデータ読み込みから前処理(レシピ)、モデル構築(ランダムフォレスト)、学習、評価までの一連のワークフローを体験しました。
recipe()で前処理を定義し、workflow()でモデルと組み合わせる流れは、Tidyで(整然としていて)管理しやすいですね。