インストール等
brew tap brewsci/bio
brew install mafft
brew install raxml-ng
conda install -c bioconda trimal
conda install -c bioconda modeltest-ng
Figtreeのインストール
Figtreeは系統樹ファイルの可視化と編集ができるソフトウェア
http://tree.bio.ed.ac.uk/software/figtree/
ちなみに
ETE toolkitでワークフローを簡略化しようと思ってやってみたけどinstall errorでete3を起動できない,python3.5以降だとだめっぽい,pipでもanacondaでもダメ
参照:https://qiita.com/aical/items/2231198d3d75e72bee39
まあ系統樹作るだけならそんなに手間じゃないのでいっか
配列を適当にとってくる
NCBIでrat, torafugu, medakaのsodium chanel voltage-gated alpha subunitを調べる
大体9-10個くらいの遺伝子がある,aaが違いそうなバリアントも含めてアミノ酸配列をとってくる
ついでに外群としてdrosphiaのsodium chanelをとってきた
前処理
mafftでアライメント
mafft --auto input.txt > align_res.aln
trimalでトリミング
trimal -in align_res.aln -out out_trimal.aln -htmlout out.html -automated1
モデルの選定
modeltest-ng -i out_trimal.aln -o test_res -T raxml -t ml -r 123413 -p 12 -d aa
☆オプション
-T 後解析で用いるsoftware(指定しなくても良い)
-t ml 最尤法
-r random seed
-p thread数
-d nt/aa
Model Score Weight
----------------------------------------------------------
BIC LG+G4 37887.7915 0.5968
AIC LG+I+G4 37506.8821 0.8959
AICc LG+I+G4 37516.8821 0.8392
LG+I+G4がいいのかな?
本当はRAxMLで自動でモデルを推定してtree作成するPROTGAMMAAUTOがあるはずなのだが,今回はmodelTest-NGで最適モデルを推定してみた
MEGA10でモデル推定してたときより圧倒的に速い,100配列くらいいれても数分で終わる,神
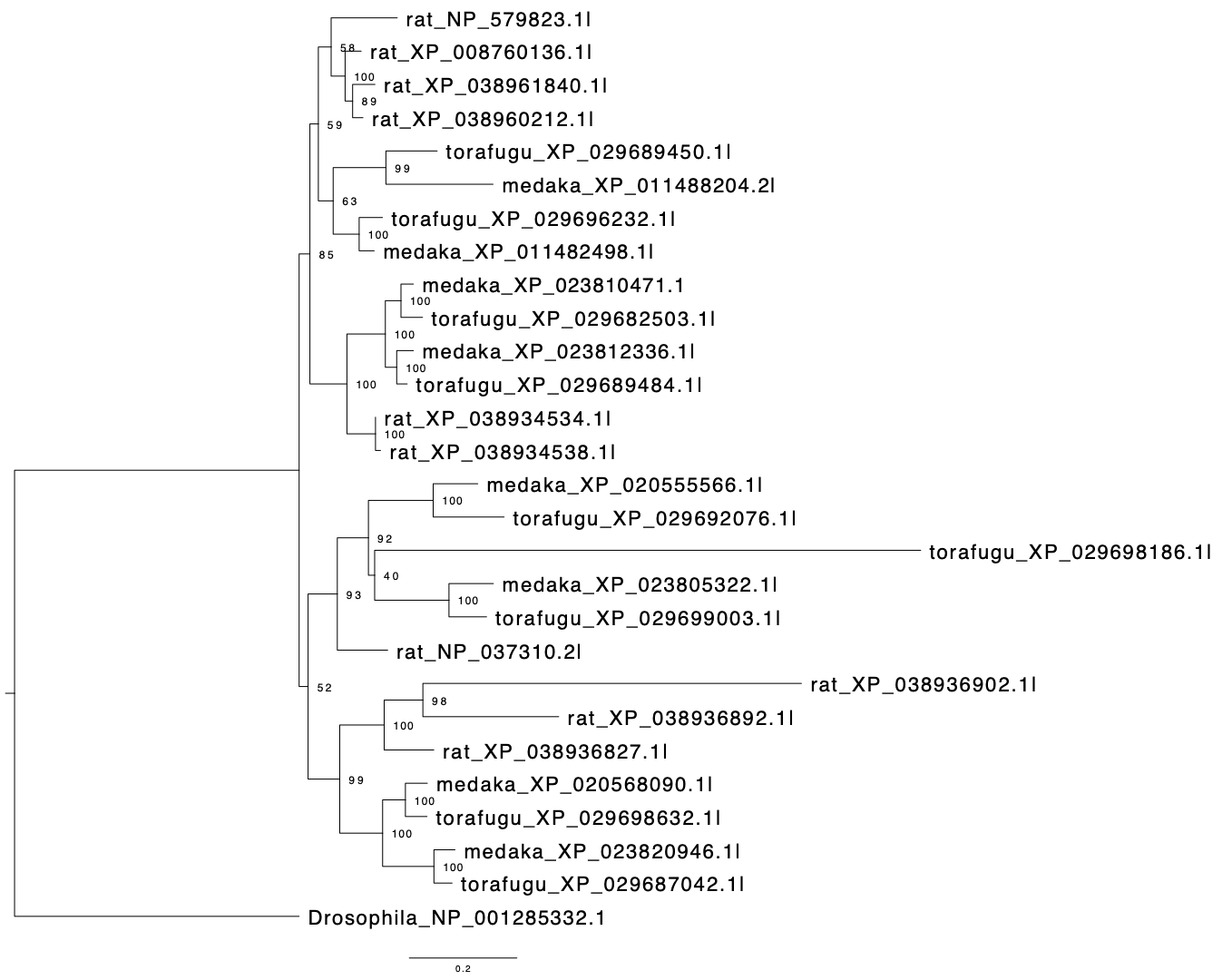
いよいよRAxMLで系統樹を作る
raxml-ng --msa out_trimal.aln --all --model LG+I+G4 --bs-trees 100 --threads 16
すぐ終わる,速い,神
なんかいっぱいファイルが出来た
.phylip.raxml.supportをFigtreeで開く,node labelsにチェックを入れてlabelを選択するとbootstrap値が表示された系統樹を得ることができる
色を変えたり,進化距離を表すスケールバーを表示したり,rootを変えたり,枝を回転したり,etcできる
まあ形だけ決めて,後はイラレなどのやり慣れた編集ツールで手直しするのがいいらしい(伝聞)