生存時間解析のPythonパッケージである lifelines を用いて、Kaplan-Meierによる生存関数の推定を行います。本記事は以下の記事の和訳になります。
Kaplan-Meierによる生存関数の推定
この例では、世界の政治リーダーの在任期間について解析していく。ここで、政治リーダーとは法治政権を統治する個人の在任期間と定義される。ここでの政治リーダーは選出された大統領などであり、選出されていない独裁者や君主などは含まれない。誕生イベントを「在職期間の始まり」、死亡イベントを「退任」とする。
生存関数を推定するには、次のように定義されるKaplan-Meier推定量を用いる。
$$\hat{S}(t)=\prod_{t_i \ < t} \frac{n_i-d_i}{n_i}$$
ここで$d_i$は時刻tにおける死亡イベント数、$n_i$は時刻tの直前に死亡する可能性のあるものの数である。
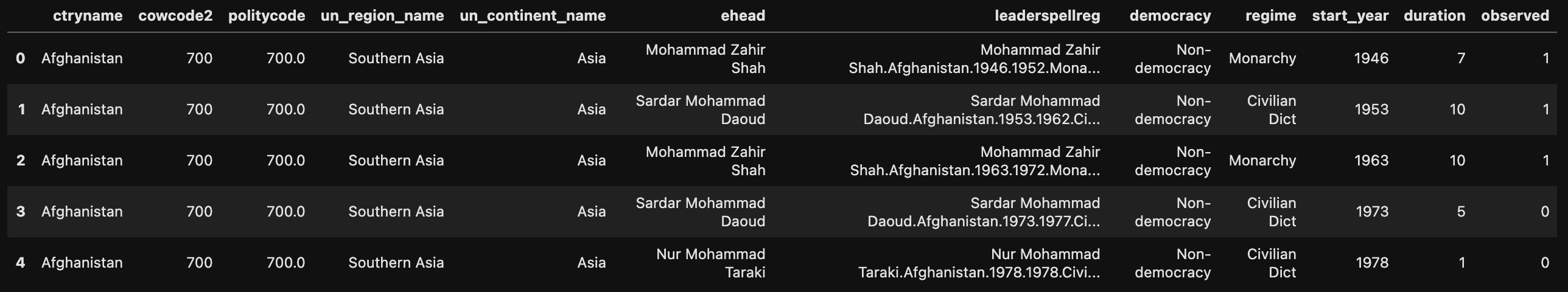
from lifelines.datasets import load_dd
data = load_dd()
data.head()
この演習では、 lifelines ライブラリに含まれる KaplanMeierFitter が必要になる。
from lifelines import KaplanMeierFitter
kmf = KaplanMeierFitter()
この推定では、「各リーダーの在職期間(=T)」と「退任が観測されたかどうか(=E)」の情報が必要である。モデルをデータに適合させるには fit() メソッドを使用する。
T = data["duration"]
E = data["observed"]
kmf.fit(T, event_observed=E)
fit() メソッドが呼ばれた後、KaplanMeierFitter は survival_function_ というプロパティを持つ。これはPandasのDataFrameであり、 plot メソッドを呼び出すことで生存関数をプロットできる。
from matplotlib import pyplot as plt
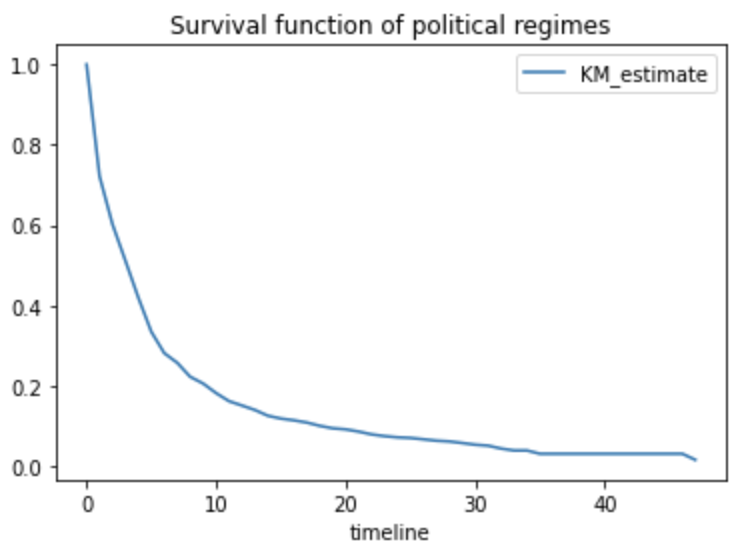
kmf.survival_function_.plot()
plt.title('Survival function of political regimes');
この結果をどう解釈したら良いだろうか?
x軸は時間経過t(年)を表し、y軸はt年後にそのリーダーがまだ在任している確率を表している。20年間在任しているリーダーはごくわずかであることが分かる。もちろん、この点推定の結果がどの程度不確かなものであるか、すなわち信頼区間を把握する必要がある。信頼区間は fit() 呼び出し時に計算され、confidence_interval_ プロパティに格納されている。
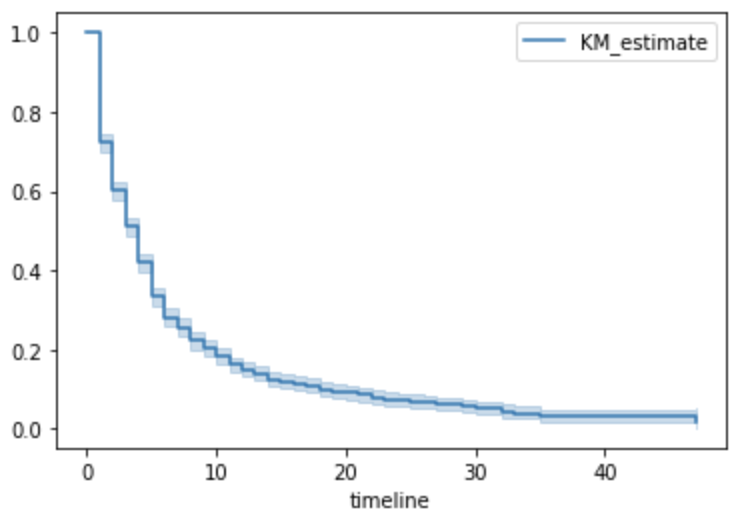
KM推定量とその信頼区間をプロットするには KaplanMeierFitter 自身の plot() メソッドを呼び出せば良い。
kmf.plot()
在任期間の中央値は median_survival_time_ プロパティに格納されている。
kmf.median_survival_time_
# 4.0
興味深いことに、中央値はわずか4年である。つまり、選出されたリーダーは4年以内に交代する可能性が50%ということである。この中央値の信頼区間を得るために、次の関数が用意されている。
from lifelines.utils import median_survival_times
median_ci = median_survival_times(kmf.confidence_interval_)
ログランク検定
次に、民主政権と非民主政権に分割して考えてみよう。
推定量自身またはfitterオブジェクトの plot メソッドを呼び出すことで axis オブジェクトを返し、さらなる推定に利用できる。ある期間内の確率を推定したいときは、引数 timeline を指定する。
import numpy as np
ax = plt.subplot(111)
t = np.linspace(0, 50, 51)
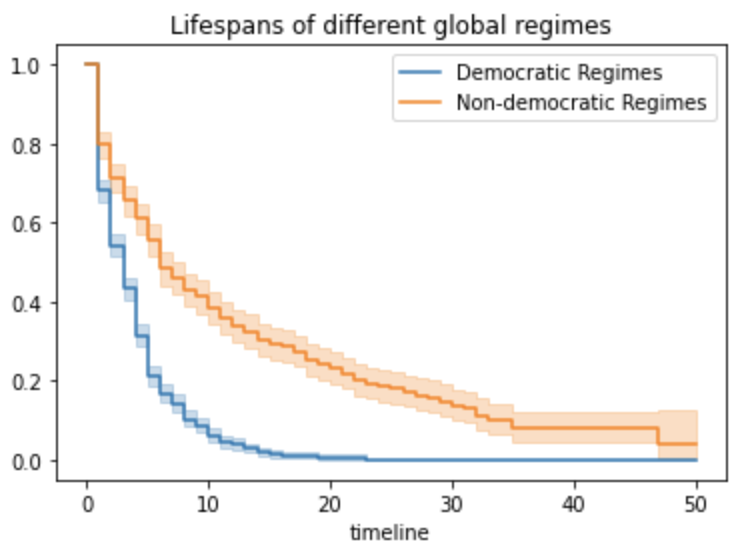
kmf.fit(T[dem], event_observed=E[dem], timeline=t, label="Democratic Regimes")
ax = kmf.plot(ax=ax)
kmf.fit(T[~dem], event_observed=E[~dem], timeline=t, label="Non-democratic Regimes")
ax = kmf.plot(ax=ax)
plt.title("Lifespans of different global regimes");
非民主政権の方がずっと在任期間が長いことには驚きである。民主政権には解任に向かうバイアスとして、「投票による解任」と「任期制限」が存在する(米国の場合は8年まで)
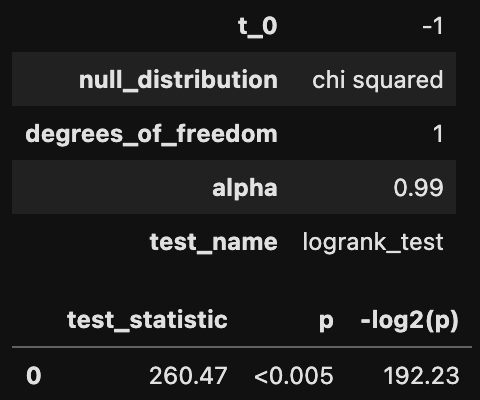
この例では両者の生存関数の違いは明らかではあるが、統計的検定を行ってみよう。二つの曲線がより似ていれば、もしくはデータが十分でなければ、統計的検定に関心を抱くはずである。この場合、lifelines は二つの生存関数を比較するための手続きを lifelines.statistics として含んでいる。lifelines.statistics.logrank_test() は二つのイベント系列生成器を比較する生存時間解析において一般的な統計的検定である。返り値が事前に求めた棄却域に含まれていれば、二つの生成器は異なるものであると結論づけられる。
from lifelines.statistics import logrank_test
results = logrank_test(T[dem], T[~dem], E[dem], E[~dem], alpha=.99)
results.print_summary()
生存関数の検定にはいくつかの手法が存在する。
詳細は次を参照のこと : Statistically compare two populations
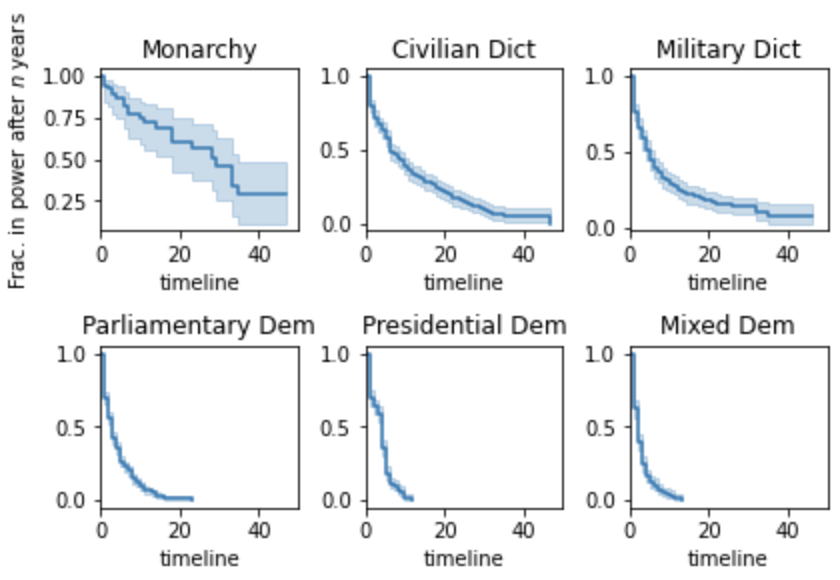
政権のタイプ毎に比較してみよう。
regime_types = data['regime'].unique()
for i, regime_type in enumerate(regime_types):
ax = plt.subplot(2, 3, i + 1)
ix = data['regime'] == regime_type
kmf.fit(T[ix], E[ix], label=regime_type)
kmf.plot(ax=ax, legend=False)
plt.title(regime_type)
plt.xlim(0, 50)
if i==0:
plt.ylabel('Frac. in power after $n$ years')
plt.tight_layout()
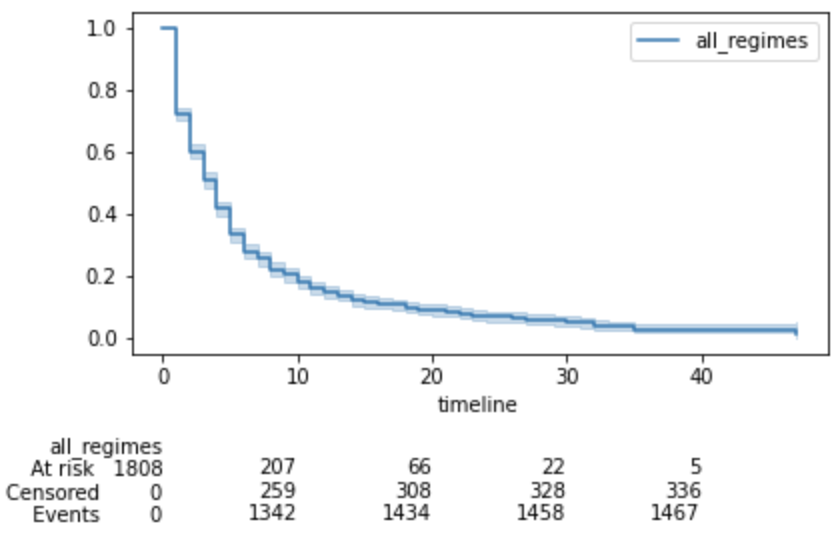
KMプロットを行う際のベストプラクティス
統計学者、医療関係者、その他のステークホルダーの最近の研究では、サマリテーブルと信頼区間の二つを、KMプロットの際に加えることを提案している。lifelines では信頼区間は自動的に追加されるが、引数 at_risk_counts をTrueに指定することで、サマリテーブルに追加することも可能である。
kmf = KaplanMeierFitter().fit(T, E, label="all_regimes")
kmf.plot(at_risk_counts=True)
plt.tight_layout()
Getting data into the right format
(割愛)