Wikipathwaysの白地図を有効活用したい
前回までの記事でリピドミクス用のpathway解析ツールを紹介しました。
前回の記事→https://qiita.com/otakaki/items/8e822bb60f93193852ed
そこで今回はWikipathwaysが提供している白地図に対して図の投影を行うことで、代謝pathway解析ツールとして使用する方法を紹介します。
事前準備
Cytoscapeのインストール(https://cytoscape.org/download.html)
R/Phython等の環境(適当な図を作成できればなんでもよい)
Cytoscape上でwikipathwaysの白地図を開いてみる
Cytoscapeを起動したらページ上部のAppタブからApp storeを開く
検索バーから"Wikipathways"と検索すると以下のページにたどり着ける。
僕はすでにインストール済みであるが、初めての場合インストールボタンが表示されるので、インストールする。
Networkタブの左上の検索バーからWikipathwaysのマークを選択し、WP5201を検索する。→Import as pathway
今回はWikipathways上でWP5201というIDで登録されているPhospholipid biosynthesisの白地図を使用する。
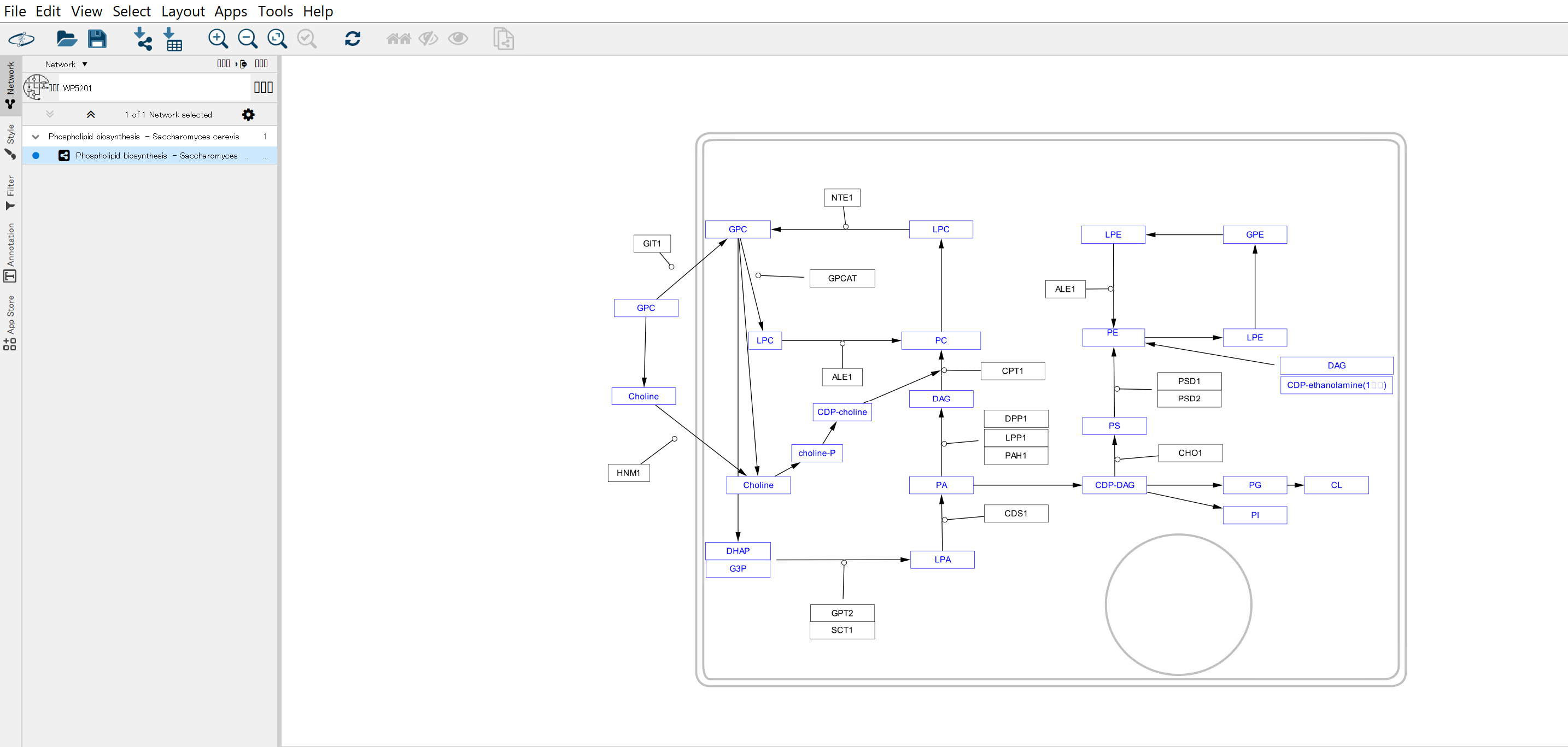
Node Tableにpathという列を追加する。
path列に以下のような文字を追加する。
file:/E:/oka/heatmap_01.svg
heatmap_01.svgはローカルに存在する画像ファイルであり、各ノードに対して載せたい図を指定する。
nodeのPropertiesからImage/Chart PositionのMap.を設定する。
Colum→path
Mapping Type→Passthogh Mapping
その後imageのサイズと位置を調整すると以下のようになります。
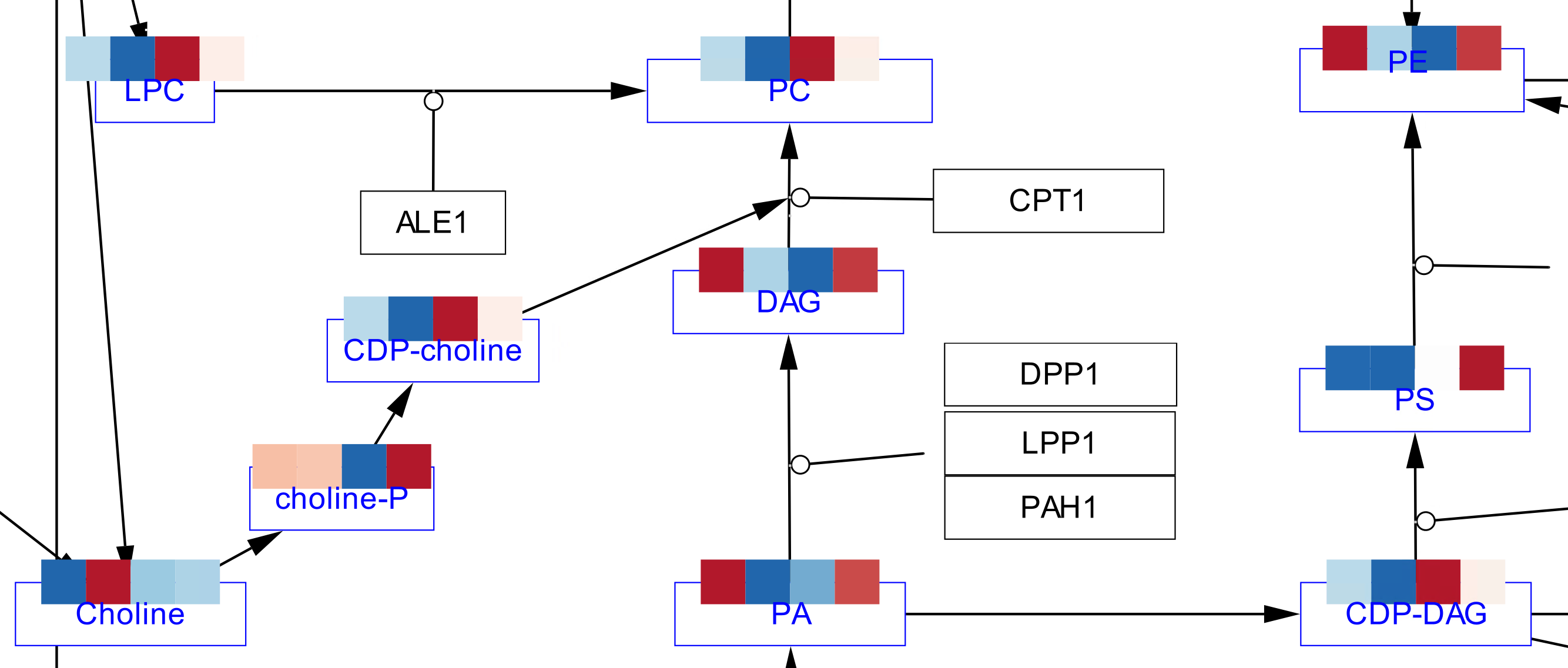
例えば4つのクラスの発現量をz-scoreのヒートマップとして表現するとしたらpathway解析っぽくなりそうですよね。
今回はヒートマップにしましたが、ノードのサイズを変更して、barplotやboxplotを投影してもいいと思います。
PS, 12/16日、文章ちゃんと書き直します。。時間が足りませんでした。。