リピドミクスの解析ツールを比較してみる
当研究室では基本的にメタボロミクスに関する研究を行っていますが、そのなかでも多くの学生が関わっているのが脂質を解析対象にしたリピドミクスです。一般的なリピドミクスのワークフローを示したのが以下の図になりますが、今回は定量化されたリピドミクスデータをどのように可視化するのか(図の7.3にあたります)という点にフォーカスしてみようと思います。
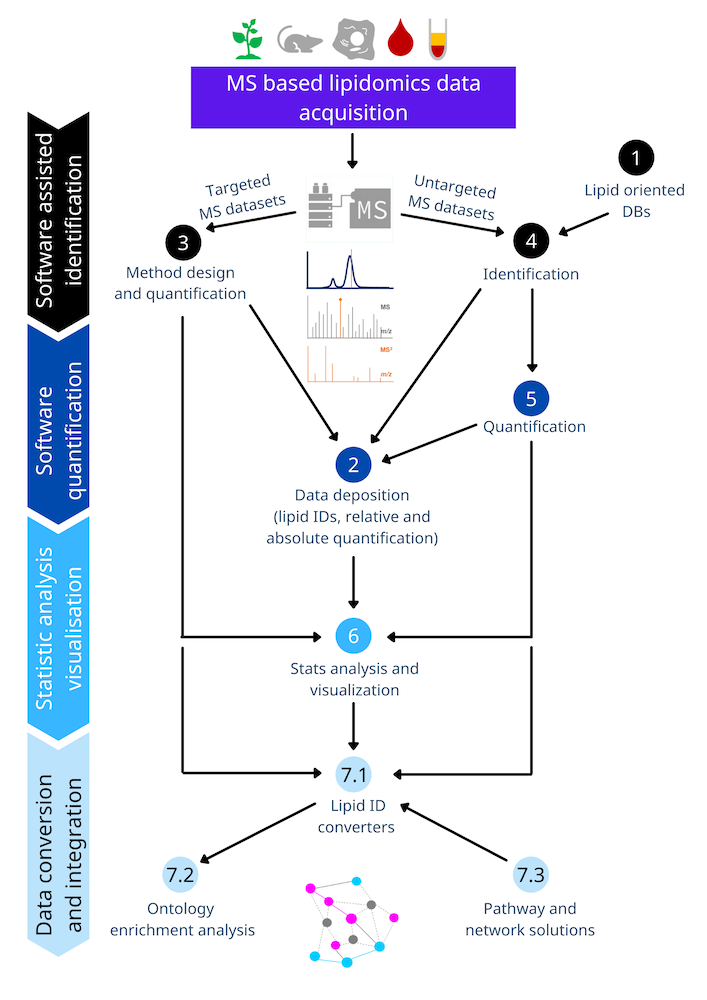
引用:Guiding the choice of informatics software and tools for lipidomics research applications, Nature Methods, 2022, DOI: 10.1038/s41592-022-01710-0
目次
メタボロミクスにおけるpathway解析とは?
メタボロミクス研究におけるpathway解析というのは分析で得られた代謝物データを代謝パスウェイ上に投影して解析する手法です。ポイントととしては個々の代謝物だけを比較するのではなく、代謝パスウェイ全体を俯瞰して差異を見ることで、代謝変動のポイントや実験的介入ポイントを探索することが目的です。
リピドミクス研究で使用できるpathway解析ツールは?
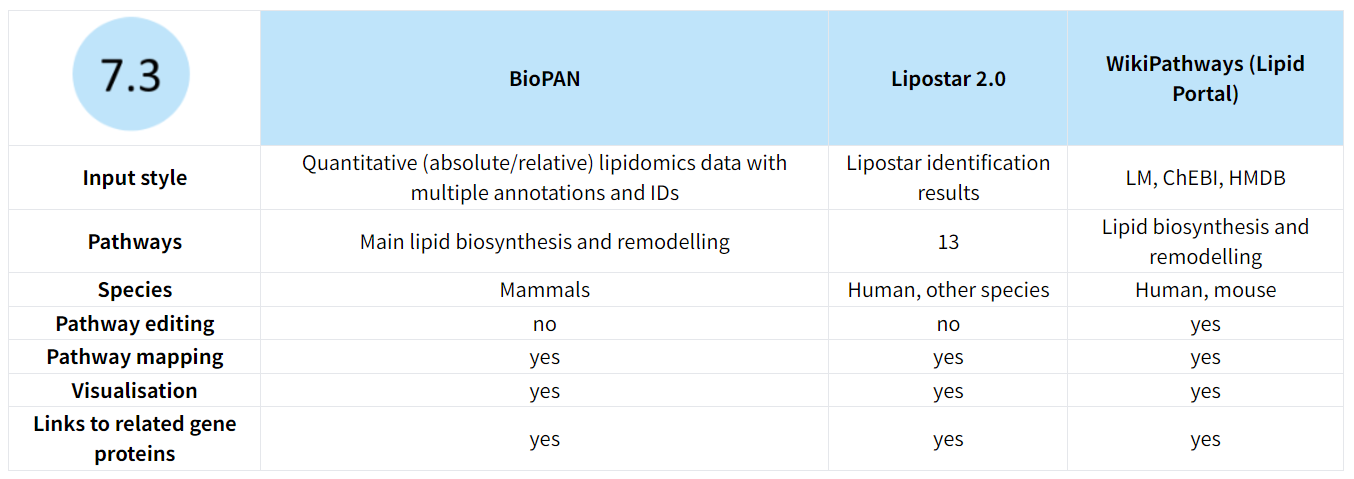
LIPIDMAPSが提供するLipidomics Tool Guideを参照すると以下の3つの解析ツールが紹介されています。
(LIPIDMAPSってなにと思った方はこちら→https://www.lipidmaps.org/)
特徴はこんな感じです。
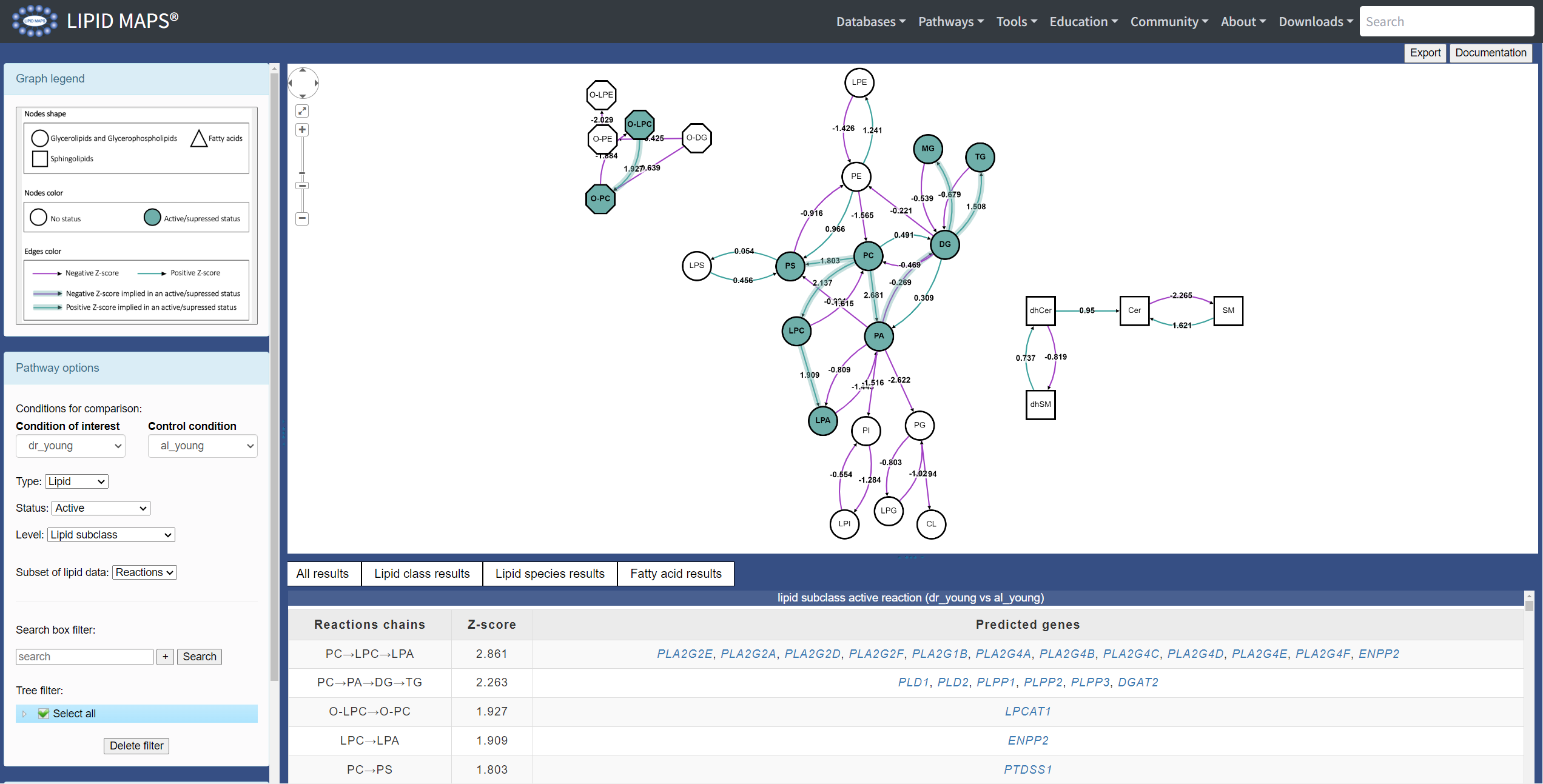
BioPAN
- 脂質の代謝経路をネットワークで表現
- 2つの条件サンプルを比較できる(コントロール群と対象群)
- 代謝変動の大きさをedge上のz-scoreで表現
- 代謝変動の大きいポイントに関して関連遺伝子のリストを表示(これ素敵)
- 脂質分子レベルでもネットワークを描画できる(ちょっとカオスすぎるけど。。)
Lipostar 2.0
- リピドミクスデータの生データ処理、定量からパスウェイ解析までをサポートしているらしい。
- ライセンスのリクエストが必要らしい(やってなかったので省略します)
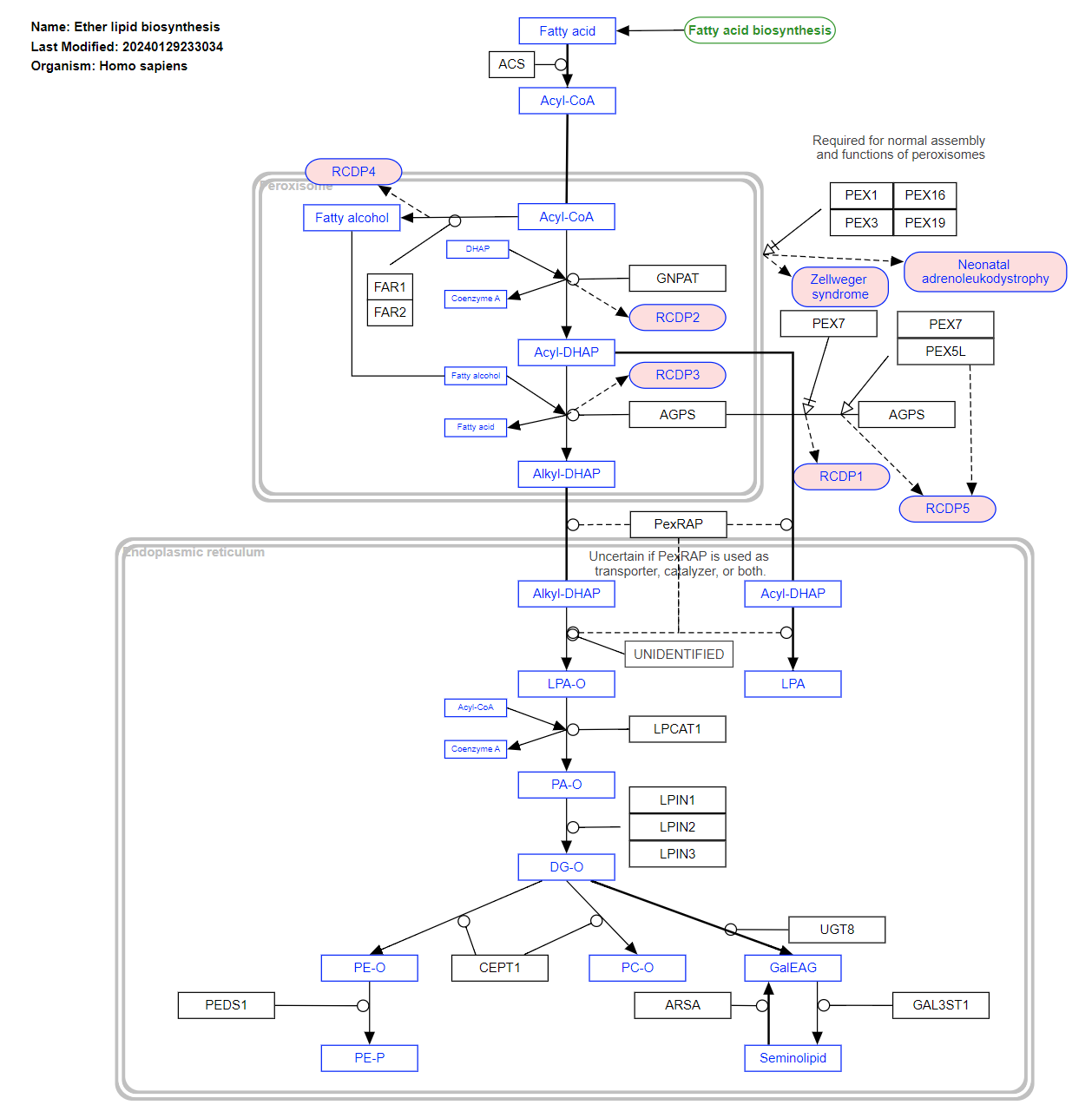
WikiPathways (Lipid portal)
- 生物学的パスウェイモデルの収集とキュレーションを行うデータベースプラットフォーム
- 基本的には代謝パスウェイの閲覧がメインの機能 (その分パスウェイの充実度はすごい)
- 定量値データを投影するのは結構大変そう
- Cytoscape上でこれらのpathwayは編集できて便利
引用: https://www.wikipathways.org/pathways/WP5275.html
ツールの比較
- BioPANは操作が簡単。とりあえず解析してみたら何かしらの結果が返ってきそう。
- でも2つのサンプル群に解析が限定されているのはのちのち不便になると思う。
- Wikipathwayはサポートしている代謝パスウェイが充実しているので、見ていて楽しい。
- でもWikipathwayの図に定量値を投影するのは難しそう。
それぞれのツールにはそれぞれの使いどころが決まっていそうですね( ̄▽ ̄)
個人的にはwikipathwayの方が好きです。
ということで次回はwikipathwayの図に定量値を投影して解析する手法を紹介します。
次の記事からはスクリプトを書けたらいいな