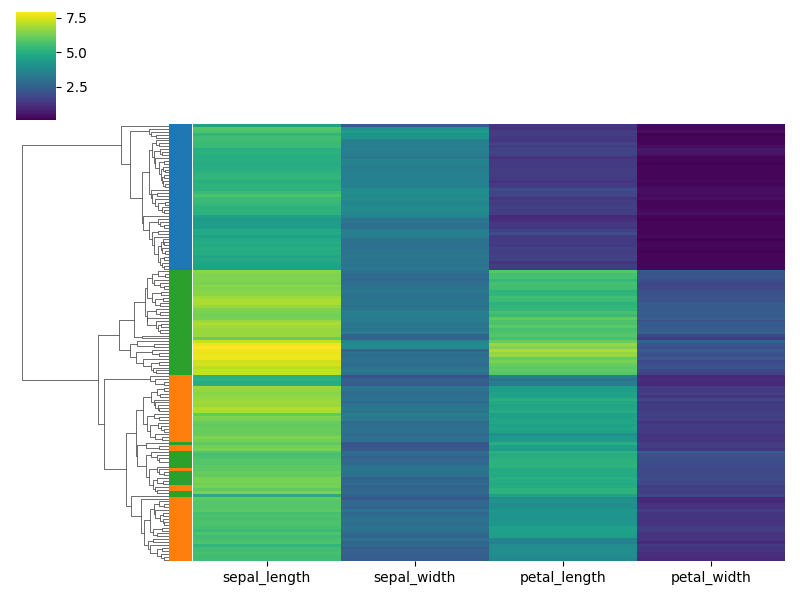
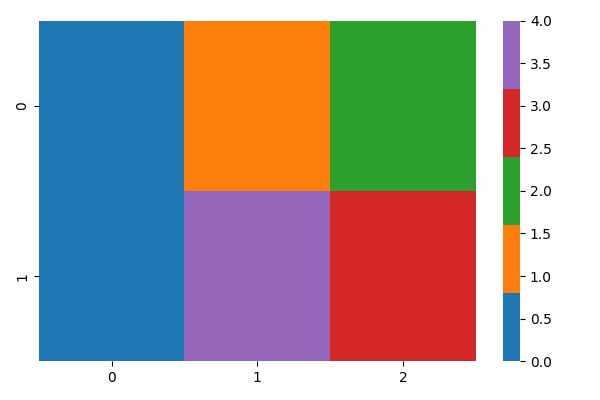
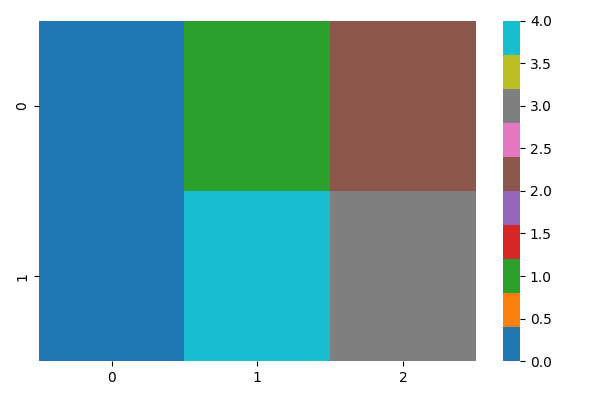Pythonのseabornパッケージのclustermap関数には、row_colors/col_colorsという便利なオプションがあります。以下の図のように、ヒートマップの左や上に任意のカテゴリカル変数の値を色で示すことが出来ます。
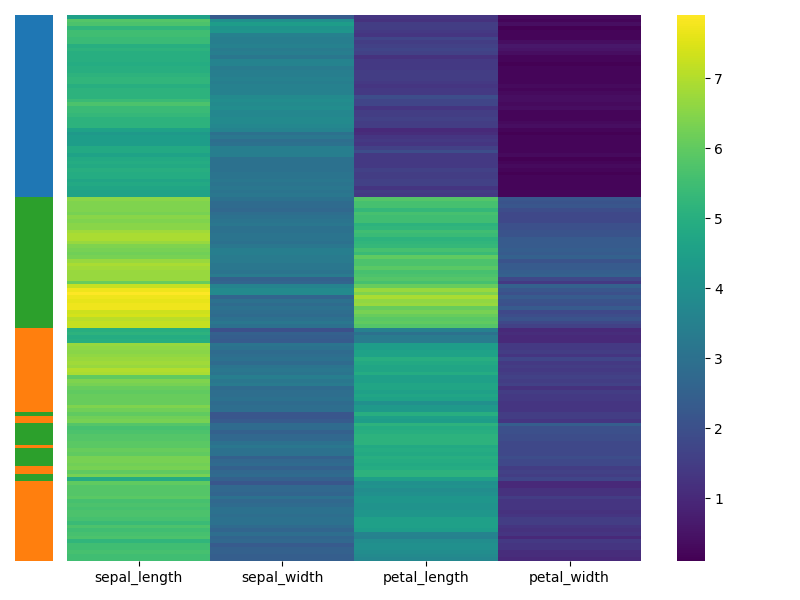
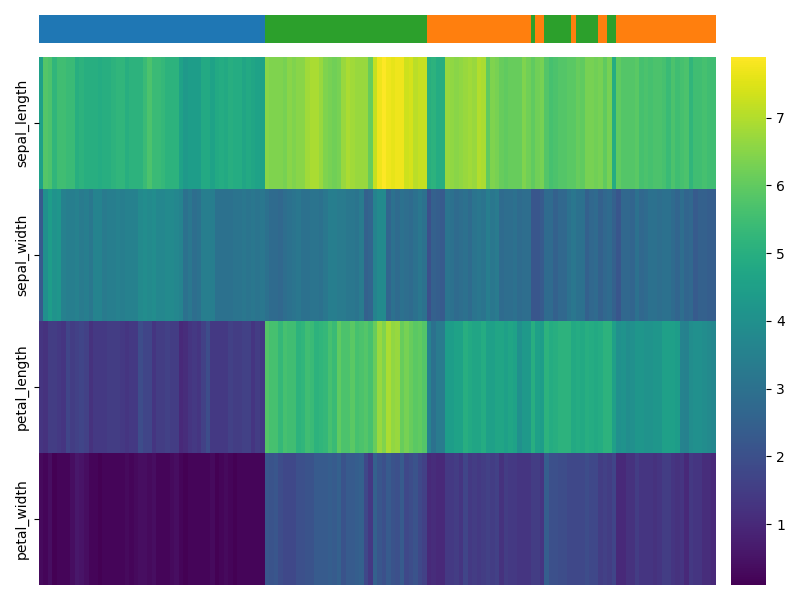
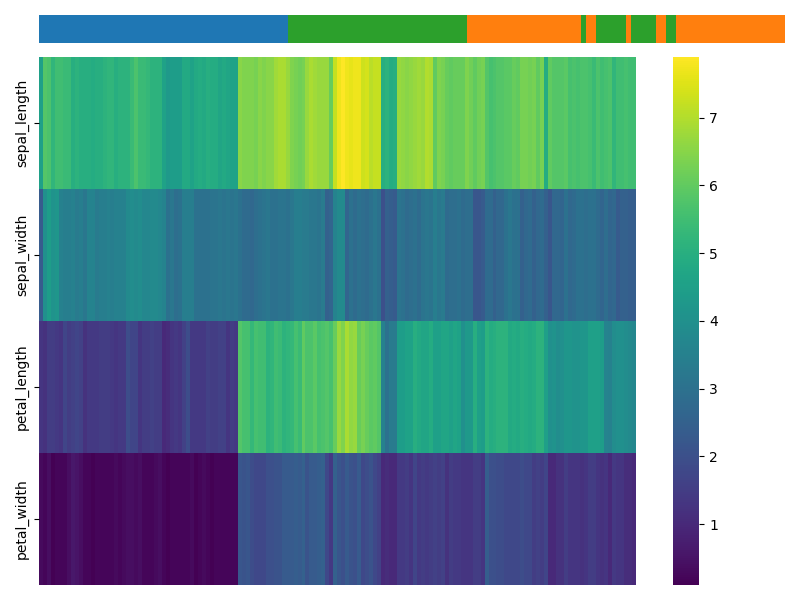
しかし、clustermap関数の出力はfigレベルでaxレベルではないため、他の図と組み合わせるのが難しいという問題があります。そこで、本記事では、似たような図をseabornのheatmap関数を使って再現する方法を説明します。heatmap関数の出力はaxレベルです。以下に例を示します。上の図は行の色、下の図は列の色を表示した場合です。
基本的な考え方
状況に応じてソースコードの書き方を色々と変える必要があるため、残念ながら単にコピペすれば済む話ではありません。そこで、先に基本的な考え方を説明します。具体的なコードはその後に載せます。
- 行や列の色は、axを分けて 整数値のヒートマップ で表す
- ヒートマップは一次元配列には適用できないので、二次元配列に変換 する
- そのままだと大き過ぎるので、axの比率 を調節して細長くする
- 余計な軸やカラーバーは消す
- 列の色の場合、右端がズレるので、メインデータのcaxを明示的に指定 する
整数値のヒートマップ
整数値の場合、普通にcmap='tab10' とかだとダメです。以下のように色が飛び飛びに使われるためです。
sns.heatmap([[0,1,2],[0,4,3]], cmap='tab10')
どうすれば良いかというと、予め色の数を指定したcmapを作成すればOKです。以下に例を示します。
cmap = sns.color_palette('tab10', n_colors=5)
sns.heatmap([[0,1,2],[0,4,3]], cmap=cmap)
カテゴリカル変数に使用可能なカラーマップの一覧は以下に載っています。
この他、seabornのみで使えるhuslというカラーマップもあります。
sns.color_palette('husl', n_colors=10)
一次元配列を二次元配列にする
$n$個の要素を持つ一次元配列 (ベクトル) を、$n\times 1$または$1\times n$の二次元配列 (行列) に変換してから、heatmap関数にわたします。やり方は色々ありますが、例えばreshapeで出来ます。
x = np.array([0,1,2,3])
y1 = x.reshape(1,-1) # => 1 x 4 の行列
y2 = x.reshape(-1,1) # => 4 x 1 の行列
axの比率の調節
普通にsubplotsで複数のaxを生成すると、同じサイズのものが出てきます。
fig, axes = plt.subplots(figsize=(6,4), nrows=2, ncols=2)
縦横の比率は、gridspec_kwオプションの中に辞書型でheight_ratiosやwidth_ratiosを指定すると変えられます。以下は横方向を1:2に、縦方向を1:3にしたものです。比率の合計は1でなくて構いません。ややこしいですが、gridspec_kwは末尾にsなし、height_ratiosとwidth_ratiosは末尾にsありです。
fig, axes = plt.subplots(figsize=(6,4), nrows=2, ncols=2,
gridspec_kw=dict(width_ratios=(1,2),
height_ratios=(1,3)))
余計な軸やカラーバーは消す
行や列の色をヒートマップで表す場合、軸や目盛りは不要なので消します。
ax.axis('off')
ややこしいんですが、axは、実はaxisの略ではなくてaxesの略で、axes自体は残しつつ、その中のaxisのみ消すという意味です。一方で、慣習的にaxと書いたら単数形、axesと書いたら複数形を意味します。
カラーバーは、heatmap関数を使うと既定で表示されてしまうので、非表示にしておきます。あくまで行や列の色に関するカラーバーの話で、メインデータのカラーバーはまた別です。
sns.heatmap(..., cbar=False)
右端のズレを直す
普通、heatmap関数は自動でカラーバーの位置を調節してくれるので、明示的にaxを分けなくて良いのですが、列の色を表示する場合、素直に実装すると以下のようにズレてしまいます。
上に細長く表示されている列の色が横幅を100%使おうとするのに対し、その下のメインのヒートマップの部分は右にカラーバー用のスペースを確保するため、ズレています。
解決方法は2つあって、カラーバーを水平方向にするか、全体をあらかじめ$2\times 2$のaxesに分割しておくかです。
カラーバーを水平方向にするには、cbar_kwsオプションに辞書形式でorientation='horizontal'を指定するとできます。
sns.heatmap(..., cbar_kws = dict(orientation='horizontal'))
全体を$2\times 2$のaxesに分割する方は、普通にsubplotsでncols=2, nrows=2とするだけです。
コード例1: clustermapを使う場合
先にclustermapの例をお示しします。便利とはいっても、色のリストを用意する部分は結構ややこしいです。最終的にRGBの3つの値の組が列の数だけ並んだ配列を作ってrow_colorsにわたすことを目指してコードを書くと良いと思います。
# 必要なパッケージをインポートする
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
# サンプルデータirisを読み込む
df = sns.load_dataset('iris')
# 文字列型のspecies列を分離する
sr = df.pop('species')
# species列を整数値に変換する
row_codes = sr.factorize()[0]
# 行の色のカラーマップを用意する
row_cmap = sns.color_palette('tab10', n_colors=3)
# 行の色のリストを用意する
row_colors = [row_cmap[i] for i in row_codes]
# clustermap関数で描画する
g = sns.clustermap(data = df, # メインデータ
cmap = 'viridis', # メインデータのカラーマップ
row_colors = row_colors, # 行の色のリスト
figsize = (8,6), # 描画サイズ
yticklabels = False, # y軸の目盛りを非表示にする
col_cluster = False # 列方向のクラスタリングを中止
)
# 画像ファイルに出力
g.fig.savefig('tmp.png')
コード例2: 行の色を表示する場合
次にheatmap関数を使って行の色を表示する場合です。途中まではclustermapと同様ですが、冒頭でlinkage, leaves_listを追加インポートしているので注意して下さい。クラスタリングの際に行の順番が変わるため、メインデータと色番号の配列の両方を並び替えています。樹形図は省いていますが、もちろん入れることは可能です。
# 必要なパッケージをインポートする
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
from scipy.cluster.hierarchy import linkage, leaves_list
# サンプルデータirisを読み込む
df = sns.load_dataset('iris')
# 文字列型のspecies列を分離する
sr = df.pop('species')
# species列を整数値に変換する (以下、色番号と呼ぶ)
row_codes = sr.factorize()[0]
# 行の色のカラーマップを用意する
row_cmap = sns.color_palette('tab10', n_colors=3)
# figとaxesを用意する
fig, axes = plt.subplots(figsize = (8,6), # 描画サイズ
ncols = 2, # 列の数 (行の色用、メインデータ用)
gridspec_kw = dict(width_ratios=(0.05, 0.95)) # 横方向の比率
)
# 行方向のクラスタリングを実行
Z = linkage(df, method='average', metric='euclidean')
# クラスタリングの結果に基づく並び順を取得する
leaves = leaves_list(Z)
# メインデータと行の色番号の配列を並び替える
df_sorted = df.iloc[leaves]
row_codes_sorted = row_codes[leaves]
# 行の色番号の配列を縦一列の行列にする
row_codes_matrix = row_codes_sorted.reshape(-1,1)
##### 行の色の描画 #####
# 左のaxを取得
ax = axes[0]
# heatmap関数で行の色を描画する
sns.heatmap(data = row_codes_matrix, # 行の色番号の行列
cmap = row_cmap, # 行の色のカラーマップ
ax = ax, # 描画先のax
cbar = False # (行の色に対する) カラーバーを非表示にする
)
# 軸を非表示にする
ax.axis('off')
##### メインデータの描画 #####
# 右のaxを取得
ax = axes[1]
# heatmap関数でメインデータを描画する
sns.heatmap(data = df_sorted, # メインデータ
cmap = 'viridis', # メインデータのカラーマップ
ax = ax, # 描画先のax
yticklabels = False # y軸の目盛りを非表示にする
)
# 余白を調節
fig.tight_layout(w_pad=1)
# 画像ファイルに出力
fig.savefig('tmp.png')
コード例3: 列の色を表示する場合
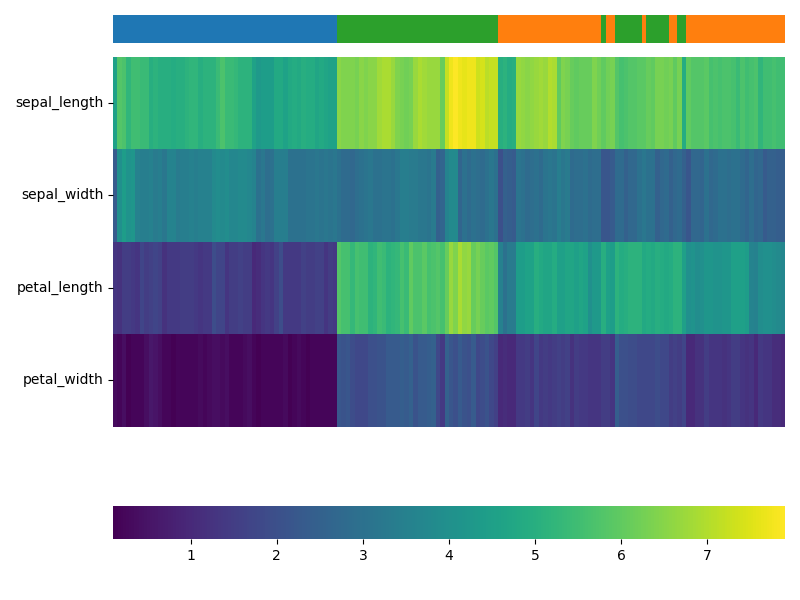
最後にheatmap関数で行の色を表示する場合です。簡単のため、irisデータを転置して使っています。行の色の場合と完全に対称ではなく、$2\times 2$のaxesに分割することに注意して下さい。
# 必要なパッケージをインポートする
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
from scipy.cluster.hierarchy import linkage, leaves_list
# サンプルデータirisを読み込む
df = sns.load_dataset('iris')
# 文字列型のspecies列を分離する
sr = df.pop('species')
# データフレームを転置する
df = df.T
# species列を整数値に変換する (以下、色番号と呼ぶ)
col_codes = sr.factorize()[0]
# 列の色のカラーマップを用意する
col_cmap = sns.color_palette('tab10', n_colors=3)
# figとaxesを用意する
# 注意: 行方向と列方向をそれぞれ2分割する
fig, axes = plt.subplots(figsize = (8,6), # 描画サイズ
nrows = 2, # 行の数 (列の色用、メインデータ&カラーバー用)
ncols = 2, # 列の数 (列の色&メインデータ用、カラーバー用)
gridspec_kw = dict(height_ratios=(0.05, 0.95), # 縦方向の比率
width_ratios=(0.95, 0.05)) # 横方向の比率
)
# 列方向のクラスタリングを実行 (注、この例では本来の行方向に相当)
Z = linkage(df.T, method='average', metric='euclidean')
# クラスタリングの結果に基づく並び順を取得する
leaves = leaves_list(Z)
# メインデータと列の色番号の配列を並び替える
df_sorted = df.iloc[:,leaves]
col_codes_sorted = col_codes[leaves]
# 列の色番号の配列を横一列の行列にする
col_codes_matrix = col_codes_sorted.reshape(1,-1)
##### 列の色の描画 #####
# 左上のaxを取得
ax = axes[0,0]
# heatmap関数で列の色を描画する
sns.heatmap(data = col_codes_matrix, # 列の色番号の行列
cmap = col_cmap, # 列の色のカラーマップ
ax = ax, # 描画先のax
cbar = False # (列の色に対する) カラーバーを非表示にする
)
# 軸を非表示にする
ax.axis('off')
##### メインデータの描画 #####
# 左下のaxを取得
ax = axes[1,0]
# 右下のaxも取得 (カラーバー用)
cax = axes[1,1]
# heatmap関数でメインデータを描画する
sns.heatmap(data = df_sorted, # メインデータ
cmap = 'viridis', # メインデータのカラーマップ
ax = ax, # 描画先のax
xticklabels = False, # x軸の目盛りを非表示にする,
cbar_ax = cax # カラーバーの表示先をcaxに指定
)
# 右上のaxは使わないので軸を非表示する
axes[0,1].axis('off')
# 余白を調節
fig.tight_layout(h_pad=1)
# 画像ファイルに出力
fig.savefig('tmp.png')
応用
様々なバリエーションが考えられます。行と列を両方ともクラスタリングしたり、行と列の色を両方表示したり、樹形図を表示したり、行の色を複数列にしたり、列の色を複数行にしたり、などです。なので、前述したとおり、コードをコピペしてすぐ使える感じではありません。各自で意味を理解した上でカスタマイズして使って下さい。