はじめに
分子は結合軸周りの回転によってさまざまな3次元構造をとる。Pythonで分子の3次元構造を生成する方法を紹介する。ここで生成した3次元構造はGaussianなどの量子化学計算の良い初期構造になりうる。
手順
①3次元構造を生成
②分子力学法によって最適化
今回のプログラムではこれらを一連の動作として実行
環境
- python 3.7.13
- rdkit 2022.3.4
- py3dmol 1.8.1
- pandas 1.2.5
参考サイト
3次元構造の生成
3次元構造生成には
- ディスタンス・ジオメトリー(DG)法
- KDG
- ETDG
- ETKDG
およびその改良法があるが、基本的にはETKDGを使えばよい。
DG法に対して、
- ベンゼン環は平面
- アルキンは直線
といった一般的な化学常識を加えたのがKDGである。またケンブリッジ結晶構造データベースの実験データから得た二面角の分布傾向(experimental torsion angle preferences)を考慮したものがETDGであり、それらの両方を加えたのがETKDGである。
#ライブラリーのインポート
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem
from rdkit.Chem.Draw import IPythonConsole
import py3Dmol
import pandas as pd
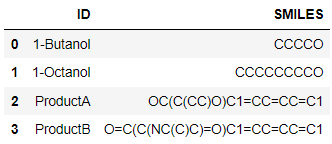
データの読み込み。ここでは例としてSMILESが分かっている4つの分子が記載されたcsvファイルを読み込みます。

# データの取得
df = pd.read_csv("sample_products.csv")
df
各化合物のSMILESをmol形式に変換しておきます。また水素原子を付加した状態のmol形式の取得しておきます。RDKitの公式にも記述がありますが「良い3Dコンフォメーションを得るには、ほとんどの場合、まず初めに水素原子を分子に付加すること上手くいきます。
# データフレームの化合物名を定義(のちのファイル生成で使う)
labels = df["ID"]
# 各化合物のmol(水素付加なし)を取得
mols = [Chem.MolFromSmiles(mol) for mol in df["SMILES"]]
# 各化合物のmol(水素付加済み)を取得
molHs = [ Chem.AddHs(Chem.MolFromSmiles(mol)) for mol in df["SMILES"]]
コンフォーマー生成のパラメータを定義しておきます。AllChem.EmbedMultipleConfsの引数として代表的なものは以下です。
- どの分子の3次元構造を生成するか(mol)
- いくつのコンフォマーを生成するか(numConfs)
- どの程度類似した構造を排除するか(pruneRmsThresh)
RMSの値を大きくすると得られるコンフォーマーの数は減少します。
# コンフォーマー生成のパラメータの定義
confs = 1000
rms = 0.5
フォースフィールド(力場)をかけて最適化する
以下のコードでは
- 1000個のコンフォマーを発生
- 各コンフォマーをUFFまたはMMFFで構造最適化(UFFもしくはMMFFの力場をかける)
- 各コンフォマーのエネルギーを計算
- 最安定構造から順番にソートして(相対エネルギー,コンフォマーID)のタプルのリストを取得
という作業を行っています。
# コンフォーマーの生成とフォースフィールド法で構造最適化
def opt_sp_mm(m_h, confs, rms, ff):
# 1. 1000個のコンフォマーを発生(ETKDG法)
cids = AllChem.EmbedMultipleConfs(m_h, numConfs=confs, randomSeed=1234, pruneRmsThresh=rms, numThreads=0)
# 2,3. 各コンフォマーを最適化し,エネルギー計算
energy = []
if ff == 'uff':
for cid in cids:
uff = AllChem.UFFGetMoleculeForceField(m_h, confId=cid)
uff.Minimize()
energy.append((uff.CalcEnergy(), cid))
if ff == 'mmff':
prop = AllChem.MMFFGetMoleculeProperties(m_h)
for cid in cids:
mmff = AllChem.MMFFGetMoleculeForceField(m_h, prop, confId=cid)
mmff.Minimize()
energy.append((mmff.CalcEnergy(), cid))
# 4. エネルギーをソートし,相対エネルギーとIDをリストに格納
energy.sort()
return [(i-energy[0][0],j) for i,j in energy]
まずはuffで最適化してエネルギー計算を実行し、最安定構造から順番に5つのコンフォーメーションを抽出します。この後Gaussianで量子化学計算を行うことを想定して、Gaussianの計算実行ファイルを作るためのテキストを準備します。
# uffとmmffで各化合物の最適構造を求めていく
uff_gaussian_filenames = []
# 最適化した構造の上位何件を取得するか設定する
pickup = 5
for i in range(len(labels)):
# uff法での構造最適化
uff_e = opt_sp_mm(molHs[i], confs, rms, 'uff')
# uffで最適化した構造のID上位を取得する
uff_confIds = [m for l,m in uff_e[:pickup]]
# 繰り返し回数の定義
num_confIds = len(uff_confIds)
if num_confIds<pickup:
iter = num_confIds
else:
iter = pickup
# 上位をmolファイルで保存していく
for j in range(iter):
uff_filename = f"{labels[i]}_uff_"+str(j)+".mol"
uff_gaussian_filename = f"g16 {labels[i]}_uff_"+str(j)+".gjf"
uff_gaussian_filenames.append(uff_gaussian_filename)
Chem.MolToMolFile(molHs[i], confId=uff_confIds[j], filename=uff_filename)
Gaussianの計算実行ファイルを作るためのテキストを作成します。
# Gaussian計算実行時のテキストを作成
txt = ["#!/bin/bash"]
txt.extend(uff_gaussian_filenames)
# 各要素に改行コードを付加
obj = map(lambda x: x + "\n", txt)
with open("uff_gaussian.txt", "w", encoding="utf-8") as f:
f.writelines(obj)
次にmmffでも同様の手順でコンフォーメーションを抽出していきます。
# uffとmmffで各化合物の最適構造を求めていく
mmff_gaussian_filenames = []
# 最適化した構造の上位何件を取得するか設定する(DC-02がconfs = 1000、rms = 0.5で3件しかヒットしない)
pickup = 5
for i in range(len(labels)):
# mmff法での構造最適化
mmff_e = opt_sp_mm(molHs[i], confs, rms, 'mmff')
# muffで最適化した構造のID上位を取得する
mmff_confIds = [m for l,m in mmff_e[:pickup]]
# 繰り返し回数の定義
num_confIds = len(mmff_confIds)
if num_confIds<pickup:
iter = num_confIds
else:
iter = pickup
# 上位をmolファイルで保存していく
for j in range(iter):
mmff_filename = f"{labels[i]}_mmff_"+str(j)+".mol"
mmff_gaussian_filename = f"g16 {labels[i]}_mmff_"+str(j)+".gjf"
mmff_gaussian_filenames.append(mmff_gaussian_filename)
Chem.MolToMolFile(molHs[i], confId=mmff_confIds[j], filename=mmff_filename)
# Gaussian計算実行時のテキストを作成
txt = ["#!/bin/bash"]
txt.extend(mmff_gaussian_filenames)
# 各要素に改行コードを付加
obj = map(lambda x: x + "\n", txt)
with open("mmff_gaussian.txt", "w", encoding="utf-8") as f:
f.writelines(obj)
個人的な感覚としてはuffとmmffであまり大きな差異はない印象。ただ、力場をかけた方が何もしない(ETKDG法で求めた)構造よりも量子化学計算後の最適化エネルギーがより安定な気がする。