前準備
モジュールのインストール
conda install -c conda-forge rdkit
pip install py3Dmol
分子の準備
適当な分子を準備する。
# 分子の準備
mol = Chem.MolFromSmiles('CN[C@H](C)[C@H](O)c1ccccc1')
# もしくは,SDFファイルの読み込みの際に,removeHs=Falseとすることで、水素原子を保持したまま分子の読み込みが可能
# suppl = Chem.SDMolSupplier('XXXX.sdf', removeHs=False)
# mols = [mol for mol in suppl if mol is not None]
最適化前の構造を確認
とりあえず、未処理の分子の3D構造を確認してみる。
# Jupyter notebook上で描画してみる
IPythonConsole.drawMol3D(mol)
# MOLファイルを出力して、座標から構造を確認することも可能
mb = Chem.MolToMolBlock(mol)
with open('mol_2D.mol', 'w') as f:
f.write(mb)
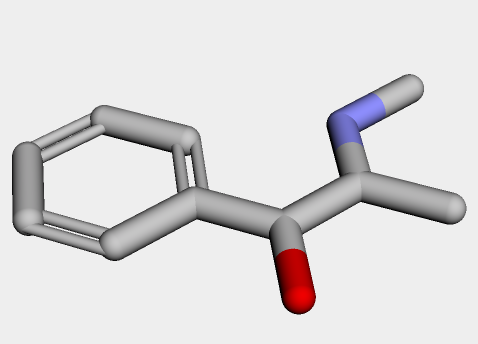
sp3炭素も全て平面に押し込まれてしまっている。
(実際は、マウスで構造をぐりぐり動かして確認することが可能)
水素の付加と三次元構造の最適化
水素の付加について
通常、RDkitに格納された分子は水素原子が暗に扱われている。
3次元の帰化構造を生成したり、最適化する場合には、良い3Dコンフォメーションを得るには、水素原子を分子に付加することが必要
三次元構造の最適化について
Molオブジェクトの3次元構造の最適化は、AllChem.EmbedMoleculeで行うことが可能。
RDkitでは以下の4つの方法が実装されており、デフォルトはETKDG法になっているとのこと。(普段使う分には、ETKDGの使用で問題ないらしい参考)
- ディスタンス・ジオメトリー(DG)法:Stochasticな立体構造の生成方法としてはかなり精度がよい
- KDG:DG法に一般的な化学常識(ベンゼン環は平面、アルキンは直線)を加えた手法
- ETDG:ケンブリッジ結晶構造データベースの実験データから得た二面角の分布傾向を考慮した手法
- ETKDG:KDGとETDGの両方を合わせた手法
- ETKDGv2
# 水素の付加
molH = Chem.AddHs(mol)
# ETKDGによる構造最適化
p = AllChem.ETKDG()
AllChem.EmbedMolecule(molH, p)
# Jupyter notebook上で描画してみる
IPythonConsole.drawMol3D(molH)
# MOLファイルを出力して、座標から構造を確認。
mb = Chem.MolToMolBlock(molH)
with open('mol_3D.mol', 'w') as f:
f.write(mb)
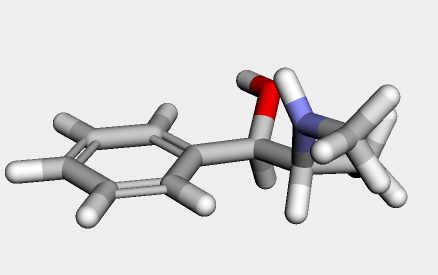
画像だとわかりにくいが、水素が付加され立体構造も妥当なものになっている。