はじめに
マテリアルズインフォマティクスで化合物の構造を分析していく際によく用いられるフィンガープリントについて深堀していきます。今回は__Morganフィンガープリントの生成ルール__(radiusに着目してどのようにフィンガープリントが生成されるのか)についてご紹介していきます。
ここに至るまでRDKitで表示した化合物の各原子に番号を表示することができず苦戦していました。解決方法に関しては、こちらの記事にまとめましたので、同様の問題で悩んでいる方は参考にして頂ければ幸いです。
※マテリアルズインフォマティクス関係の内容を他にも投稿していますので、よろしければこちらの一覧から他の投稿も見て頂けますと幸いです。
フィンガープリントとは?
フィンガープリント(fingerprint)とは日本語で「指紋」を意味します。指紋と言われるとあまりピンときませんが、個人的には部分構造というイメージで理解しています。このフィンガープリントは類似度の評価などに使われます。
フィンガープリントには今回紹介する"Morganフィンガープリント"以外にも"RDKitフィンガープリント"や"MinHashフィンガープリント"など様々な種類のフィンガープリントが存在します。
環境
- windows10
- conda 4.9.2
- python 3.7.1
- rdkit 2020.03.2.0
Morganフィンガープリントの解釈
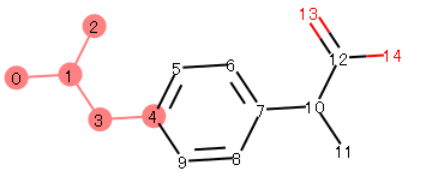
イブプロフェンのフィンガープリントについて解釈していきます。コードは 実践 マテリアルズインフォマティクスを参考にしています。
Morganフィンガープリントでは対象とする原子からの結合距離(radius)の設定が重要です。ここではまずradius=2としてフィンガープリントを取り出してみます。
from rdkit.Chem import rdMolDescriptors
# フィンガープリントの情報を入れる空の辞書を作成しておきます
bi = {}
fp = rdMolDescriptors.GetMorganFingerprintAsBitVect(ibuprofen, radius=2, bitInfo=bi)
辞書形式でフィンガープリントが取り出されるので、その一つを可視化してみます。(283は別途コードを実行して確認した数字です)
from rdkit.Chem import Draw
img = Draw.DrawMorganBit(ibuprofen, 283,bi)
img
このフィンガープリントがどういったルールで生成されているのか疑問に思ったので、以下でraduisを変えながら調べていきます。
ちなみにMorganフィンガープリントでは以下のルールで原子に色がつけられます。
- 紫色:部分構造の中心原子
- 黄色:芳香環に含まれる原子
- 灰色:脂肪族環に含まれる原子
radius=0 の場合
from rdkit.Chem import rdMolDescriptors
bi = {}
fp = rdMolDescriptors.GetMorganFingerprintAsBitVect(ibuprofen, radius=0, bitInfo=bi) # ここでradius=0を定義
print(len(list(fp.GetOnBits()))) # フィンガープリントの個数の表示
print(list(fp.GetOnBits())) # フィンガープリントの表示
7
[1, 80, 650, 807, 1057, 1380, 1873]
[1, 80, 650, 807, 1057, 1380, 1873]が何を意味するのか?なぜ7つなのか?読み解いていきます。
# biの中身を読み解くために、リストを一覧にします
list(bi.items())
[(1, ((1, 0), (10, 0))),
(79, ((3, 1),)),
(80, ((3, 0),)),
(283, ((0, 1), (2, 1), (11, 1))),
(310, ((5, 2), (9, 2))),
(389, ((14, 1),)),
(650, ((13, 0),)),
(807, ((12, 0), (14, 0))),
(854, ((1, 2),)),
(857, ((4, 2),)),
(900, ((10, 1),)),
(921, ((12, 2),)),
(955, ((3, 2),)),
(1057, ((0, 0), (2, 0), (11, 0))),
(1146, ((7, 2),)),
(1257, ((10, 2),)),
(1365, ((7, 1),)),
(1380, ((4, 0), (7, 0))),
(1750, ((5, 1), (6, 1), (8, 1), (9, 1))),
(1754, ((4, 1),)),
(1844, ((12, 1),)),
(1847, ((1, 1),)),
(1853, ((6, 2), (8, 2))),
(1873, ((5, 0), (6, 0), (8, 0), (9, 0))),
(1917, ((13, 1),))]
- 数(種類)は7つですが、重複を数えると総数は15でイブプロフェンを構成する原子の総数に一致
→radius=0 の場合は各原子単体を着目している
- 例えば1057は0,2,11の末端のC原子に注目し、それぞれは該当原子と結合している原子の種類と個数(C×1, H×3)が一致
- __各フィンガープリントは
[ ID, (中心原子, radius) ]を表している__と考えられます
radius=1 の場合
from rdkit.Chem import rdMolDescriptors
bi = {}
fp = rdMolDescriptors.GetMorganFingerprintAsBitVect(ibuprofen, radius=1, bitInfo=bi)
print(len(list(fp.GetOnBits())))
list(bi.items())
17
[(1, ((1, 0), (10, 0))),
(79, ((3, 1),)),
(80, ((3, 0),)),
(283, ((0, 1), (2, 1), (11, 1))),
(389, ((14, 1),)),
(650, ((13, 0),)),
(807, ((12, 0), (14, 0))),
(900, ((10, 1),)),
(1057, ((0, 0), (2, 0), (11, 0))),
(1365, ((7, 1),)),
(1380, ((4, 0), (7, 0))),
(1750, ((5, 1), (6, 1), (8, 1), (9, 1))),
(1754, ((4, 1),)),
(1844, ((12, 1),)),
(1847, ((1, 1),)),
(1873, ((5, 0), (6, 0), (8, 0), (9, 0))),
(1917, ((13, 1),))]
- フィンガープリントの総数は30個 ※15(原子の数) × 2(半径0 or 1の2水準) =30
- radius=1はradius=0も含んでおり、radiusを大きくするとフィンガープリントの数は増加
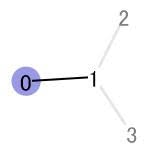
283のフィンガープリントを可視化してみます。
from rdkit.Chem import Draw
img = Draw.DrawMorganBit(ibuprofen, 283,bi)
img
- 283は0番の原子から半径1の結合(H3C-Cの部分)が該当し、これは(2,1)と(11,1)と同一である
radius=2 の場合
from rdkit.Chem import rdMolDescriptors
bi = {}
fp = rdMolDescriptors.GetMorganFingerprintAsBitVect(ibuprofen, radius=2, bitInfo=bi)
print(len(list(fp.GetOnBits())))
list(bi.items())
25
[(1, ((1, 0), (10, 0))),
(79, ((3, 1),)),
(80, ((3, 0),)),
(283, ((0, 1), (2, 1), (11, 1))),
(310, ((5, 2), (9, 2))),
(389, ((14, 1),)),
(650, ((13, 0),)),
(807, ((12, 0), (14, 0))),
(854, ((1, 2),)),
(857, ((4, 2),)),
(900, ((10, 1),)),
(921, ((12, 2),)),
(955, ((3, 2),)),
(1057, ((0, 0), (2, 0), (11, 0))),
(1146, ((7, 2),)),
(1257, ((10, 2),)),
(1365, ((7, 1),)),
(1380, ((4, 0), (7, 0))),
(1750, ((5, 1), (6, 1), (8, 1), (9, 1))),
(1754, ((4, 1),)),
(1844, ((12, 1),)),
(1847, ((1, 1),)),
(1853, ((6, 2), (8, 2))),
(1873, ((5, 0), (6, 0), (8, 0), (9, 0))),
(1917, ((13, 1),))]
- 総数は40個で15(原子の数) × 3(半径0,1,2の3水準) =45から5つ足りない
- (0,2), (2,2), (11,2), (13,2), (14,2)の5がない(最末端の原子ではradius=2が定義されていない仕様?)
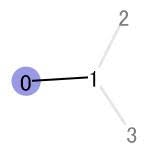
854のフィンガープリントを可視化してみます。
from rdkit.Chem import Draw
img = Draw.DrawMorganBit(ibuprofen, 854,bi)
img
- 854の(1,2)は1の原子から結合距離2の部分が該当
- 前述の通り、芳香環に含まれる原子4番は黄色で表示
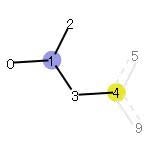
310のフィンガープリントも可視化してみます。
img2 = Draw.DrawMorganBit(ibuprofen, 310,bi)
img2

radius=3 の場合
from rdkit.Chem import rdMolDescriptors
bi = {}
fp = rdMolDescriptors.GetMorganFingerprintAsBitVect(ibuprofen, radius=3, bitInfo=bi)
print(len(list(fp.GetOnBits())))
list(bi.items())
31
[(1, ((1, 0), (10, 0))),
(79, ((3, 1),)),
(80, ((3, 0),)),
(126, ((3, 3),)),
(248, ((7, 3),)),
(283, ((0, 1), (2, 1), (11, 1))),
(310, ((5, 2), (9, 2))),
(389, ((14, 1),)),
(650, ((13, 0),)),
(718, ((5, 3),)),
(807, ((12, 0), (14, 0))),
(854, ((1, 2),)),
(857, ((4, 2),)),
(900, ((10, 1),)),
(921, ((12, 2),)),
(955, ((3, 2),)),
(978, ((6, 3),)),
(1025, ((10, 3),)),
(1057, ((0, 0), (2, 0), (11, 0))),
(1146, ((7, 2),)),
(1257, ((10, 2),)),
(1365, ((7, 1),)),
(1380, ((4, 0), (7, 0))),
(1568, ((4, 3),)),
(1750, ((5, 1), (6, 1), (8, 1), (9, 1))),
(1754, ((4, 1),)),
(1844, ((12, 1),)),
(1847, ((1, 1),)),
(1853, ((6, 2), (8, 2))),
(1873, ((5, 0), (6, 0), (8, 0), (9, 0))),
(1917, ((13, 1),))]
- 総数は46個で15(原子の数) × 4(半径0,1,2,3の4水準) =60からの不足数は14個
- 不足14個の内訳はradius=2で既に定義されなかった5つの原子(0,2,11,13,14)×2に加えて(1,3), (8,3), (9,3), (12,3)の4つ
- (5,3)や(6,3)が定義されて(9,3)や(8,3)が定義されていない点はよく分からない??
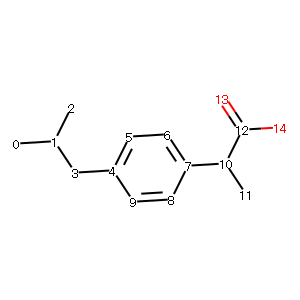
radius=3の718のフィンガープリントを可視化してみます。
from rdkit.Chem import Draw
img = Draw.DrawMorganBit(ibuprofen, 718,bi)
img

5番の原子を中心に結合距離3つ分の範囲がフィンガープリントとして含まれていることが分かります。
化合物中のフィンガープリントの表示
化合物とフィンガープリントを同時に表示してみます。以下のコードではradius=2で表示した854のフィンガープリントを選択します。
atom_id, radius = bi[854][0] # 854の最初のフィンガープリントの表示(実際には854は1つのフィンガープリントしかありませんが)
path_ids = Chem.FindAtomEnvironmentOfRadiusN(ibuprofen, radius, atom_id)
substructure = Chem.PathToSubmol(ibuprofen, path_ids)
ibuprofen.GetSubstructMatch(substructure)
ibuprofen
まとめ
Morganフィンガープリントの生成ルールについてご紹介致しました。
- 各フィンガープリントは [ ID, (中心原子, radius) ] を表している
- radiusを大きくすると含まれるフィンガープリントの数は増加する
といったことが分かりました。ただ、フィンガープリントが定義される/定義されないの基準はよく分からなかったので、そこはまた調べていきたいと思います。