はじめに
化合物中の特定の部分構造(官能基など)の数を数えていきたいと思います。今回は例として、自ら用意した化合物中のカルボキシル基の数を数えてみたいと思います。例えばこれができれば、スクリーニングしてきた化合物の中からカルボキシル基を1つだけ持つ化合物を抽出したいといったシーンで役立つはずです。
※マテリアルズインフォマティクス関係の内容を他にも投稿していますので、よろしければこちらの一覧から他の投稿も見て頂けますと幸いです。
SMILESとSMARTS
本題に入る前に「SMILES」と「SMARTS」について整理していきましょう。一般的な内容は次のサイトなどを参考にしてください。
SMILESは分子の表記方法でDr. David Weiningerが考案した分子記述言語です。
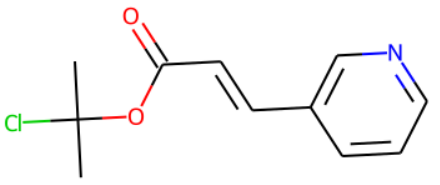
上記の分子は次のSMILESで表現されます。
ClC(C)(C)OC(/C=C/C1=CN=CC=C1)=O
SMARTSはSMILES言語を検索クエリ用に拡張した構造検索のための表記方法です。例えば[C,N]は脂肪族炭素または脂肪族窒素を表し、SMILESよりもより曖昧な表現が可能です。SMARTSに関する詳細はDAYLIGHT社のHPが参考になります。こちらのリンクにも詳しく記載があります。※なお、このリンクはインフォマジシャン研究所の藤様に教えていただきました
またSMARTSはSMARTS PLUSで確認することが可能です。SMARTSはSMILESよりも曖昧に表記できて検索性が高い分、使いこなすのはかなり難しい印象です。
環境
- python 3.7.13
- rdkit 2022.03.4
化合物中のカルボキシル基を数えていく
化合物の準備(実際にデータがある場合は不要)
まずは自分でサンプルとなる化合物を作っていきます。
from rdkit import Chem
# それぞれカルボキシル基を0,1,2,1,0個持つ化合物を定義します
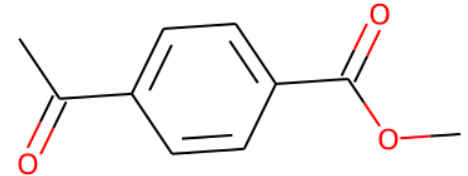
m0 = "CC(C1=CC=C(C(OC)=O)C=C1)=O"
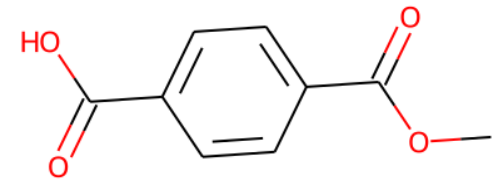
m1 = "OC(C1=CC=C(C(OC)=O)C=C1)=O"
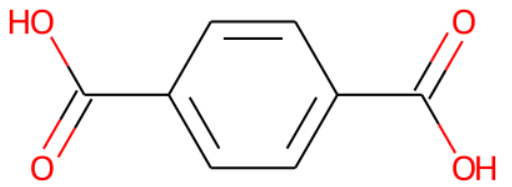
m2 = "OC(C1=CC=C(C(O)=O)C=C1)=O"
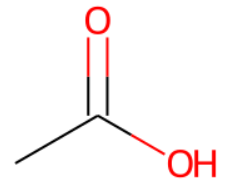
m3 = "CC(O)=O"
m4 = "OOCC"
それぞれの化合物を可視化します。
Chem.MolFromSmiles(m0)
Chem.MolFromSmiles(m1)
Chem.MolFromSmiles(m2)
Chem.MolFromSmiles(m3)
Chem.MolFromSmiles(m4)
それぞれの化合物を一括で処理できるようにデータフレームに格納します。
import pandas as pd
df = pd.DataFrame(
data={"compound": ["m0", "m1", "m2", "m3", "m4"],
"smiles": [m0, m1, m2, m3, m4]}
)
df

実際にデータがある場合はこのようなデータフレームを読み込んでここから実行していくことが想定されます。次にsmilesをMolに変換してリストで保持します。
mol_list = [Chem.MolFromSmiles(x) for x in list(df["smiles"]) if x is not None]
数える部分構造の定義
今回はカルボキシル基をSMARTS表記で定義します。
q = Chem.MolFromSmarts("*C(=O)-[OH1]")
q
部分構造の探索
HasSubstructMatchは指定した部分構造を持つか否かで返ってきます。参考
mol_list[0].HasSubstructMatch(q)
False
mol_list[1].HasSubstructMatch(q)
True
0番目の分子はカルボキシル基を含み、1番目の分子はカルボキシル基を含まないのでこのような出力になります。
GetSubstructMatchesは指定した部分構造がマッチする原子インデックスが返ってくるので、これをlenで取り出して部分構造の数を数えます。
import numpy as np
for i in np.arange(len(df)):
print(len(mol_list[i].GetSubstructMatches(q)))
0
1
2
1
0
実用を意識した例
実際の使用を想定して、部分構造を指定した個数分だけ持っている化合物のみを抽出していきます。ここでは、「カルボキシル基を1つだけ持つ化合物」を選抜していくことにします。
# 着目する構造な数を指定する
num = 1
index_list = []
for i in np.arange(len(df)):
count = len(mol_list[i].GetSubstructMatches(q)) # 着目する構造の数をカウント
if count == num:
index_list.append(i) # 条件を満たす番号をリストに格納
new_df = df.iloc[index_list,:]
new_df
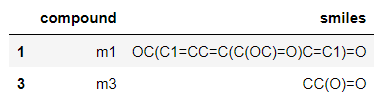
このようにカルボキシル基を1つだけ持つリストの1番目と3番目の化合物のみが抽出されました。
まとめ
今回はカルボキシル基を例として化合物中の部分構造を数える方法を紹介いたしました。次は別の部分構造でトライしてみたいと思います。