iNaturalistとは
iNaturalistは市民科学のプロジェクトであり、ナチュラリスト、市民科学者と生物学者を対象としたオンラインのソーシャル・ネットワーキング・サービスでもある。
iNaturalist - ウィキペディアより
iNatulalistは全世界の参加者から得られた生物の観測記録を共有・公開するためのSNSです。カリフォルニア科学アカデミーとナショナルジオグラフィック協会によって共同設立されました。公式サイトはこちら、背景についてはこちらを参照してください。
観察記録を登録するには、スマホのアプリ(iOS、Android)で位置情報が付いた生物の観察写真を投稿するだけでOKです。日時や位置情報は自動入力さますし、種名はわからなくても詳しい人が同定して提案してくれます(画像による自動同定もあります)。
使い方はYoutubeなどで解説されています。
外出ができない昨今ですがiNaturalistには外出しなくても参加できます(詳しくはこちらExploring Nature When You're Stuck at Home)。例えば家の中や庭先などにいる生物を登録したり(例えばこちらのプロジェクト(昆虫注意))、過去の写真を登録したりすることで観察記録をアップできます。
※ただし自宅の位置情報が漏れないよう注意してください。不安であれば位置情報を「不明瞭」や「登録しない」にして投稿すると良いでしょう。
また以下のように既存の観測記録を見て楽しむこともできます。マップから詳細情報がプレビューできるようになっているのでストレスなく検索できます。
公式サイトより
日本ではやっている人が少ないそうなので(日本の記録はとても少ない)、是非参加してみてください。
ここまではiNaturalistの宣伝になってしまったのですが、実はiNaturalistにはAPIがあってかなり簡単に観察記録が抽出できます。
そこでここでは、RからiNaturalist APIを使って生物の観察記録を抽出して地図上にマッピングしてみます。こんなサービスもあるのかということで、あまり生物に興味がない方にも興味を持っていただけたらと思います。
iNaturalist API
APIの詳細についてはこちらから確認できます。各APIの詳細はプルダウンで確認できます。
投稿や編集等のAPIには認証が必要ですが検索には認証が必要ないようです。
今回はObservation Searchを使用します。
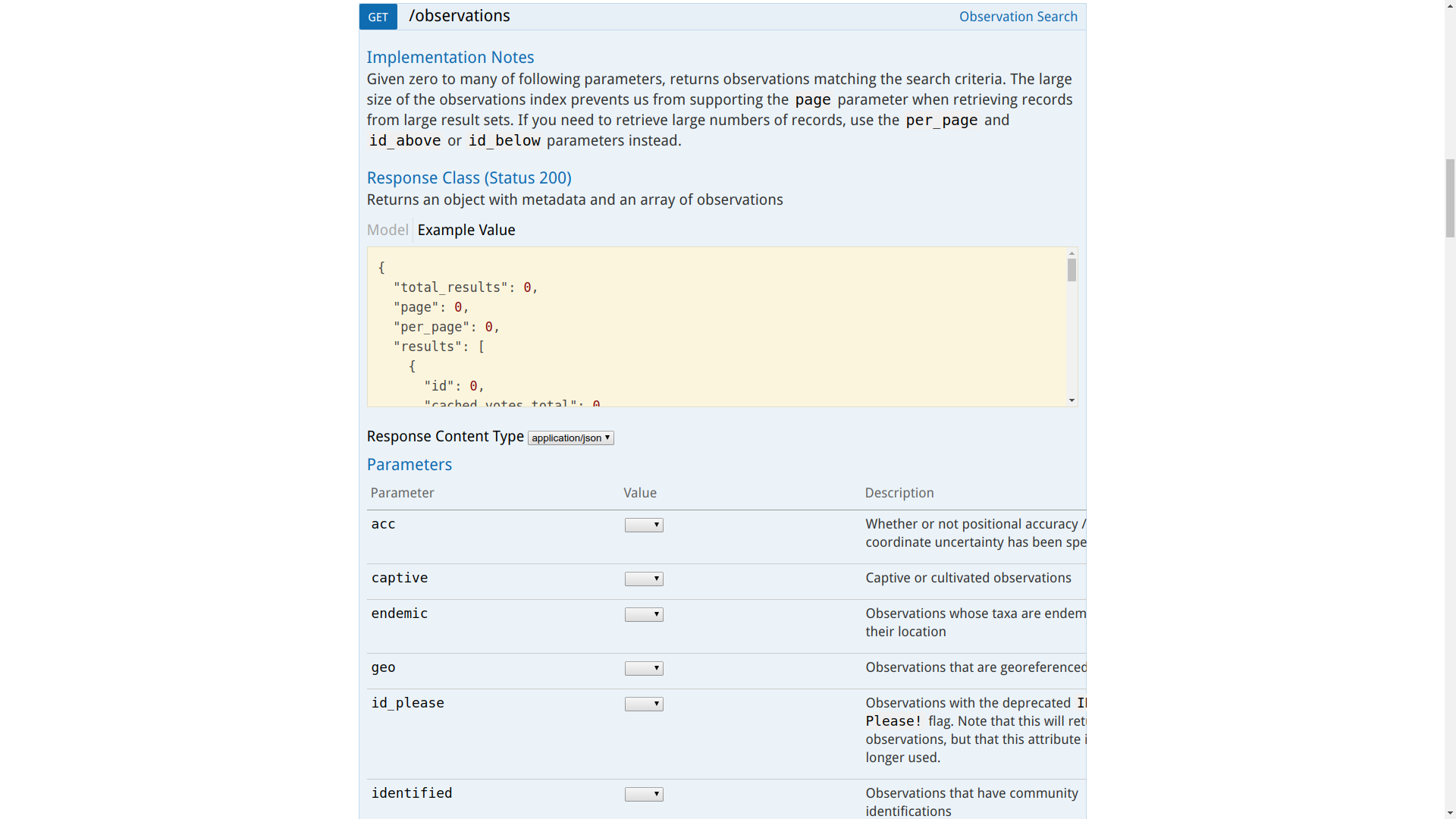
公式サイトより
なお上記APIのページによると、「リクエストは1分間で100回までに制限しているが、できれば60回までにしてほしい。1日あたりでも10000回以下にしてほしい」とのことです。
Rのコード
必要パッケージ
パッケージはデータの取得と整形にhttr、tidyverse、magrittrを、地図への描画にsfとtmapを使用します。インストールされていない場合は以下のコマンドでインストールしてください。
install.packages("httr")
install.packages("tidyverse")
install.packages("magrittr")
install.packages("sf")
install.packages("tmap")
インストールできたらRに読み込んでおきます(以下のコードでは::を使っているのであまり必要ありませんが一応)。
library(httr)
library(tidyverse)
library(magrittr)
library(sf)
library(tmap)
データ取得用の関数を定義
さきほどのObservation Searchにリクエストを送ってレスポンスを受け取る関数を定義します。
以下が関数のコードです。
クエリをリストにしてhttr::GETに渡すとURLに変換してレスポンスを取得してくれます。
結果は複数のページに別れているのでpurrr::mapで繰り返し処理をして全ページの結果を取得します。
getObservations <- function (query) {
url <- "https://api.inaturalist.org/v1/observations" # ベースurlを設定
query.preview <- c(query, list(page = 1)) # 参照用のページを指定
preview <- httr::GET(url = url, query = query.preview) # レスポンスを取得
total.results <- preview %>% httr::content() %>% magrittr::extract2("total_results") # 総記録数を取得
nPage <- floor(total.results / query.preview$per_page) + 1 # 総ページ数を取得
Sys.sleep(10) # サーバー負荷軽減のため10秒遅延
response <- purrr::map(1:nPage, function(i) { # 各ページに対して繰り返し処理を行い、レスポンスをリストに格納
query.page <- c(query, list(page = i)) # ページを指定
response.page <- httr::GET(url = url, query = query.page) # レスポンスを取得
Sys.sleep(10) # サーバー負荷軽減のため10秒遅延
return(response.page)
})
return(response)
}
これで各ページについてのレスポンスをリストの各要素に格納できます。
参考:Querying APIs in R
データを整形する関数を定義
レスポンスの内容はhttr::contentで確認できます。さらにstrを使うとリストの構造を見ることができて、大量の情報が含まれていることを確認できます。
今回はIDと学名、観察地点の緯度経度を使うので、以下の関数を用いて結果を抽出してtibbleに整形しました。
getCoords <- function(response) {
coords.list <- purrr::map(response, function(response) { # リスト内のレスポンスに対して繰り返し処理
result <- httr::content(response) # レスポンスをリストに変換
id <- purrr::map(result$results, function(x) {x %>% magrittr::extract2("id")}) %>% unlist %>% as.character # 観測IDをまとめる
name <- purrr::map(result$results, function(x) {x %>% magrittr::extract2("taxon") %>% magrittr::extract2("name")}) %>% unlist # 学名をまとめる
long <- purrr::map(result$results, function(x) {x %>% magrittr::extract2("geojson") %>% magrittr::extract2("coordinates") %>% magrittr::extract2(1)}) %>% unlist # 経度をまとめる
lat <- purrr::map(result$results, function(x) {x %>% magrittr::extract2("geojson") %>% magrittr::extract2("coordinates") %>% magrittr::extract2(2)}) %>% unlist # 緯度をまとめる
coords.tmp <- tibble::tibble(id = id, name = name, long = long, lat = lat) # 各データをtibbleに格納
})
coords <- dplyr::bind_rows(coords.list) # ページごとのtibbleを結合して一つにする
return(coords)
}
magrittr::extract2を多用していますが、これはリストの要素を抽出する際の[[のエイリアスです。これとpurrr::mapを組み合わせることでネストしたリストから観察記録ごとのデータを抽出しています。
Tidyverse系の関数の使い方については以前の記事(R パッケージとチートシートの対応表)から該当パッケージのチートシートを見てもらうとわかりやすいと思います。またmagrittrの使い方についてはパッケージのヘルプが参考になります。
処理の実行
今回はナミアゲハ Papilio xuthusとクロアゲハ Papilio protenorの分布を比較してみたいと思います。
クエリを作成
クエリは以下のように設定しました。「2020年4月21日以降のデータで信頼性が高く、位置情報精度が高い」ものを抽出します。各パラメータの詳細はさきほどのAPIのページで確認できます。
ただしgeoprivacy = "open"を抜くと、位置情報がない観察記録も含まれてしまい整形時にエラーが出るようになるので注意してください。またこの設定だとオブジェクトサイズが少し大きくなるので(1種についてのレスポンスをまとめると40MBくらい)、スペックが不安であれば期間を短くするなどすれば良いと思います。
# Papilio xuthus
Query.xu <- list(verifiable = "true", taxon_name = "Papilio xuthus", d1 = "2010-04-21", geoprivacy = "open", per_page = 100, order = "desc", order_by = "created_at")
# Papilio protenor
Query.pr <- list(verifiable = "true", taxon_name = "Papilio protenor", d1 = "2010-04-21", geoprivacy = "open", per_page = 100, order = "desc", order_by = "created_at")
データを取得
定義した関数とクエリを用いてデータを取得します。10秒遅延させているので数分程度かかります。
# Papilio xuthus
Observation.xu <- getObservations(Query.xu)
# Papilio protenor
Observation.pr <- getObservations(Query.pr)
データを整形
定義した関数を用いてデータをtibbleに整形します。
# Papilio xuthus
Coords.xu <- getCoords(Observation.xu)
# Papilio protenor
Coords.pr <- getCoords(Observation.pr)
すると以下のようなtibbleが取得できます。
> Coords.xu
# A tibble: 534 x 4
id name long lat
<chr> <chr> <dbl> <dbl>
1 42721383 Papilio xuthus 102. 30.0
2 42721333 Papilio xuthus 102. 30.0
3 42721329 Papilio xuthus 102. 30.0
4 42098215 Papilio xuthus 137. 35.0
5 41895327 Papilio xuthus 114. 22.5
6 41889596 Papilio xuthus 140. 35.7
7 41813391 Papilio xuthus 114. 22.5
8 41733726 Papilio xuthus 114. 22.4
9 41588008 Papilio xuthus -158. 21.3
10 41429604 Papilio xuthus 114. 22.3
# … with 524 more rows
> Coords.pr
# A tibble: 593 x 4
id name long lat
<chr> <chr> <dbl> <dbl>
1 42740576 Papilio protenor 114. 22.4
2 42561416 Papilio protenor 114. 22.4
3 42547829 Papilio protenor 114. 22.3
4 42448074 Papilio protenor 99.5 12.8
5 42246448 Papilio protenor 122. 25.0
6 42239153 Papilio protenor 121. 23.3
7 42213416 Papilio protenor 101. 15.9
8 42148119 Papilio protenor 114. 22.3
9 42056501 Papilio protenor 101. 21.0
10 41899142 Papilio protenor 114. 22.2
# … with 583 more rows
地図にマッピング
以上で座標が取得できたので地図にマッピングします。
座標をポイントデータに変換
座標をsfクラスのポイントデータに変化します。
# Papilio xuthus
Point.xu <- sf::st_as_sf(Coords.xu, coords = c("long", "lat"), crs = 4326)
# Papilio protenor
Point.pr <- sf::st_as_sf(Coords.pr, coords = c("long", "lat"), crs = 4326)
tmapでインタラクティブマップに描画
sfクラスにしてしまえばggplot2でもleafletでも描画できるのですが、今回はtmapを使用してインタラクティブマップ上にポイントを表示します(tmapの使い方は以前書いた記事などを参考にしてください)。
tmap::tmap_mode("view")
tm <- tmap::tm_shape(Point.xu) + tmap::tm_dots(col = "yellow", alpha = 0.5, size = 0.1) +
tmap::tm_shape(Point.pr) + tmap::tm_dots(col = "black", alpha = 0.5, size = 0.1)
tm
実行するとブラウザ(RStudioだとViewer)で以下のような地図が開きます。ハワイにも記録がありますがここではアジア域にズームしています。
ナミアゲハが黄色でクロアゲハが黒色です。
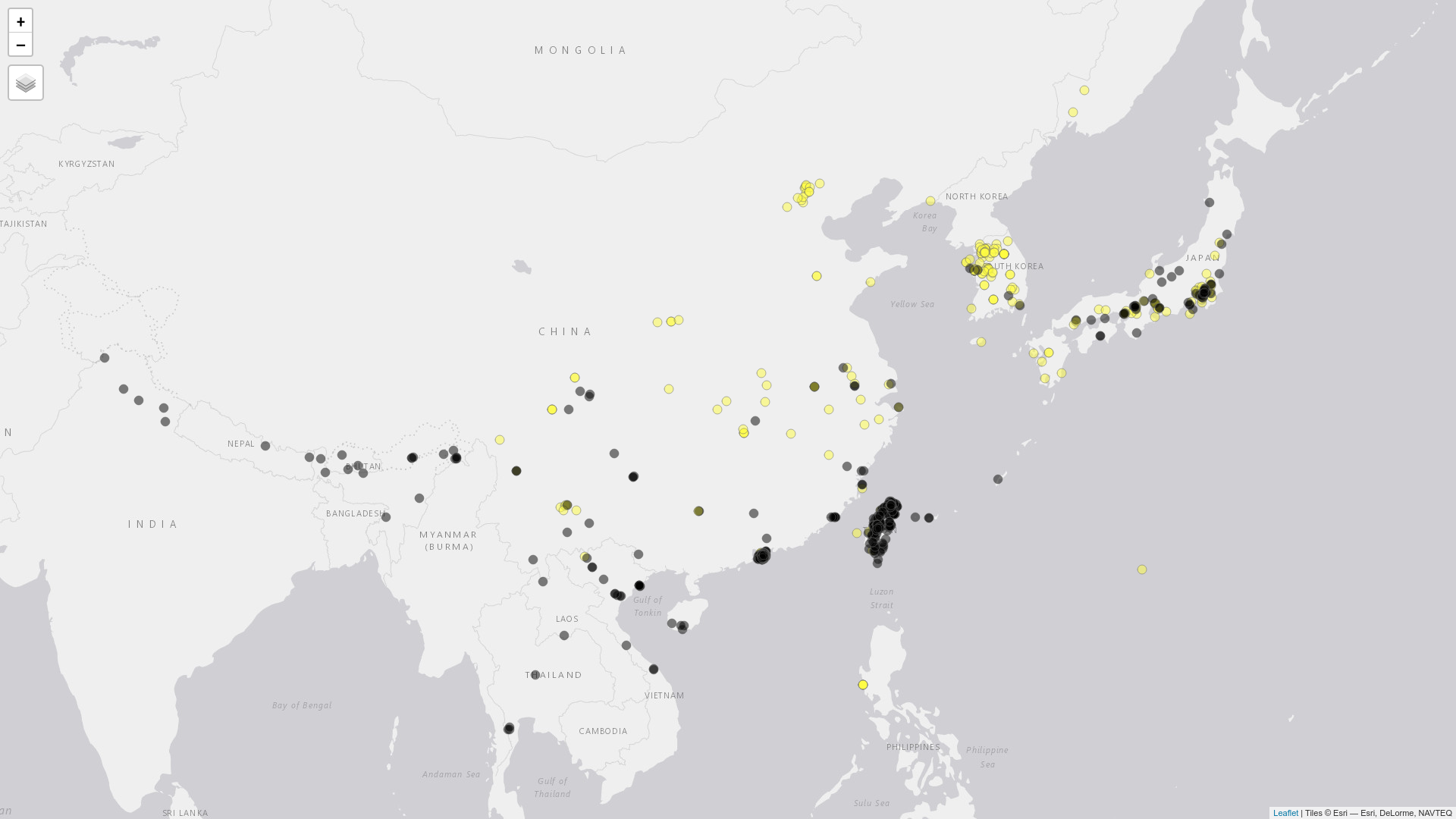
クロアゲハはヒマラヤ沿いに分布しているのが特徴的ですね。ナミアゲハは相対的に北の方に分布しています。このデータからでもいろいろなことに気づけそうです。
ただ一方で、自然分布を完全にはカバーできていないだろうという点には注意しなければいけません。実際、記録は一部地域(特に大都市)にかなり集中してしまっています。あくまでも「観察記録の分布」なので、「実際の分布」を推定するためには更に踏み込んだ解析が必要です。
iNaturalist APIの使い方の一例を示してみましたがいかがでしたでしょうか?
このような形で自然に触れてみるのも楽しいと思います!