初めに
今回の記事では以下の論文のデータで解析を行い、論文の結果と比較していきます。
Haney, M.S., Pálovics, R., Munson, C.N. et al. APOE4/4 is linked to damaging lipid droplets in Alzheimer’s disease microglia. Nature 628, 154–161 (2024). https://doi.org/10.1038/s41586-024-07185-7
特に論文の前半で述べられているミクログリアはACSL1によって定義される脂質転写状態があるという部分を検証します。
論文概要
この論文はアルツハイマー病でのAPOE4/4と脂肪滴との関連を示したものとなります。
ここでアルツハイマー病について確認します。
アルツハイマー病については以下のテキストが参考になります。
ここではアミロイドカスケード仮説について説明します。
この仮説を図示すると以下となります。
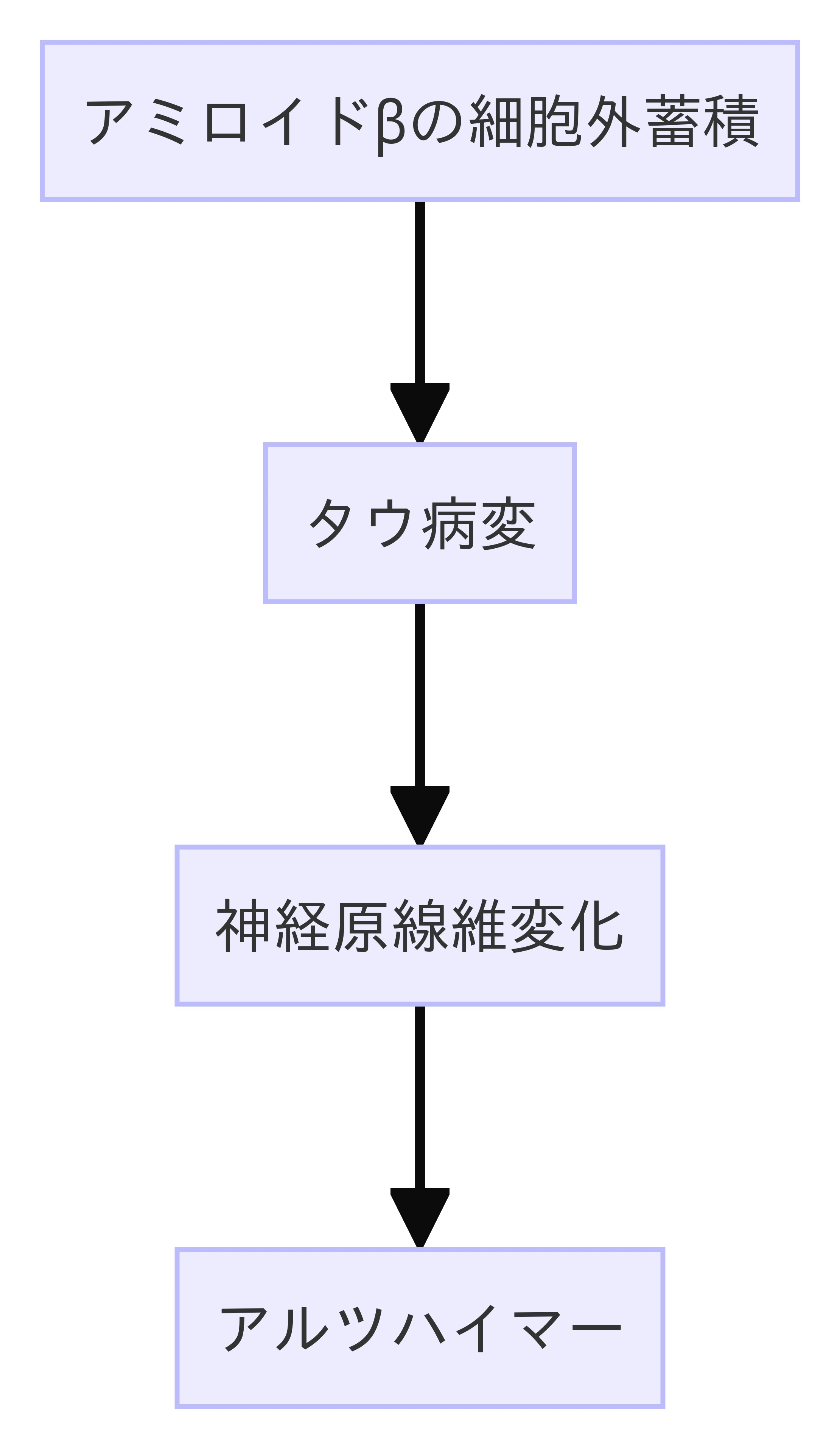
この論文の目的は以下となります。
Several genetic risk factors for Alzheimer’s disease implicate genes involved in lipid metabolism and many of these lipid genes are highly expressed in glial cells1. However, the relationship between lipid metabolism in glia and Alzheimer’s disease pathology remains poorly understood.
つまりここではアルツハイマー病とグリア細胞中での脂肪滴との関連をみつけることを目的としています。
そしてこの論文で示されることは以下となります。
In human induced pluripotent stem cell-derived microglia, fibrillar Aβ induces ACSL1 expression, triglyceride synthesis and lipid droplet accumulation in an APOE-dependent manner. Additionally, conditioned media from lipid droplet-containing microglia lead to Tau phosphorylation and neurotoxicity in an APOE-dependent manner. Our findings suggest a link between genetic risk factors for Alzheimer’s disease with microglial lipid droplet accumulation and neurotoxic microglia-derived factors, potentially providing therapeutic strategies for Alzheimer’s disease.
つまりAPOE4/4 genotypeでは、上のアミロイドカスケード仮説の図の中の最初の矢印の間にACSL1発現、トリグリセリド、脂質滴などが入ることが今回示されたということです。
環境、用いたデータ
・環境
今回環境は様々なものを利用したので後で経緯を記します。
具体的にはGoogle colabolatory, 個人のwindows, 研究室のマシンの順に試しました。
・データ
GSE254205
使うコードはこの記事で作成したものに少し変更を加えたものを使用します。
scRNAseqが何なのかというのはこちらを参考にしてください
環境の試行錯誤
環境で苦労したので記載しています。
Google colabolatory編
まずはgoogle colabolatoryで行います。
まずはデータを取り込みましょう。
adata = an.read_h5ad('/content/drive/MyDrive/GSE254205_ad_raw.h5ad/ad_raw_doublet_scores.h5ad')
このまま進んでいくと
adata = adata[:, adata.var.highly_variable]
ここでメモリを大量に使ってしまいます。
なので一度この処理が終わったら処理後のデータを保存し、読み込むことで次はここから作業を始めることができメモリの節約となります。
# AnnDataオブジェクトを保存
adata.write('/content/drive/MyDrive/GSE254205_ad_raw.h5ad/ad_raw_doublet_processed1.h5ad')
# 保存されたAnnDataオブジェクトを読み込む
adata = sc.read('/content/drive/MyDrive/GSE254205_ad_raw.h5ad/ad_raw_doublet_processed1.h5ad')
ここはうまく切り抜けられましたが、次が問題です。
sc.pp.regress_out(adata, ['total_counts', 'pct_counts_mt'])
ここでメモリを使い果たしてしまいます。この問題はgoogle colabolatory上では解決できず、localで.pyで動かせばうまくいくのではないかと考えました。
個人のwindows編
実行環境
OS - Windows 11 Home 23H2
CPU - 12th Gen Intel(R) Core(TM) i5-12400F 2.50 GHz
メモリ - 16.0 GB
wsl2で実行しました。しかしここでkilledされてしまいます。
sc.pp.regress_out(adata, ['total_counts', 'pct_counts_mt'])
https://zenn.dev/suzuki5080/articles/1438d52377b9df
この記事を参考にメモリを14Gに設定してやりましたが駄目でした。
ここでメモリが全然足りていないので研究室の計算機で行うことにしました。
研究室のマシン編
実行環境
OS - 5.4.0-67-generic #75-Ubuntu SMP Fri Feb 19 18:03:38 UTC 2021 x86_64 x86_64 x86_64 GNU/Linux
CPU - Intel(R) Core(TM) i9-10920X CPU @ 3.50GHz ×23
メモリ - 131.6 GB
研究室の計算機に入るためには2段階sshが必要です。
2以下のサイトが参考になります。
https://bi.biopapyrus.jp/os/linux/ssh.html
https://dev.classmethod.jp/articles/bastion-multi-stage-ssh-only-local-pem/
まずは公開鍵と秘密鍵を作成し、公開鍵をサーバー管理者に送る必要があります。
鍵の作成方法は以下を参照してください
https://sc.ddbj.nig.ac.jp/application/ssh_keys_ssh-keygen_win/
公開鍵をサーバー管理者に送ることができたら~/.ssh/configを設定します。以下が参考になります。
https://s10i.me/whitenote/post/42
~/.ssh/configがなければ作成します。
# 空ディレクトリ作成
$ mkdir ~/.ssh
# 空ファイル作成
$ touch ~/.ssh/config
ここに以下の様に設定しました。(具体的なものはサーバー管理者に聞いてください)
host serverA
HostName リモートホストのIPアドレス or ホスト名(名前解決可能な場合)
User リモートホストのユーザー名
Port 22222
ProxyCommand ssh -W %h:%p serverB
IdentityFile ~/.ssh/id_rsa(秘密鍵ファイル名)
host serverB
hostname リモートホストのIPアドレス or ホスト名(名前解決可能な場合)
user リモートホストのユーザー名
Port 22
identityfile ~/.ssh/id_rsa(秘密鍵ファイル名)
これはserverBを踏み台にserverAに接続するものです。
ssg serverA
これで接続できます。接続を切るときは
exit
です。
次にAnacondaをserverAにインストールしていきます。以下を参照してください。
https://qiita.com/L40S38/items/32115e0683218c621d88
ここでlinuxのダウンロードは3つありますが、今回は上の環境を参照してx86の一番上を選択します。

最後のデフォルトでconda環境に入るか聞かれるところはyesにしました。
インストールができたら動作確認します。
conda -Vで結果が表示されれば大丈夫です。
次にパッケージの更新をします。conda update --allこれで最新に更新されます。
次はファイルをserverAにあげましょう。今回はscpコマンドを使います。scpコマンドの基本的な使い方は以下が参考になります。
https://qiita.com/ritukiii/items/c724f09fe66fedf2618b
今回は2段階でのscpなので以下を参考にします。
https://qiita.com/ponsuke0531/items/4721ac64f82a8e191580
~/.ssh/configは上で作成したので
scp {自分ディレクトリパス} {置き場のホスト名}:{置き場ディレクトリパス}
で楽をすることができます。ここで注意することはwindowsのパスとLinuxのパスは書き方が違うということです。以下が参考になります。
https://dev.classmethod.jp/articles/windows-wsl-path/
windowsではC:\Usersから始まりますが、これをwslpathコマンドを使うとLinuxのパスに変更することができます。
wsl wslpath 'C:\Users'
/mnt/c/Users
この様にしてscpコマンドで踏み台の向こうのホストにファイルを送ることができます。
準備が整ったらVSCODEを使ってssh接続して解析を始めていきます。
解析
論文での最初の議論であるミクログリアはACSL1によって定義される脂質転写状態があるという部分を独自に検証しました。
データの概要
サンプルはBanner Sun Health Research InstituteのBrain and Body Donation Programによるヒト脳です。
このサンプルから遠心分離で核を抽出して解析したものです。核を1つずつ解析する分析をsnRNA-seq (single nuclei RNA-seq)といいます。
snRNA-seqライブラリは10x GenomicsのChromium Next GEM Single Cell 3′ v.3.1によって作成されました。
サンプルは3つの群からなっています。すなわちcontrol, ad APOE3/3, ad APOE4/4です。
controlはAPOE3/3遺伝子を持つ対照群であり、今回関連を指摘したいAPOE4/4遺伝子をもたない、非アルツハマーの群です。ad APOE3/3はAPOE4/4遺伝子をもたない、アルツハイマーの群でad APOE4/4はAPOE4/4遺伝子を持つアルツハイマーの群です。
| type | number of sample | explanation |
|---|---|---|
| control | 36295 | APOE3/3遺伝子を持つ対照群 |
| ad APOE3/3 | 31108 | APOE3/3遺伝子を持つアルツハイマー患者 |
| ad APOE4/4 | 32937 | APOE4/4遺伝子を持つアルツハイマー患者 |
QC(クオリティーチェック)
・ダブレット除去
ダブレットとは2つの細胞を1つの細胞として認識してしまうことです。これは当然解析に影響を与えるのでQCの1つとして除去します。ダブレットについて詳しくは以下が参考になります。
ダブレット除去は論文と同じコードで行いました。結果は以下です。
・ミトコンドリア、リボソームで足切り、フィルタリング
ミトコンドリアは質の悪い細胞でよく発現します。ミトコンドリアはそれ自体が膜で囲まれているため、細胞の膜が破壊された後でもRNAが流出しにくいからです。
adata = adata[adata.obs.total_counts >= 1000, :]
adata = adata[adata.obs.n_genes_by_counts >= 500, :]
adata = adata[adata.obs.pct_counts_mt < 10, :]
adata = adata[adata.obs.pct_counts_rb < 10, :]
以上で設定しました。
アノテーション
論文でのアノテーションはクラスタリングして、そのクラスターに対して手動でアノテーションしていました。アノテーションの結果は以下となっています。
論文ではクラスターへのアノテーション手で行っていましたが、今回はdecoupler 1.8.0を使用して自動で行いました。
Pythonを使った実装方法は以下の記事を参考にしてください。
decouplerの方がクラスターを多くとっているもののおおむね同じであることが見て取れます。ただし名前がすべて一致するわけでないのには注意してください。
例えば今回ターゲットとなってくるMicrogliaはdecouplerではMacrophagesとなっています。Microgliaは免疫をつかさどるグリア細胞でMacrophagesと機能はにているので得に問題はありません。
decouplerの方がより細かいセルタイプをアノテーションできていることがわかります。
Psedobulk解析
Psedobulk解析とは、シングルセルで個別に解析していたものを、グループを定めて1つの集団として、集団同士を比較する解析方法です。
今回は、nd APOE3/3, ad APOE3/3, ad APOE4/4の3グループに分けて、これらの間で比較していきます。
ここではMicrogliaでの遺伝子発現の違いをVolcano Plotで見ていきます。Volcano Plotは以下の中で言及しています。
x軸のlogFCsとはlog fold changeの略で、2つの条件間での遺伝子発現量の変化を対数スケールで表したものです。具体的にはlogFC > 0は発現量が増加、logFC < 0は発現量が減少を表します。
y軸の-logpvalueはpvalueの対数にマイナスしたもので値が大きいほど統計的に有意であることを表します。
それぞれの点は遺伝子を表します。
・Volcano Plot (nd apoe 33 v.s. ad apoe 33)
ACSL1はlogFC、-logpvalue共に大きな値となっているので、ACSL1はnd apoe 33に比べてad apoe 33において発現量が多くかつそれは統計的に有意であることがわかります。
・Volcano Plot (nd apoe 33 v.s. ad apoe 44)
こちらも同様にACSL1はlogFC、-logpvalue共に大きな値となっているので、ACSL1はnd apoe 33に比べてad apoe 33において発現量が多くかつそれは統計的に有意であることがわかります。
比較として論文でのVolcano Plotは以下となります。
論文のプロットとかなり異なっていることがわかります。原因は不明です。しかし今回の結果ではad apoe 33とad apoe 44を比較すると依然としてad apoe 44のほうがx軸y軸ともに値が大きく、有意に発現量が増加していることがわかります。
クラスターごとのACSL1の発現の様子は以下となります。赤枠で囲ったところがミクログリア(マクロファージ)のクラスターとなります。
MacrophagesでACSL1の発現が高いことが見て取れます。
論文でのクラスターごとのACSL1発現量は下となっており結果が似通っていることがわかります。
結果まとめ
以上よりミクログリアにおいてACSL1がよく発現していることがわかりました。
さらに元論文の1eより引用した以下の図よりACSL1が脂肪に関係することがわかります。
よってミクログリアにはACSL1によって定義される脂質転写状態があることを確認することができました。
最後に
今回は環境に苦労しました。つまり自前で用意できるものではRAMが足りず、マシンパワーの重要性を痛感しました。
また上のようにミクログリアにはACSL1によって定義される脂質転写状態があることを示すことができましたが、元論文ではさらにミクログリアをサブクラスタリングして詳細に脂質転写状態について議論しています。機会があればさらに詳しく解析してみたいと思います。
(この記事は研究室インターンで取り組みました:https://kojima-r.github.io/kojima/)








