はじめに
概要
GraphVizによる決定木描画の不満点
機械学習が流行の今、pythonにおいてはscikit-learnを使う方が多いですよね。
その第一歩として、sklearnのDecisionTreeClassifierでIrisやTitanicを決定木分析するかと思います。
(ぼくはそうでした)
sklearnのDecisionTreeClassifierでは、学習した決定木をDOT言語を介してGraphVizで可視化することができます。
↓こんなふうに。
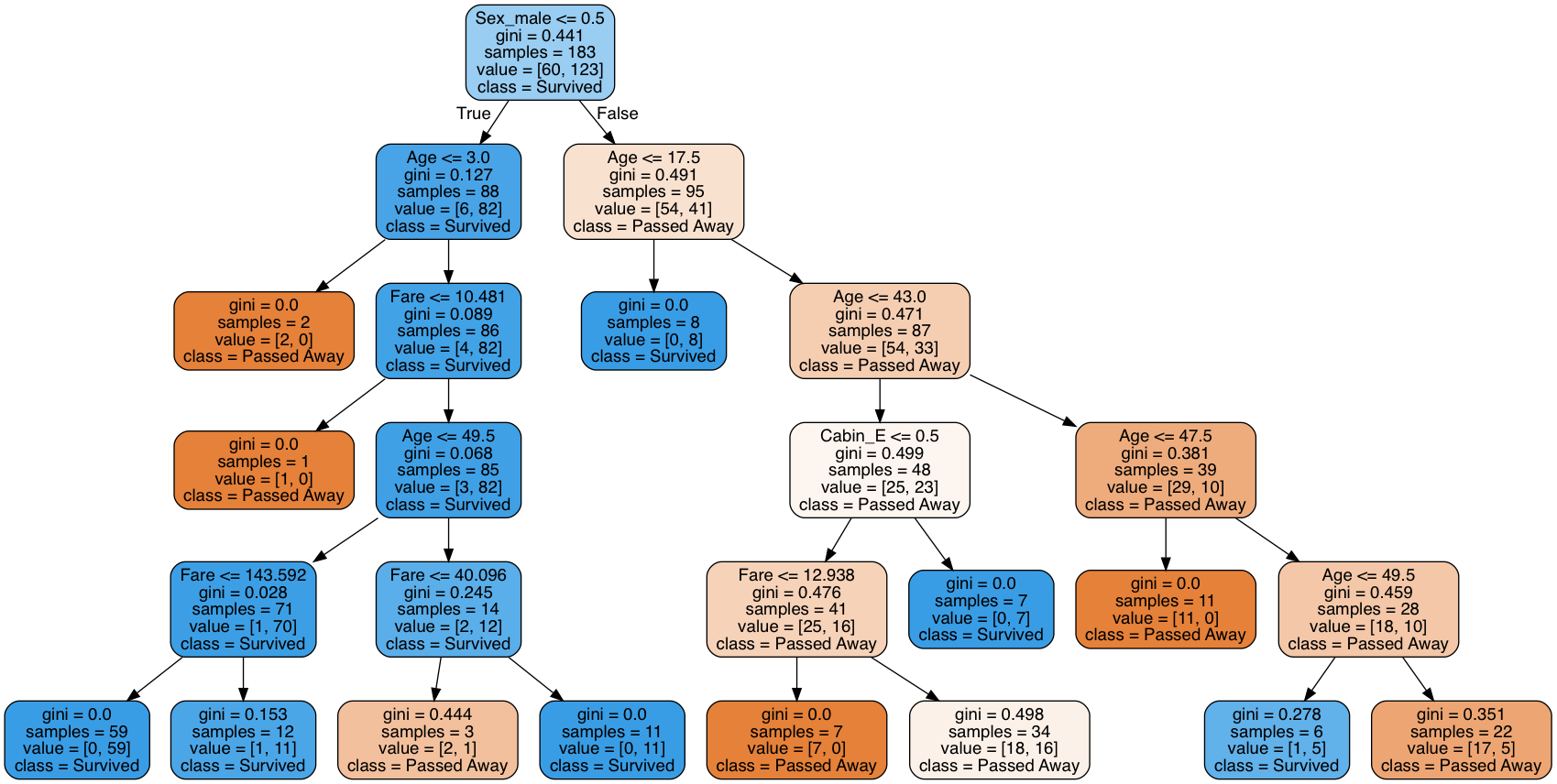
ダサい
勿論、分析に必要な情報は揃っていてわかりやすい秀逸な図だと思います。でもダサさが溢れ出てますね。
Tableauとかオサレなツールが登場している時代に、この古臭さ。
これではインスタグラムに投稿できないです。
もっとフォトジェニックに描画できないものでしょうか。
そこで本記事では、plotlyの3-D scatterをつかって決定木を描画しました。
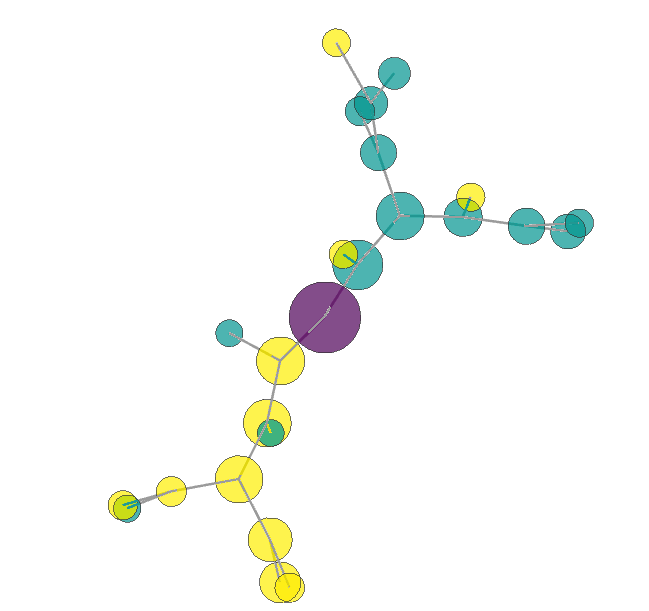
結果1: 分子構造みたいなの
※マウス操作で拡大縮小、回転いろいろできます。
https://nekoumei.github.io/photogenic_decision_tree/Photogenic_DecisionTree_molecular.html
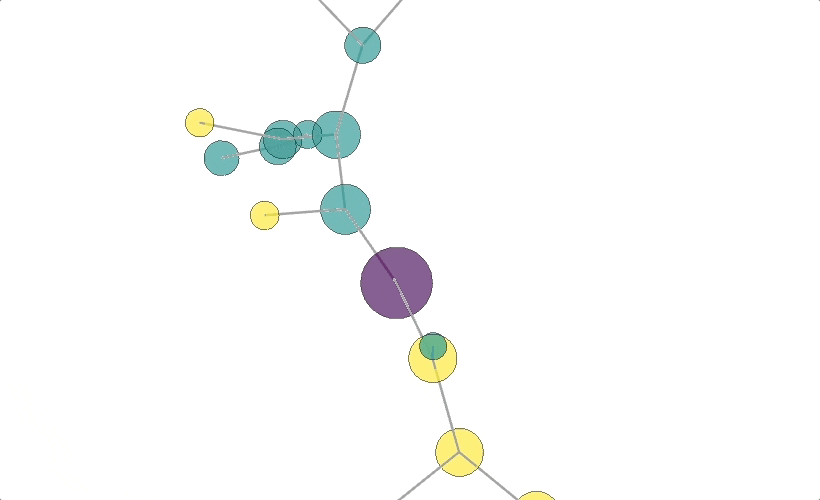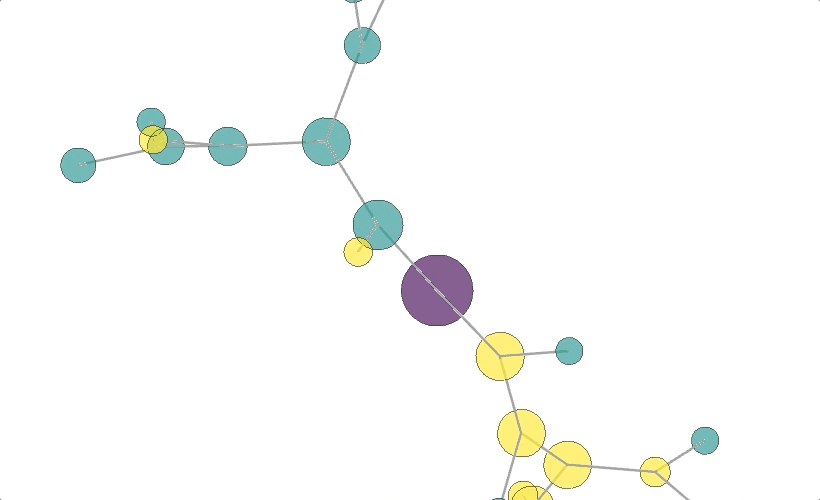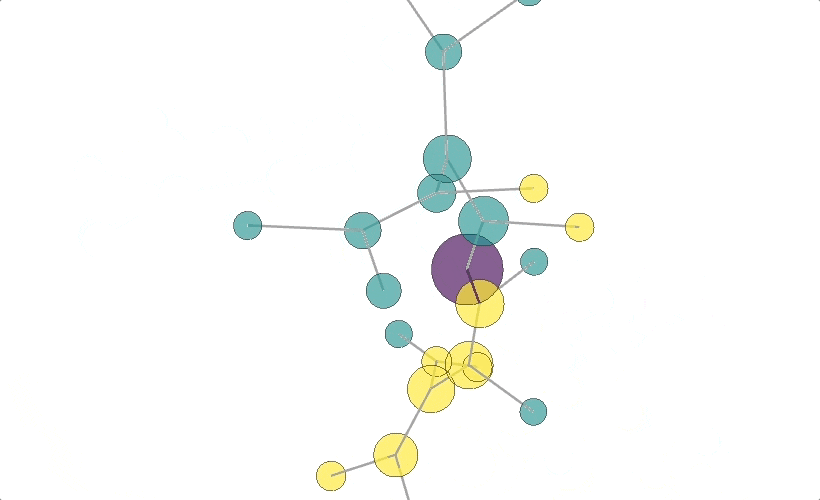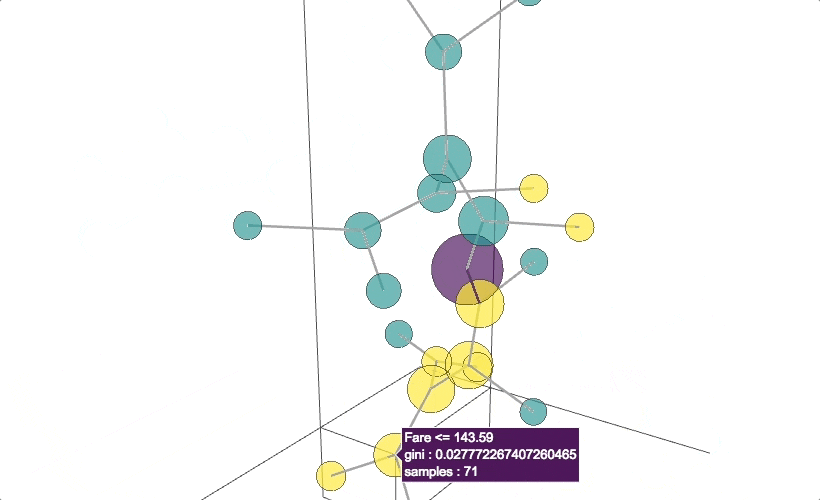
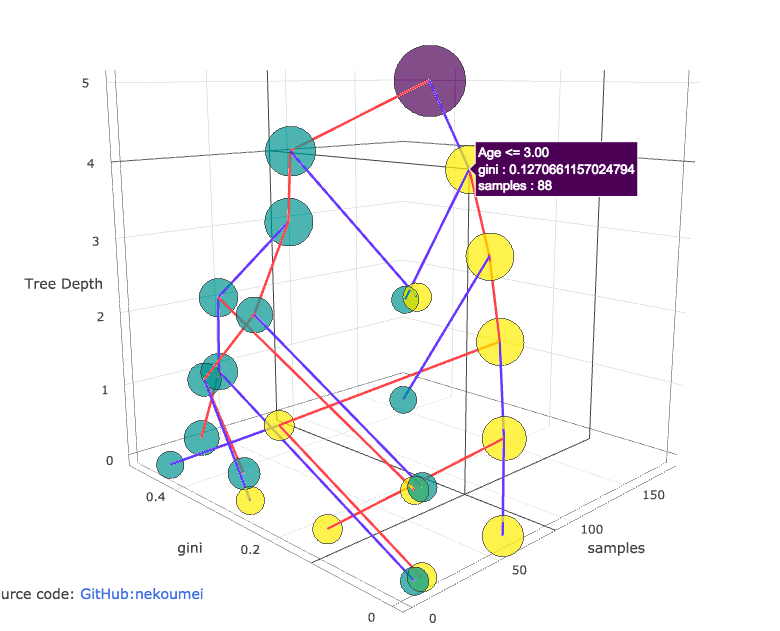
結果2: 各情報を可視化した木構造
https://nekoumei.github.io/photogenic_decision_tree/Photogenic_DecisionTree_tree.html
解説
詳細はGithubのJupyterNotebookを参照ください。
下記でおおまかな流れを解説します。
①ふつうにsklearnでDecisionTreeClassifierする
Titanicデータで決定木をつくります。データはKaggleのtrain.csvをつかいます。
https://www.kaggle.com/c/titanic/data
※TitanicはKaggleのチュートリアルとして非常に有名な問題で、Titanicの乗客の生死を予測する問題です。
train = pd.read_csv('../data/titanic.csv')
train = train.dropna().reset_index(drop=True)
train.drop(['PassengerId','Name','Ticket'],axis=1,inplace=True)
train['Cabin'] = train.Cabin.apply(lambda x: x[0])
train = pd.get_dummies(train,drop_first=True)
X = train.drop('Survived',axis=1)
y = train.Survived
clf = tree.DecisionTreeClassifier(max_depth=5)
clf.fit(X, y)
前処理をぶっ飛ばしてとりあえず決定木のモデルができました。
これを素直にGraphVizで描画すると、冒頭のダサい決定木が描けます。
②iGraphのグラフオブジェクトを作って、plotlyで描画する
iGraphはGraphVizやNetworkX同様、有名なグラフ図を描画してくれるパッケージです。
iGraphに決定木のノードのつながりを渡して、ノードとエッジの座標を作ってもらい、それをplotlyの3-Dscatterで描画する、という流れです。
そのために必要な情報は、さきほど作ったモデルから取得することができます。
↓各ノードが左側および右側でつながるノードを取得したり(今回の場合左がTrue、生存ですね)
children_left = clf.tree_.children_left
children_right = clf.tree_.children_right
↓各ノードのsamplesもとったりできる。
samples = clf.tree_.n_node_samples
上記結果①はそれで描画した結果になります。
この場合、各ノードの配置、エッジの距離はiGraphがいいかんじに配置してくれるので、座標に意味はありません。
ちなみに、ノードの色が生存or死亡の2値を表しています。
決定木の頂点だけ色を変えてみましたが、ちょうどそのノードを中心に分子構造みたいな形になりました。
なかなかフォトジェニックだしグリグリ動くインタラクティブでナイスな図ですが、GraphVizの決定木と比べて情報量が少なすぎますね。
③座標を自分で指定してplotlyで描画する
そこで、せっかく3軸あるので各座標に意味をもたせたのが上記結果②です。
X軸は各ノードのSamples、Y軸はジニ係数、Z軸は決定木内の深さにしました。
Xn = samples
Yn = impurities
Zn = calc_nodes_height(node_count, clf.max_depth, links)
実はSamplesはノード自体の大きさを(MinMaxスケーリングして)渡しているので、情報が重複していますが、まあ今回はこれで妥協します。。
結果①ではTrue, Falseのエッジが分かりませんでしたが、今回は自分で色分けすることで分かるようになりました。
更にsamplesとジニ係数をX軸、Y軸にしているので、X方向が大きく、かつY軸が0に近いノードが良さげなやつだと可視化できました。
ノードのつながりを目で追うのはGraphVizの方が圧倒的に見やすいですが・・・
ともかく、無事インスタ映えする決定木を描くことができました。
参考
- iGraph + plotlyは下記をすごく参考しました
https://plot.ly/python/3d-network-graph/ - DecisionTreeClassifierについては公式ドキュメントと、下記Githubがすごくためになりました。
http://scikit-learn.org/stable/auto_examples/tree/plot_unveil_tree_structure.html
https://github.com/ikegami-yukino/misc/blob/master/machinelearning/dt2code.py - plotly自体については下記Qiitaがいい感じにまとまってました。
https://qiita.com/inoory/items/12028af62018bf367722
終わりに
見栄えがよくてグリグリ動く決定木がかけるようになりましたが、いちいちグリグリ動かすのはめんどくさいので普段はGraphVizのダサい図を使います。