概要
GO(Gene Ontology)は、さまざまな生物の遺伝子(がコードするタンパク質)の機能に関する情報を統一化された語彙により表現したもの。
生物医学研究をする上でGOは重要な役割を果たしており、オミックス(ゲノミクス・トランスクリプトミクス・プロテオミクス・メタボロミクス等の総称)実験にて、生物の構造・機能・ダイナミクスを解明するため、一般的によく利用される。
本記事では、PythonのGO解析ツールである「goatools」を利用して、特定コントロール実験条件下での発現変動遺伝子のような遺伝子データセットにおいて統計的有意に多く含まれる機能情報を発見する手法である「GOenrichment解析」や「GOデータ可視化」について記載する。
実行環境設定
下記pipコマンドにより、goatoolsインストール
pip install goatools
GOデータの可視化を行う場合は、pygraphvizのインストールも必要
pip install pygraphviz
またpygraphvizはグラフ構造可視化ツール「graphviz」のラッパーライブラリのため、
本体のgraphvizのインストールが合わせて必要
- Ubuntuの場合、下記コマンドで簡単にインストールが可能
sudo apt install -y graphviz libgraphviz-dev pkg-config - Windowsの場合のインストール参考記事も記載しておく。
【Windows10】Graphvizのインストール
本記事では、Ubuntu20.04 & Python3.8.10 にて実行環境構築を実施
GO解析用データ取得
GO解析に必要なGOデータを定義したOBOファイルのダウンロードを行う。
以降の解析時には、ダウンロードファイルが常にカレントディレクトリに存在する必要があるので注意。
wget http://geneontology.org/ontology/go-basic.obo
wget http://www.geneontology.org/ontology/subsets/goslim_generic.obo
-
go-basic.obo
GOの機能や階層構造の定義等を記述したファイル -
goslim_generic.obo
go-basic.oboの縮小版であるGOslim(GO subsets)ファイル。
GOslimを利用することでGO解析結果が簡素化され、全体像の効率的な把握に役立つ。
GO解析結果の情報量が非常に多い場合に利用するが、個人的にあまり使う機会はない。
GOenrichment解析
シロイヌナズナ(Arabidopsis thaliana)のストレス環境条件下における発現変動遺伝子リストが手元にあり、これから機能解析を実行する状況を想定したGOenrichment解析デモについて記載する。
以降のデータ解析を自身で実行する場合は、GitHubに格納したデモ用データをダウンロードしてしてください。
GO解析デモ用データ一覧
-
at_stress_deg_list.txt
シロイヌナズナのストレス環境条件下での発現変動遺伝子(DEG:Differencial Expression Gene)IDリストファイル -
at_all_gene_list.txt
シロイヌナズナの全遺伝子IDリストファイル
(1.に記載された発現変動遺伝子IDを全て包括する) -
at_go_association.txt
シロイヌナズナの各遺伝子とGO情報の紐づきを記載したファイル
(2.に記載された遺伝子IDを含む)
GOenrichment解析実行コマンド
find_enrichment.pyにより「① 発現変動遺伝子」と「② 全遺伝子セット」のGOアノテーション割合を算出・比較することで、①のGOアノテーションが有意に多く観測されるかどうかの検定を行う。
複数回検定に起因する偽陽性(FDR:False Discovery Rate)の発生を制御するため、一般的によく利用されるBH(Benjamini-Hochberg)法による多重検定補正を行う設定で実行する。
find_enrichment.py ./data/at_stress_deg_list.txt ./data/at_all_gene_list.txt ./data/at_go_association.txt \
--pval=0.05 --method=fdr_bh --pval_field=fdr_bh \
--outfile=at_go_enrichment.tsv,at_go_enrichment.xlsx
コマンドオプション概要
「find_enrichment.py」の最初の引数には順番通りに3つのファイルを指定する。
- ターゲット遺伝子IDリストファイル
- 全遺伝子IDリストファイル
- 遺伝子IDとGO情報の紐づきを記載したファイル
| オプション | 内容 |
|---|---|
| pval | P-value閾値設定。閾値未満(P-value < 0.05)のGO機能を出力。 |
| method | 多重検定補正の手法を指定 |
| pval_field | 閾値未満(指定した多重検定補正済P-Value < 0.05)のGO機能を出力。 |
| outfile | 出力ファイル設定。tsv形式とxlsx形式の両方に対応。同時出力可。 |
出力結果
tsv/xlsxフォーマットで下記のように有意に多い(p_fdr_bhが充分に低い)発現変動遺伝子のGO機能が出力される。
出力結果からストレス環境要因に関連する機能(response to cold, abiotic stimulus, stress, etc...)が統計的有意に多く観測されていることが分かる。
# GO NS enrichment name ratio_in_study ratio_in_pop p_uncorrected depth study_count p_fdr_bh study_items
GO:0009409 BP e response to cold 21/168 241/31819 1.2953970111720632e-19 4 21 4.425076190163768e-16 AT1G09350, AT1G20440, AT1G20450, AT1G76180, AT2G15970, AT2G17840, AT2G17870, AT2G42530, AT2G45660, AT3G22840, AT3G50970, AT3G53990, AT4G03430, AT4G13850, AT4G24960, AT4G36020, AT5G15960, AT5G15970, AT5G20830, AT5G52310, AT5G58070
GO:0009628 BP e response to abiotic stimulus 36/168 1185/31819 1.1559257428995517e-17 2 36 1.703993929100776e-14 AT1G01470, AT1G09350, AT1G20440, AT1G20450, AT1G76180, AT1G77120, AT2G15970, AT2G17840, AT2G17870, AT2G21620, AT2G39800, AT2G42530, AT2G45660, AT3G02230, AT3G03250, AT3G14440, AT3G22840, AT3G50970, AT3G53990, AT3G55610, AT4G03430, AT4G11600, AT4G13850, AT4G15480, AT4G24960, AT4G27410, AT4G36020, AT4G38580, AT5G15650, AT5G15960, AT5G15970, AT5G20830, AT5G40390, AT5G52310, AT5G58070, AT5G62640
GO:0006950 BP e response to stress 45/168 1943/31819 1.4964817878519694e-17 2 45 1.703993929100776e-14 AT1G01470, AT1G02820, AT1G09350, AT1G20440, AT1G20450, AT1G76180, AT1G77120, AT2G02100, AT2G15970, AT2G17840, AT2G17870, AT2G21620, AT2G22080, AT2G39800, AT2G42530, AT2G45660, AT3G02230, AT3G03250, AT3G05660, AT3G14440, AT3G22840, AT3G50970, AT3G53990, AT3G55610, AT4G03430, AT4G11600, AT4G12000, AT4G12470, AT4G13850, AT4G24220, AT4G24960, AT4G27410, AT4G36020, AT4G38580, AT5G13160, AT5G15650, AT5G15960, AT5G15970, AT5G19875, AT5G20830, AT5G25110, AT5G40390, AT5G47120, AT5G52310, AT5G58070
GO:0009266 BP e response to temperature stimulus 22/168 358/31819 3.02332975446427e-17 3 22 2.581923610312487e-14 AT1G09350, AT1G20440, AT1G20450, AT1G76180, AT2G15970, AT2G17840, AT2G17870, AT2G42530, AT2G45660, AT3G22840, AT3G50970, AT3G53990, AT4G03430, AT4G13850, AT4G24960, AT4G36020, AT4G38580, AT5G15960, AT5G15970, AT5G20830, AT5G52310, AT5G58070
GO:0009415 BP e response to water 17/168 178/31819 9.36720486046985e-17 4 17 6.399674360673002e-14 AT1G01470, AT1G20440, AT1G20450, AT1G76180, AT2G15970, AT2G17840, AT2G21620, AT2G39800, AT3G14440, AT3G50970, AT4G24960, AT4G27410, AT5G15960, AT5G15970, AT5G20830, AT5G40390, AT5G52310
〜〜〜(以下省略)〜〜〜
GOenrichment解析実行コマンド(GOslim)
map_to_slim.pyにより「at_go_association.txt」の各遺伝子とGO情報の紐づきを縮小(GOslim)化する。
縮小(GOslim)化したファイルを利用して、前述と同様にGOenrichment解析を実行する。
map_to_slim.py --association_file=./data/at_go_association.txt go-basic.obo goslim_generic.obo > ./data/at_go_association_slim.txt
find_enrichment.py ./data/at_stress_deg_list.txt ./data/at_all_gene_list.txt ./data/at_go_association_slim.txt \
--pval=0.05 --method=fdr_bh --pval_field=fdr_bh \
--outfile=at_go_enrichment_slim.tsv,at_go_enrichment_slim.xlsx
出力結果(GOslim)
GOslimを利用した出力結果において、ストレス環境要因に関連する機能は2つ(response to stress, stimulus)のみとなり、GOslimを利用しない場合と比較してGO情報量が大幅に削減された。
今回のサンプル程度のGO情報量であれば、GOslimは利用しないほうがデータ解析に適していると考えられる。
# GO NS enrichment name ratio_in_study ratio_in_pop p_uncorrected depth study_count p_fdr_bh study_items
GO:0006950 BP e response to stress 45/168 1943/31819 1.4964817878519694e-17 2 45 1.137326158767497e-15 AT1G01470, AT1G02820, AT1G09350, AT1G20440, AT1G20450, AT1G76180, AT1G77120, AT2G02100, AT2G15970, AT2G17840, AT2G17870, AT2G21620, AT2G22080, AT2G39800, AT2G42530, AT2G45660, AT3G02230, AT3G03250, AT3G05660, AT3G14440, AT3G22840, AT3G50970, AT3G53990, AT3G55610, AT4G03430, AT4G11600, AT4G12000, AT4G12470, AT4G13850, AT4G24220, AT4G24960, AT4G27410, AT4G36020, AT4G38580, AT5G13160, AT5G15650, AT5G15960, AT5G15970, AT5G19875, AT5G20830, AT5G25110, AT5G40390, AT5G47120, AT5G52310, AT5G58070
GO:0050896 BP e response to stimulus 45/168 1943/31819 1.4964817878519694e-17 1 45 1.137326158767497e-15 AT1G01470, AT1G02820, AT1G09350, AT1G20440, AT1G20450, AT1G76180, AT1G77120, AT2G02100, AT2G15970, AT2G17840, AT2G17870, AT2G21620, AT2G22080, AT2G39800, AT2G42530, AT2G45660, AT3G02230, AT3G03250, AT3G05660, AT3G14440, AT3G22840, AT3G50970, AT3G53990, AT3G55610, AT4G03430, AT4G11600, AT4G12000, AT4G12470, AT4G13850, AT4G24220, AT4G24960, AT4G27410, AT4G36020, AT4G38580, AT5G13160, AT5G15650, AT5G15960, AT5G15970, AT5G19875, AT5G20830, AT5G25110, AT5G40390, AT5G47120, AT5G52310, AT5G58070
GO:0016020 CC e membrane 31/168 1892/31819 1.6725763607803712e-08 2 31 3.846925629794854e-07 AT1G11960, AT1G30360, AT1G58360, AT1G76180, AT1G77120, AT2G02100, AT2G15970, AT2G17840, AT2G21130, AT2G23120, AT2G38465, AT2G45820, AT3G02230, AT3G03250, AT3G15730, AT3G23810, AT3G27210, AT3G45600, AT3G57340, AT4G02710, AT4G05520, AT4G11600, AT4G20890, AT4G23630, AT4G27520, AT4G35300, AT4G38580, AT5G15970, AT5G41600, AT5G57110, AT5G58070
GO:0005886 CC e plasma membrane 31/168 1892/31819 1.6725763607803712e-08 3 31 3.846925629794854e-07 AT1G11960, AT1G30360, AT1G58360, AT1G76180, AT1G77120, AT2G02100, AT2G15970, AT2G17840, AT2G21130, AT2G23120, AT2G38465, AT2G45820, AT3G02230, AT3G03250, AT3G15730, AT3G23810, AT3G27210, AT3G45600, AT3G57340, AT4G02710, AT4G05520, AT4G11600, AT4G20890, AT4G23630, AT4G27520, AT4G35300, AT4G38580, AT5G15970, AT5G41600, AT5G57110, AT5G58070
GO:0110165 CC e cellular anatomical entity 83/168 9528/31819 1.670443498419094e-07 1 83 2.561346697575944e-06 AT1G08880, AT1G11960, AT1G14580, AT1G20450, AT1G21670, AT1G30360, AT1G47710, AT1G53910, AT1G55110, AT1G58360, AT1G61730, AT1G67310, AT1G67360, AT1G73630, AT1G76180, AT1G77120, AT2G02100, AT2G15970, AT2G17840, AT2G17870, AT2G20370, AT2G21130, AT2G22080, AT2G22500, AT2G22860, AT2G23120, AT2G23760, AT2G37520, AT2G38465, AT2G39800, AT2G42530, AT2G45660, AT2G45820, AT3G02230, AT3G02750, AT3G03250, AT3G05660, AT3G15730, AT3G23810, AT3G24520, AT3G24840, AT3G25140, AT3G27210, AT3G29360, AT3G45600, AT3G45960, AT3G49220, AT3G53620, AT3G55610, AT3G55760, AT3G57010, AT3G57340, AT3G59820, AT4G02710, AT4G03430, AT4G05520, AT4G11600, AT4G13850, AT4G20880, AT4G20890, AT4G22590, AT4G23630, AT4G27410, AT4G27520, AT4G32400, AT4G34740, AT4G35300, AT4G38580, AT5G11420, AT5G15190, AT5G15650, AT5G15970, AT5G16010, AT5G17490, AT5G40390, AT5G41600, AT5G47120, AT5G56630, AT5G57110, AT5G57660, AT5G58070, AT5G62190, AT5G62640
〜〜〜(以下省略)〜〜〜
その他TIPS
GOカテゴリー(BP/CC/MF)毎に結果を出力
--nsオプションを利用することで各GOカテゴリー毎に結果の出力が可能
3つのカテゴリー(BP:Biological Process, CC:Cellular Component, MF:Molecular Function)の詳細はこちらを参照
for category in BP CC MF
do
find_enrichment.py ./data/at_stress_deg_list.txt ./data/at_all_gene_list.txt ./data/at_go_association.txt \
--pval=0.05 --method=fdr_bh --pval_field=fdr_bh --ns=$category \
--outfile=at_go_enrichment_$category.tsv,at_go_enrichment_$category.xlsx
done
Pvalueの大小に関わらず全結果を出力
--pval=-1を指定することで全結果を出力することが可能
find_enrichment.py ./data/at_stress_deg_list.txt ./data/at_all_gene_list.txt ./data/at_go_association.txt \
--pval=-1 --method=fdr_bh --pval_field=fdr_bh \
--outfile=at_go_enrichment_all.tsv,at_go_enrichment_all.xlsx
GOデータ可視化
plot_go_term.pyを利用した単体GO定義の親子関係可視化、及び
go_plot.pyを利用した複数GO定義の関係可視化の手法について記載する。
単体GO定義の親子関係可視化
plot_go_term.pyにより指定したGOの親子(is-a)関係を可視化した画像を出力可能(対応フォーマット:pdf, svg, png, jpg)
※ 指定したGOの階層レベルが高い場合は、情報量が多くなり画像表示出来ない場合があるので注意
plot_go_term.py --term=GO:0009415 --output=response_to_water.jpg
親または子の関係を除外して可視化した画像出力も可能
plot_go_term.py --term=GO:0009415 --output=response_to_water_noparents.jpg --disable-draw-parents
plot_go_term.py --term=GO:0009415 --output=response_to_water_nochildren.jpg --disable-draw-children
response_to_water_noparents.jpg

response_to_water_nochildren.jpg

複数GO定義の関係可視化
go_plots.pyにより指定したGO定義間における親(root)までの関係(is-a, part-of)を可視化した画像を出力可能
※ GO定義のRelation(関係)の詳細はこちらを参照
go_plot.py GO:0003304 GO:0061371 --outfile=multi_plot.png
-r (--relationship)オプションにより、橙破線のpart-of関係を含むGO情報も出力される。
またGOにカラー情報(#ff0000=赤)を追記することで、対象GOの色を変更することができる。
go_plot.py GO:0003304 GO:0061371#ff0000 -r --outfile=multi_plot_all_relation.png
GOenrichment結果のGO定義関係可視化
例:「GOカテゴリー=BP」に属する上位(Pvalue昇順)10個のGO機能の可視化
# BPを対象としたGOenrichment結果のトップ10のGO記載のファイル作成
grep GO: at_go_enrichment.tsv | grep BP | head -n 10 | cut -f 1 > go_enrichment_list.txt
go_plot.py --go_file=go_enrichment_list.txt --outfile=plot_go_enrichment_BP.png
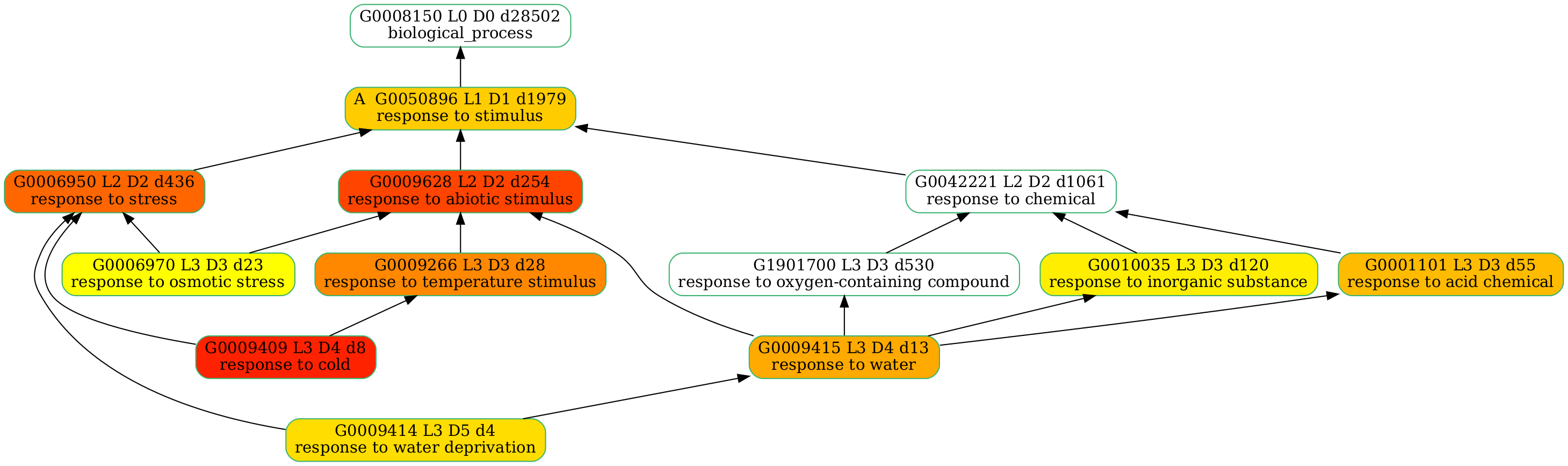
また下記のような色情報を含むGO定義ファイルを入力とすることで、発現変動遺伝子とGO機能の関連性の強さ(Pvalueの大小関係)を段階色で表現するようなことも可能となる。
追記:GO定義関係可視化の見栄え改良に関する別記事を書きました。
# ff2200 GO:0009409
# ff4400 GO:0009628
# ff6600 GO:0006950
# ff8800 GO:0009266
# ffaa00 GO:0009415
# ffbb00 GO:0001101
# ffcc00 GO:0050896
# ffdd00 GO:0009414
# ffee00 GO:0010035
# ffff00 GO:0006970
echo -e "#ff2200\tGO:0009409\n#ff4400\tGO:0009628\n#ff6600\tGO:0006950\n#ff8800\tGO:0009266\n#ffaa00\tGO:0009415\n#ffbb00\tGO:0001101\n#ffcc00\tGO:0050896\n#ffdd00\tGO:0009414\n#ffee00\tGO:0010035\n#ffff00\tGO:0006970" \
> go_enrichment_color_list.txt
go_plot.py --go_file=go_enrichment_color_list.txt --outfile=plot_color_go_enrichment_BP.png
最後に
本記事で紹介したgoatoolsを利用することでGOenrichment解析やGOデータ可視化を手元で手軽に実行することができた。
しかし、近年はWebベースのGO解析ツールも充実してきているため、「自動化されたデータ解析パイプライン構築」や「繰り返しの大量データ解析」等の必要性がない状況では、agriGOやg:ProfilerのようなWebベースのGO解析ツールの利用で十分に解析が可能である。
目的に応じてGO解析のコマンドツールとWebツールを使い分けると良さそう。
参考
GOATOOLS: A Python library for Gene Ontology analyses - scientific reports - goatools論文
goatools - GibHub - goatoolsのGitHubサイト
GENE ONTOROGY Unifying Biology - GO管理団体の公式サイト
GO解析 biopapyrus - GO解析の詳細が記載されたサイト
その他GO解析ツール
GOstats - RによるGO解析ツール。Rユーザーはgoatoolsの代替で利用できる。
GO解析Webツール
agriGO - 農学系の生物が充実したGO解析ツール。UI・出力結果ともに分かりやすい。
g:Profiler - 遺伝子ID変換ツール等も提供しているGO解析ツール。UI・出力結果ともに分かりやすい。
DAVID - 昔から利用されてきた定番GO解析ツール。しかし、UIが残念でデータ更新が遅い印象。