グラフ構造を深層学習する PyG (PyTorch Geometric) を Google Colaboratory 上で使ってみました。今回は、MetaPath2Vec を使うことがテーマです。Node2Vecとの比較も行ないました。
PyG (PyTorch Geometric) インストール
PyG (PyTorch Geometric) のレポジトリは https://github.com/pyg-team/pytorch_geometric にあります。また、コードはチュートリアルドキュメント https://pytorch-geometric.readthedocs.io/en/latest/index.html を参考にしています。
import os
import torch
torch.manual_seed(0)
os.environ['TORCH'] = torch.__version__
print(torch.__version__)
!pip install -q torch-scatter -f https://data.pyg.org/whl/torch-${TORCH}.html
!pip install -q torch-sparse -f https://data.pyg.org/whl/torch-${TORCH}.html
!pip install -q git+https://github.com/pyg-team/pytorch_geometric.git
import torch_geometric
1.12.0+cu113
[K |████████████████████████████████| 7.9 MB 56.3 MB/s
[K |████████████████████████████████| 3.5 MB 27.1 MB/s
[?25h Building wheel for torch-geometric (setup.py) ... [?25l[?25hdone
device = 'cuda' if torch.cuda.is_available() else 'cpu'
データセットの自作
今回もデータセットを自作します。自作する理由は、結果の良し悪しを自分の目で判断しやすくするためです。
小さなデータで説明
A, B, C の3タイプのノード(頂点)があるものとします。
import numpy as np
num = 3
A = np.random.rand(num * 2)
A.sort()
B = np.random.rand(num * 4)
B.sort()
C = np.random.rand(num)
C.sort()
次のような基準を満たすときに、異なるタイプのノード間にのみエッジ(辺)が作られるものとします。
def get_edges(top, X, Y):
edges = []
for i1, n1 in enumerate(X):
for i2 in np.argsort([np.abs(n1 - n2) for n2 in Y])[:top]:
edge = [i1, i2]
if edge not in edges:
edges.append(edge)
for i1, n1 in enumerate(Y):
for i2 in np.argsort([np.abs(n1 - n2) for n2 in X])[:top]:
edge = [i2, i1]
if edge not in edges:
edges.append(edge)
edges.sort()
return edges
このようにして、タイプAのノードとタイプBのノードの間、タイプBのノードとタイプCのノードの間にのみエッジが作られるものとします。タイプAのノードとタイプCのノードの間にはエッジは作りません。また、同じタイプのノード(タイプA同士、タイプB同士、タイプC同士)間にはエッジは作りません。
edges_AB = get_edges(2, A, B)
edges_BC = get_edges(2, B, C)
edges_BA = get_edges(2, B, A)
edges_CB = get_edges(2, C, B)
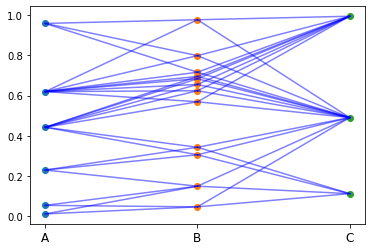
形成されるネットワーク(グラフ構造)は、たとえば次のような感じです。
import matplotlib.pyplot as plt
plt.scatter([0 for _ in range(len(A))], A)
plt.scatter([1 for _ in range(len(B))], B)
plt.scatter([2 for _ in range(len(C))], C)
for edge in edges_AB:
plt.plot([0, 1], [A[edge[0]], B[edge[1]]], color="b", alpha=0.5)
for edge in edges_BC:
plt.plot([1, 2], [B[edge[0]], C[edge[1]]], color="b", alpha=0.5)
plt.xticks([0, 1, 2], ["A", "B", "C"], color="k", fontsize=12, rotation=0)
plt.show()
話をややこしくしてしまうかもしれませんが、AC間のエッジの引き方も、説明しておきましょう。ここでは、AとCが1個のBを介して間接的につながっている場合に、AC間にエッジを引くことにします。
edges_AC = []
for x in edges_AB:
for y in edges_BC:
if x[1] == y[0]:
if [x[0], y[1]] not in edges_AC:
edges_AC.append([x[0], y[1]])
そうすると、AC間のエッジは次のようになります。ただし、話をややこしくしてしまうかもしれませんが、このエッジは学習には用いません。学習結果を確認するときにのみ使う予定です。
import matplotlib.pyplot as plt
plt.scatter([0 for _ in range(len(A))], A)
plt.scatter([2 for _ in range(len(C))], C)
for edge in edges_AC:
plt.plot([0, 2], [A[edge[0]], C[edge[1]]], color="b", alpha=0.5)
plt.xticks([0, 2], ["A", "C"], color="k", fontsize=12, rotation=0)
plt.show()
実際に使うデータ
以上、データ構造を説明するため、小さなサイズのデータを用いました。ここから先は、実際に使うデータを作成します。
import numpy as np
num = 100
A = np.random.rand(num * 2)
A.sort()
B = np.random.rand(num * 4)
B.sort()
C = np.random.rand(num)
C.sort()
edges_AB = get_edges(5, A, B)
edges_BC = get_edges(5, B, C)
edges_BA = get_edges(5, B, A)
edges_CB = get_edges(5, C, B)
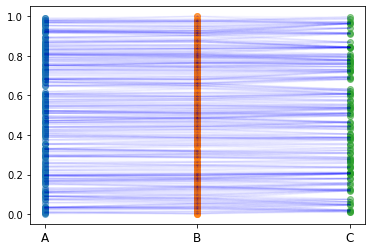
このようなネットワーク(グラフ構造)になります。
import matplotlib.pyplot as plt
plt.scatter([0 for _ in range(len(A))], A, alpha=0.5)
plt.scatter([1 for _ in range(len(B))], B, alpha=0.5)
plt.scatter([2 for _ in range(len(C))], C, alpha=0.5)
for edge in edges_AB:
plt.plot([0, 1], [A[edge[0]], B[edge[1]]], color="b", alpha=0.01)
for edge in edges_BC:
plt.plot([1, 2], [B[edge[0]], C[edge[1]]], color="b", alpha=0.01)
plt.xticks([0, 1, 2], ["A", "B", "C"], color="k", fontsize=12, rotation=0)
plt.show()
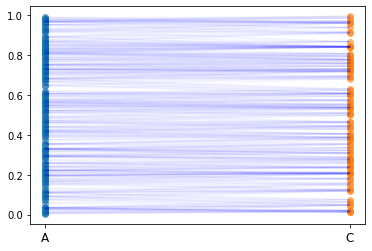
学習に用いない、AC間のエッジも計算しておきましょう。
edges_AC = []
for x in edges_AB:
for y in edges_BC:
if x[1] == y[0]:
if [x[0], y[1]] not in edges_AC:
edges_AC.append([x[0], y[1]])
こんな感じです。
import matplotlib.pyplot as plt
plt.scatter([0 for _ in range(len(A))], A, alpha=0.5)
plt.scatter([2 for _ in range(len(C))], C, alpha=0.5)
for edge in edges_AC:
plt.plot([0, 2], [A[edge[0]], C[edge[1]]], color="b", alpha=0.01)
plt.xticks([0, 2], ["A", "C"], color="k", fontsize=12, rotation=0)
plt.show()
HeteroData
作成したネットワークを、HeteroData クラスで実装しましょう。
data = torch_geometric.data.HeteroData()
data['A'].y = torch.tensor(np.where(A > 0.5, 1, 0))
data['A'].y_index = torch.argsort(torch.tensor(A))
data['A'].num_nodes = len(A)
data['B'].num_nodes = len(B)
data['B'].y_index = torch.argsort(torch.tensor(B)) ##
data['C'].y = torch.tensor(C)
data['C'].y_index = torch.argsort(torch.tensor(C))
data['C'].num_nodes = len(C)
data[('A', 'A2B', 'B')].edge_index = torch.tensor(edges_AB).T
data[('B', 'B2C', 'C')].edge_index = torch.tensor(edges_BC).T
data[('B', 'B2A', 'A')].edge_index = torch.tensor(edges_BA).T
data[('C', 'C2B', 'B')].edge_index = torch.tensor(edges_CB).T
data
HeteroData(
[1mA[0m={
y=[200],
y_index=[200],
num_nodes=200
},
[1mB[0m={
num_nodes=400,
y_index=[400]
},
[1mC[0m={
y=[100],
y_index=[100],
num_nodes=100
},
[1m(A, A2B, B)[0m={ edge_index=[2, 2046] },
[1m(B, B2C, C)[0m={ edge_index=[2, 2003] },
[1m(B, B2A, A)[0m={ edge_index=[2, 2046] },
[1m(C, C2B, B)[0m={ edge_index=[2, 2003] }
)
MetaPath2Vec (embedding_dim = 2)
次のようにして MetaPath2Vec のモデルを実装しましょう。まずは embedding_dim = 2 とします。
from torch_geometric.nn import MetaPath2Vec
embedding_dim = 2
metapath = [
('A', 'A2B', 'B'),
('B', 'B2C', 'C'),
('B', 'B2A', 'A'),
('C', 'C2B', 'B'),
]
model = MetaPath2Vec(data.edge_index_dict,
embedding_dim=embedding_dim,
metapath=metapath,
walk_length=4,
context_size=3,
walks_per_node=3,
num_negative_samples=1,
sparse=True
).to(device)
トレーニングとテストのコードです。
def train(epoch, log_steps=500, eval_steps=1000):
model.train()
total_loss = 0
for i, (pos_rw, neg_rw) in enumerate(loader):
optimizer.zero_grad()
loss = model.loss(pos_rw.to(device), neg_rw.to(device))
loss.backward()
optimizer.step()
total_loss += loss.item()
if (i + 1) % log_steps == 0:
print((f'Epoch: {epoch}, Step: {i + 1:05d}/{len(loader)}, '
f'Loss: {total_loss / log_steps:.4f}'))
total_loss = 0
if (i + 1) % eval_steps == 0:
acc = test()
print((f'Epoch: {epoch}, Step: {i + 1:05d}/{len(loader)}, '
f'Acc: {acc:.4f}'))
return total_loss
@torch.no_grad()
def test(train_ratio=0.1):
model.eval()
z = model('A', batch=data.y_index_dict['A'].to(device))
y = data.y_dict['A']
perm = torch.randperm(z.size(0))
train_perm = perm[:int(z.size(0) * train_ratio)]
test_perm = perm[int(z.size(0) * train_ratio):]
return model.test(z[train_perm], y[train_perm], z[test_perm],
y[test_perm], max_iter=150)
モデルのローダーとオプティマイザを初期化します。
loader = model.loader(batch_size=128, shuffle=True, num_workers=2)
optimizer = torch.optim.SparseAdam(list(model.parameters()), lr=0.01)
学習開始。ベストモデルを best_model として保存します。
import copy
loss_hist = []
acc_hist = []
best_score = None
for epoch in range(600):
loss = train(epoch)
acc = test()
loss_hist.append(loss)
acc_hist.append(acc)
if best_score is None or best_score < acc:
best_model = copy.deepcopy(model)
best_score = acc
print(f'Epoch: {epoch}, Accuracy: {acc:.4f}, Loss: {loss: .6f}')
Epoch: 0, Accuracy: 0.5278, Loss: 3.505347
Epoch: 4, Accuracy: 0.5333, Loss: 3.430402
Epoch: 6, Accuracy: 0.5556, Loss: 3.333481
Epoch: 50, Accuracy: 0.5667, Loss: 2.891399
Epoch: 77, Accuracy: 0.6056, Loss: 2.772557
Epoch: 81, Accuracy: 0.6500, Loss: 2.747386
Epoch: 95, Accuracy: 0.6778, Loss: 2.678413
Epoch: 99, Accuracy: 0.6833, Loss: 2.662748
Epoch: 120, Accuracy: 0.7278, Loss: 2.539922
Epoch: 140, Accuracy: 0.7444, Loss: 2.418053
Epoch: 145, Accuracy: 0.7556, Loss: 2.350473
Epoch: 148, Accuracy: 0.7722, Loss: 2.356267
Epoch: 151, Accuracy: 0.7889, Loss: 2.363546
Epoch: 155, Accuracy: 0.7944, Loss: 2.343060
Epoch: 163, Accuracy: 0.8000, Loss: 2.264058
Epoch: 167, Accuracy: 0.8056, Loss: 2.252009
Epoch: 173, Accuracy: 0.8278, Loss: 2.263199
Epoch: 180, Accuracy: 0.8444, Loss: 2.254319
Epoch: 185, Accuracy: 0.8500, Loss: 2.201037
Epoch: 195, Accuracy: 0.8722, Loss: 2.203842
Epoch: 207, Accuracy: 0.8778, Loss: 2.179795
Epoch: 220, Accuracy: 0.8944, Loss: 2.183385
Epoch: 273, Accuracy: 0.9056, Loss: 2.128915
Epoch: 315, Accuracy: 0.9111, Loss: 2.125498
Epoch: 335, Accuracy: 0.9278, Loss: 2.124234
Epoch: 375, Accuracy: 0.9500, Loss: 2.057485
Epoch: 398, Accuracy: 0.9556, Loss: 2.043905
Epoch: 400, Accuracy: 0.9611, Loss: 2.095988
Epoch: 410, Accuracy: 0.9667, Loss: 2.038597
Epoch: 419, Accuracy: 0.9833, Loss: 2.061875
Epoch: 433, Accuracy: 0.9889, Loss: 2.090283
Epoch: 457, Accuracy: 0.9944, Loss: 2.028480
Epoch: 484, Accuracy: 1.0000, Loss: 2.061711
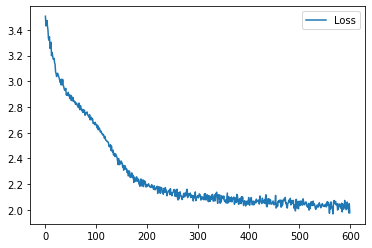
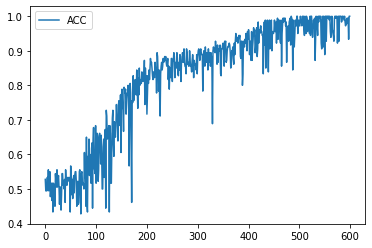
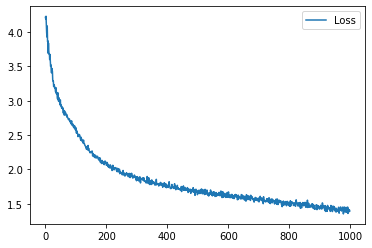
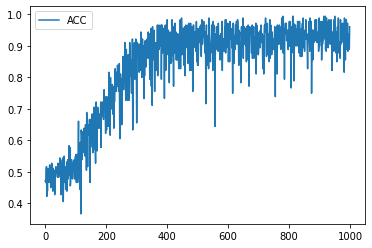
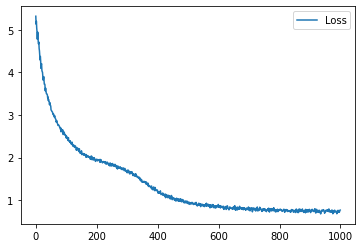
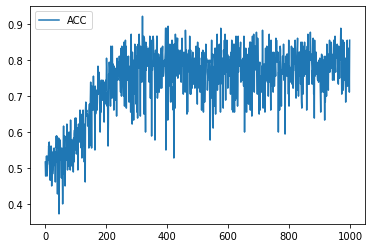
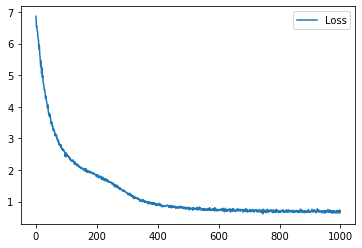
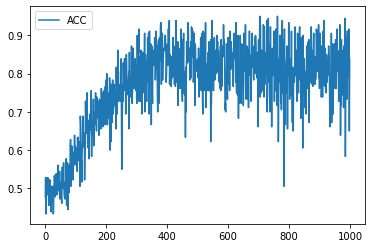
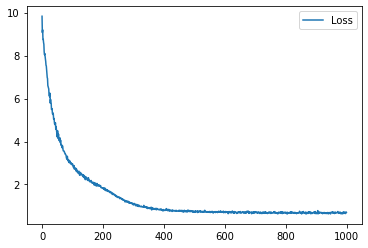
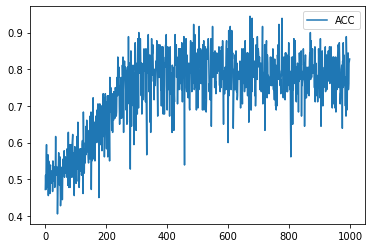
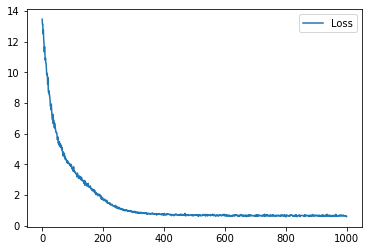
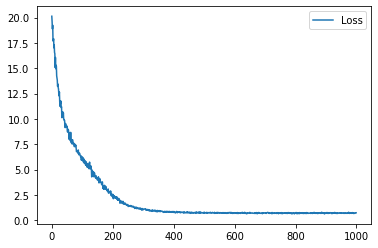
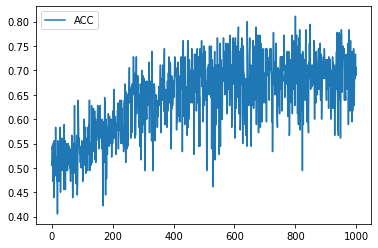
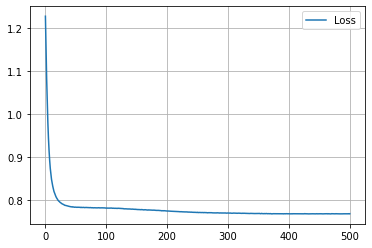
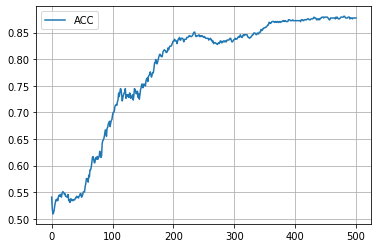
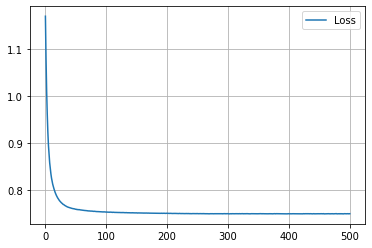
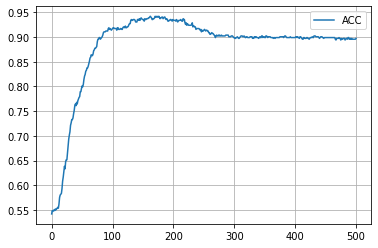
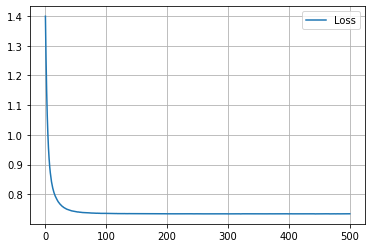
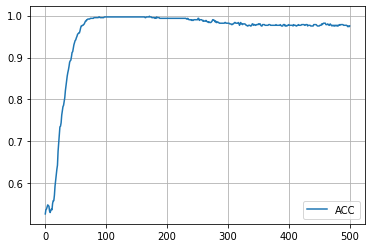
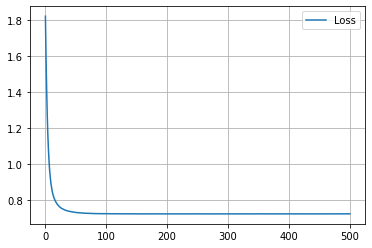
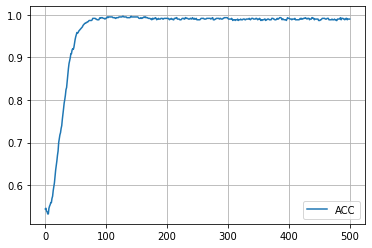
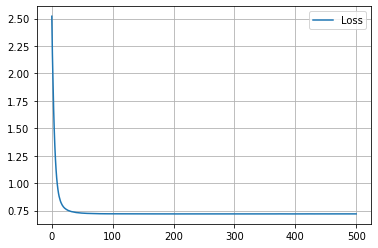
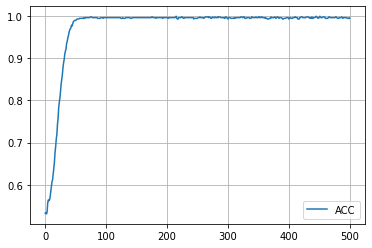
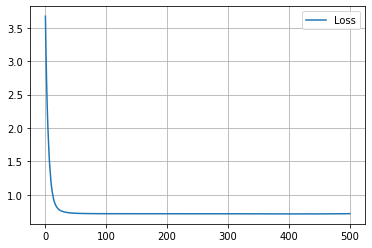
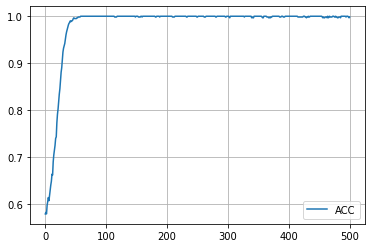
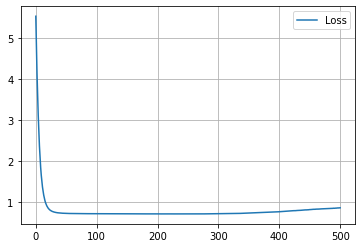
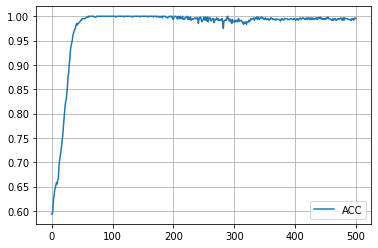
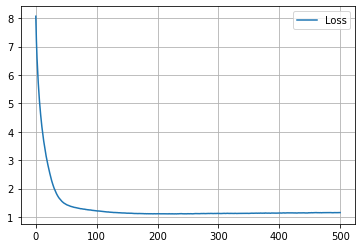
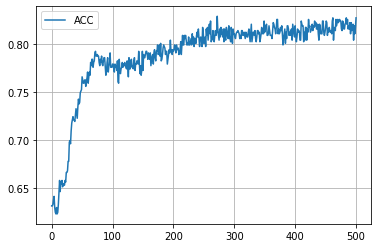
学習履歴は次のようになりました。
plt.plot(loss_hist, label="Loss")
plt.legend()
plt.show()
plt.plot(acc_hist, label="ACC")
plt.legend()
plt.show()
各々のノードを潜在空間上にマッピングします。
z_A = best_model('A', batch=data.y_index_dict['A'].to(device)).detach().cpu().numpy()
z_B = best_model('B', batch=data.y_index_dict['B'].to(device)).detach().cpu().numpy()
z_C = best_model('C', batch=data.y_index_dict['C'].to(device)).detach().cpu().numpy()
潜在空間の次元が2以上のときは、可視化のため Isomap などで次元削減します。
from sklearn.manifold import Isomap
embedding_model = Isomap()
z_A_2d = embedding_model.fit_transform(z_A)
z_B_2d = embedding_model.transform(z_B)
z_C_2d = embedding_model.transform(z_C)
潜在空間が2次元のときはそのまま使います。
z_A_2d = z_A
z_B_2d = z_B
z_C_2d = z_C
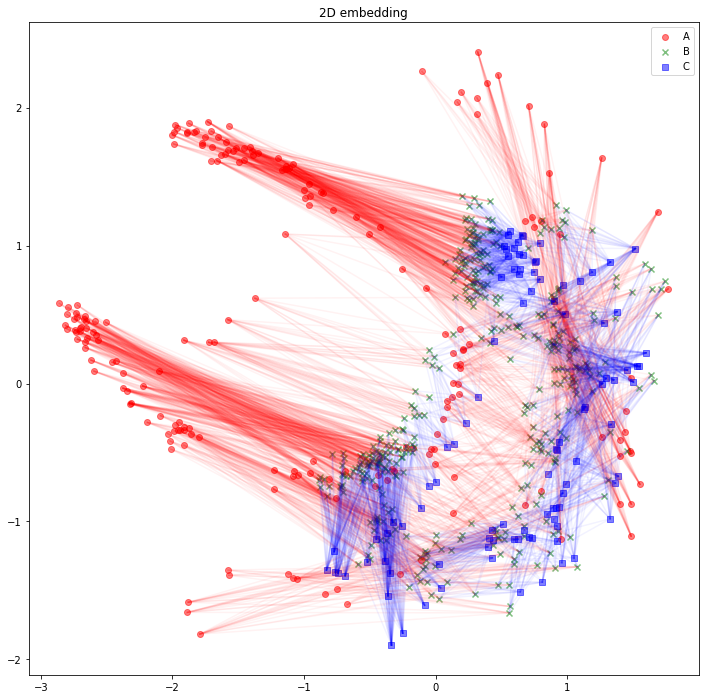
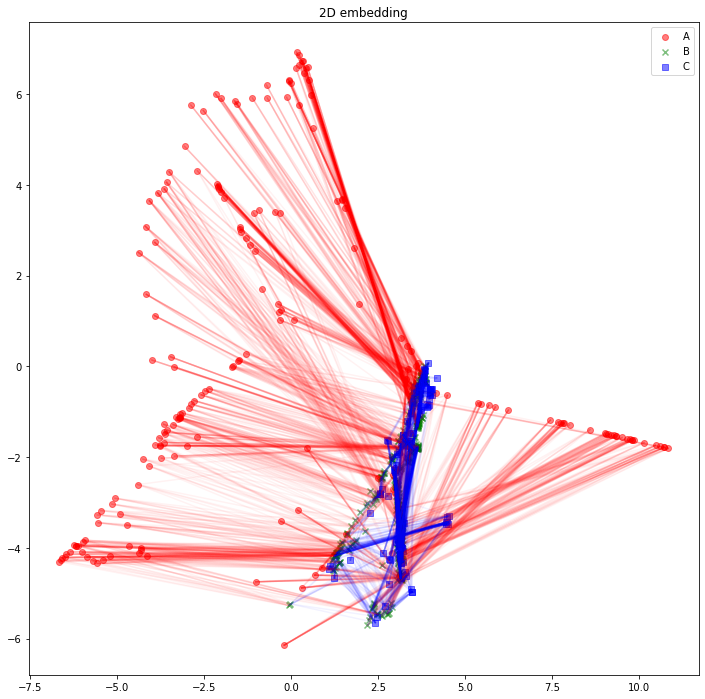
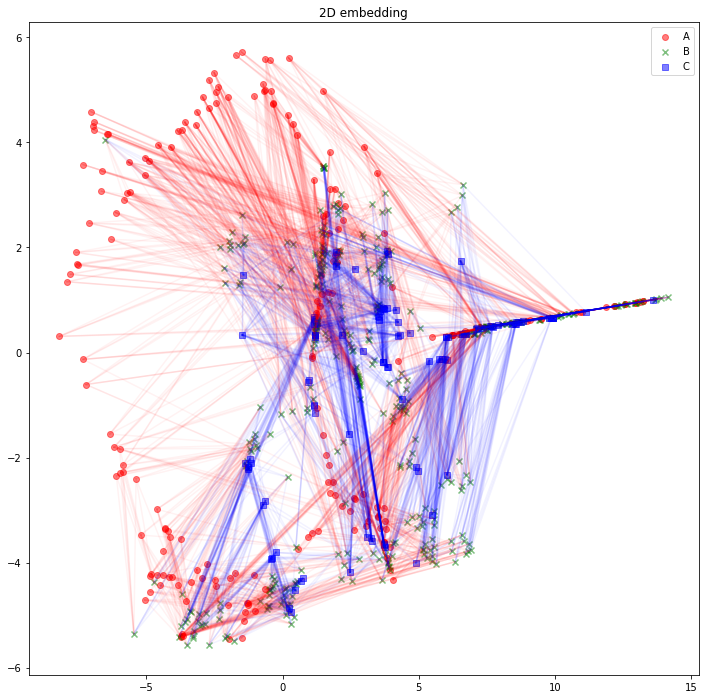
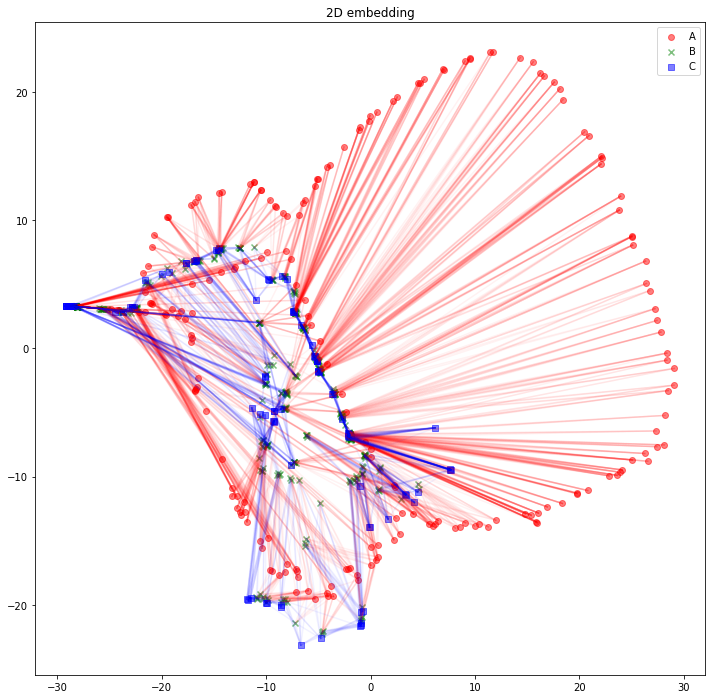
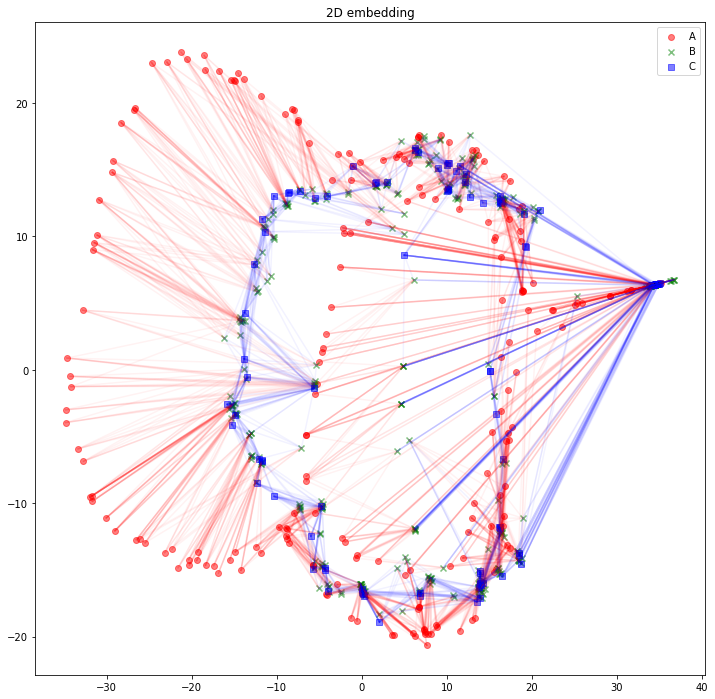
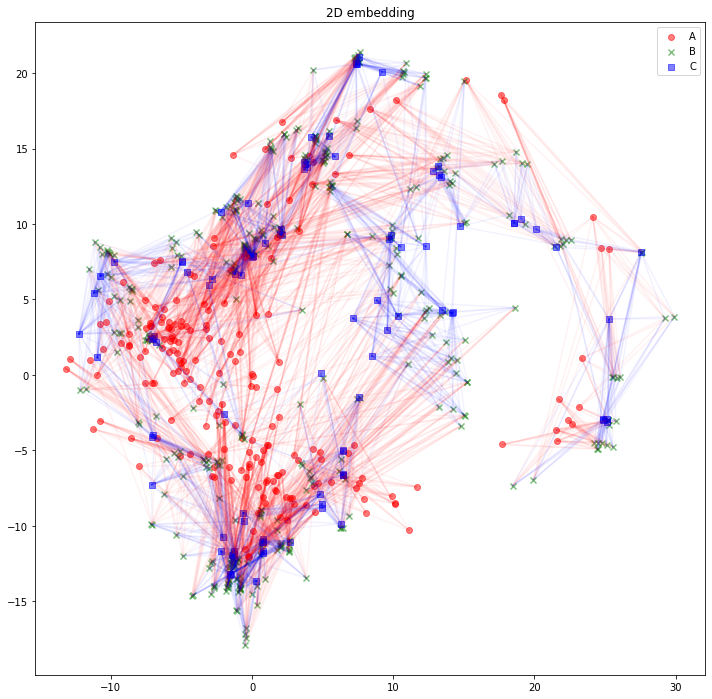
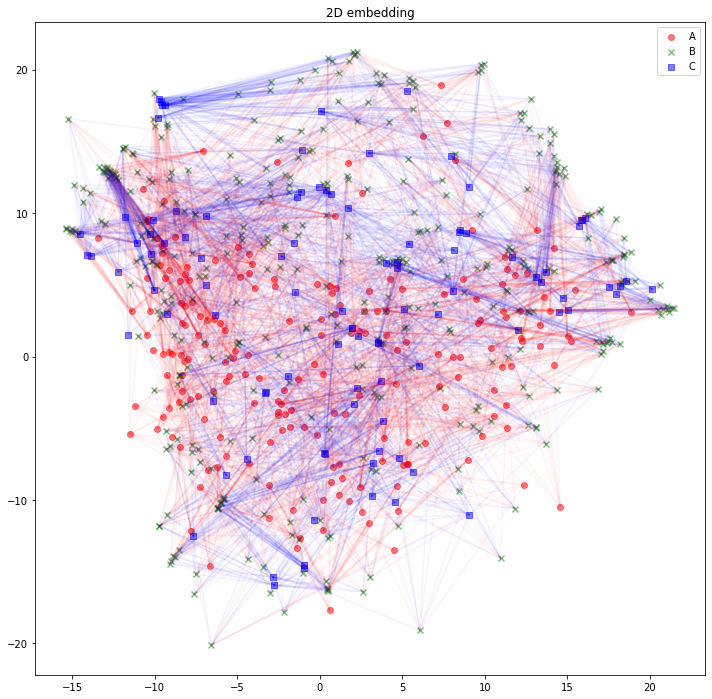
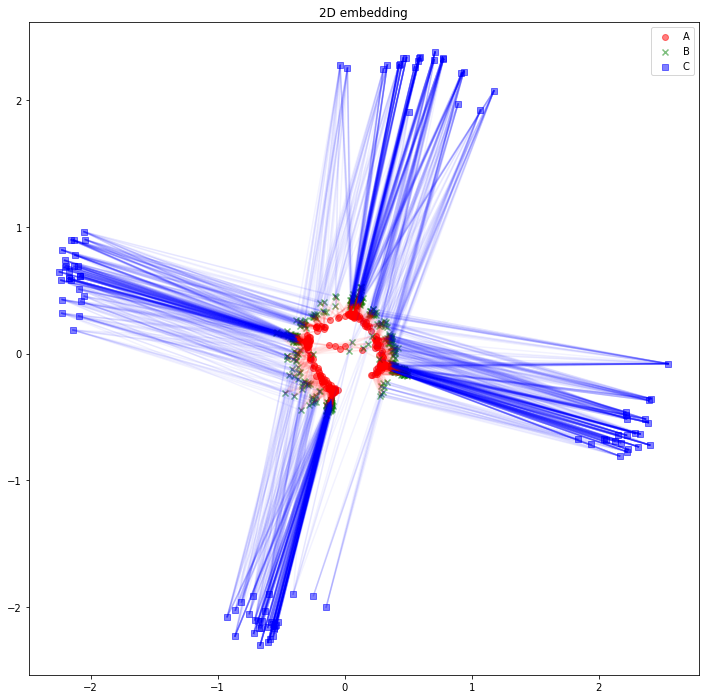
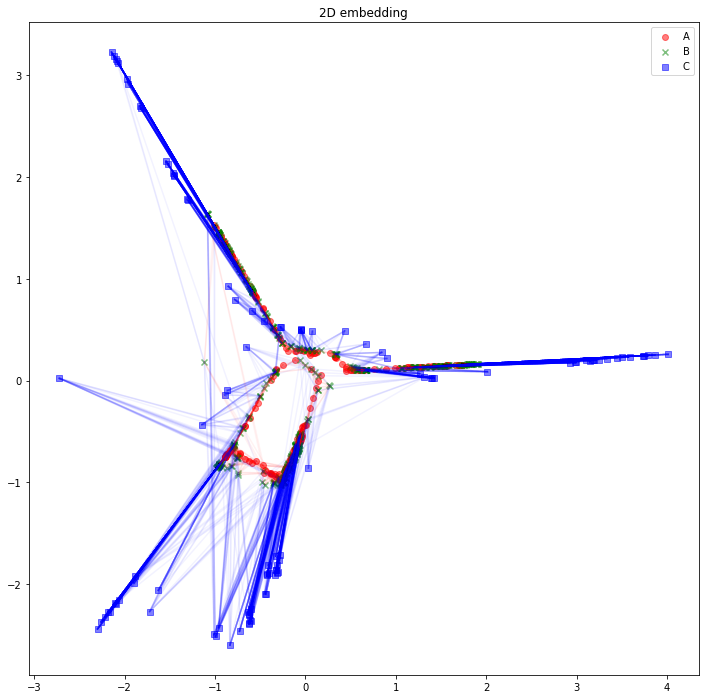
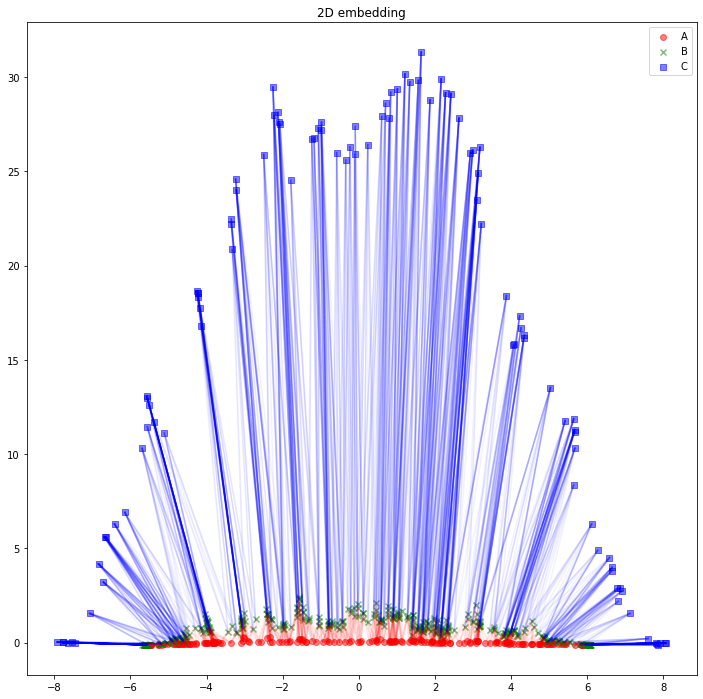
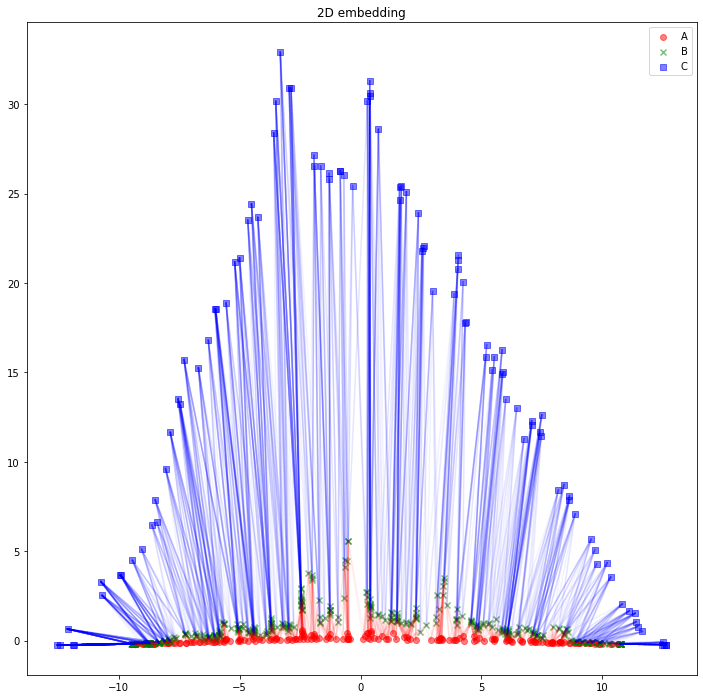
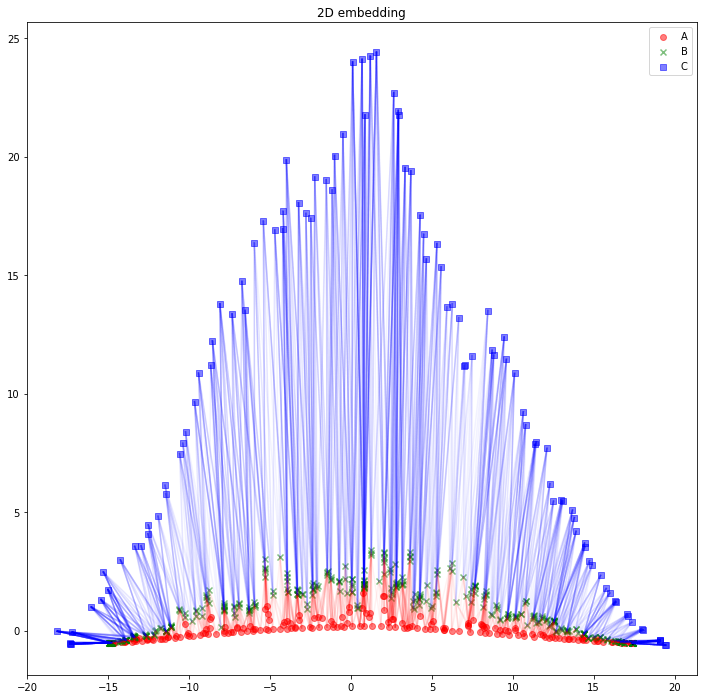
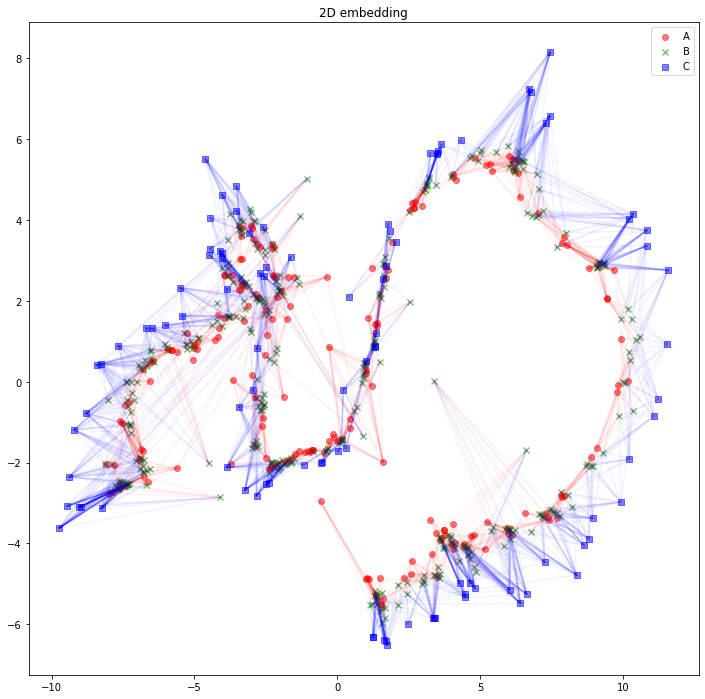
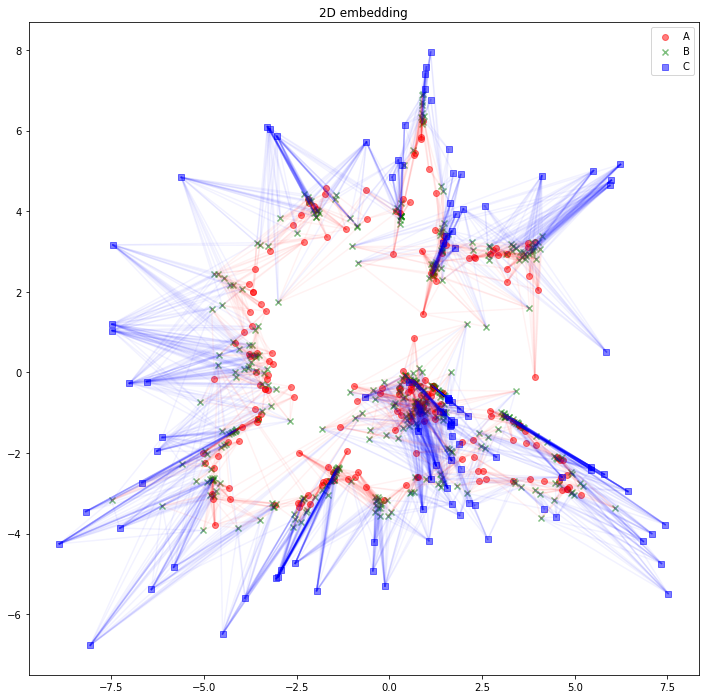
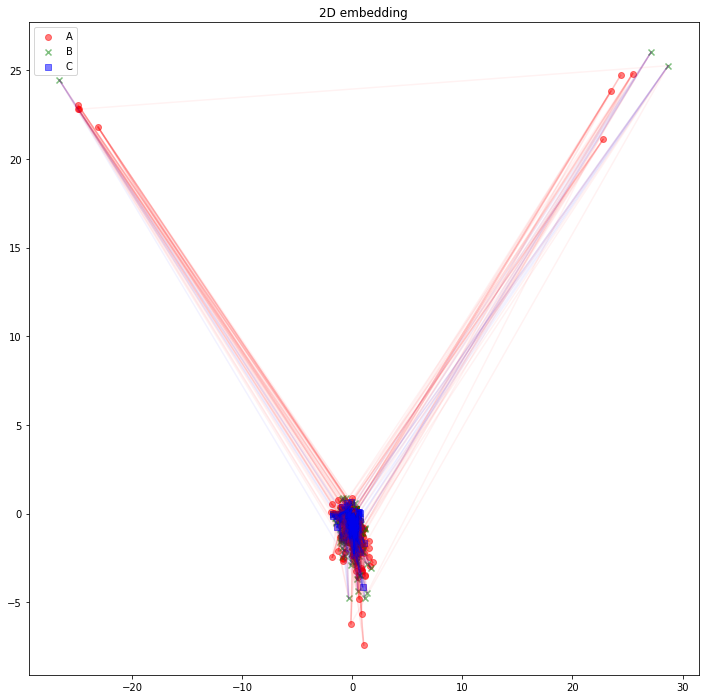
さて、可視化しましょう。Aタイプのノードは赤丸、Bタイプのノードは緑バツ、Cタイプのノードは青四角とします。AB間のエッジは赤線、BC間のエッジは青線にしました。
import matplotlib.pyplot as plt
plt.figure(figsize=(12, 12))
plt.scatter(z_A_2d[:,0],z_A_2d[:,1],color="red",alpha=0.5,label="A")
plt.scatter(z_B_2d[:,0],z_B_2d[:,1],color="green",alpha=0.5,label="B", marker="x")
plt.scatter(z_C_2d[:,0],z_C_2d[:,1],color="blue",alpha=0.5,label="C", marker="s")
for edge in edges_AB:
plt.plot(
[z_A_2d[edge[0],0], z_B_2d[edge[1],0]],
[z_A_2d[edge[0],1], z_B_2d[edge[1],1]],
color='red', alpha=0.05)
for edge in edges_BC:
plt.plot(
[z_B_2d[edge[0],0], z_C_2d[edge[1],0]],
[z_B_2d[edge[0],1], z_C_2d[edge[1],1]],
color='blue', alpha=0.05)
plt.legend()
plt.title("2D embedding")
plt.show()
うーん、解釈が難しそうですね。「似たようなノードと繋がっている同じタイプのノードは、近くに配置されている」と解釈できるでしょうか。「互いに接続しているノード同士が、近くに配置されている」わけではなさそうです。
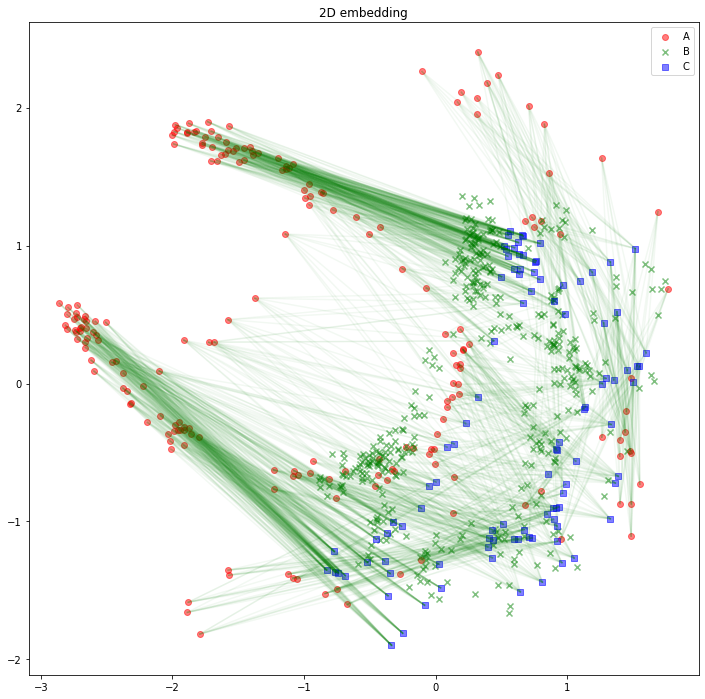
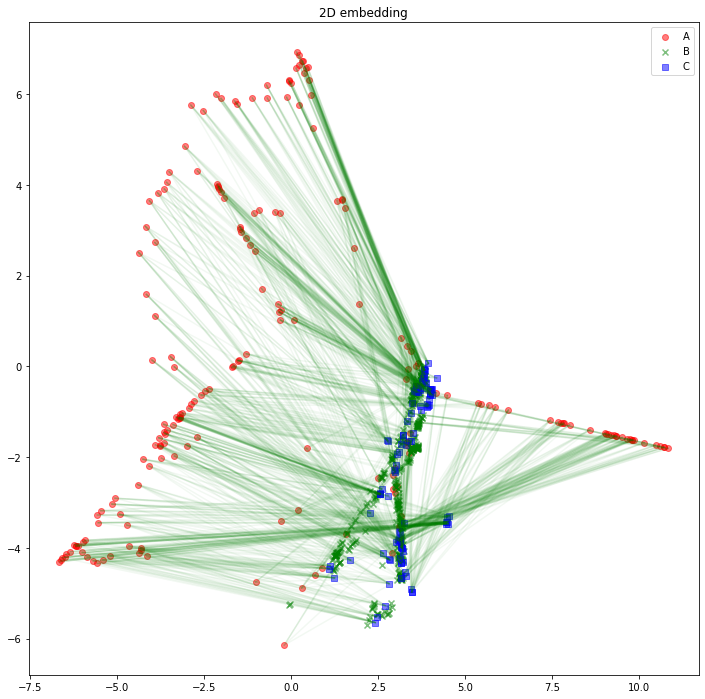
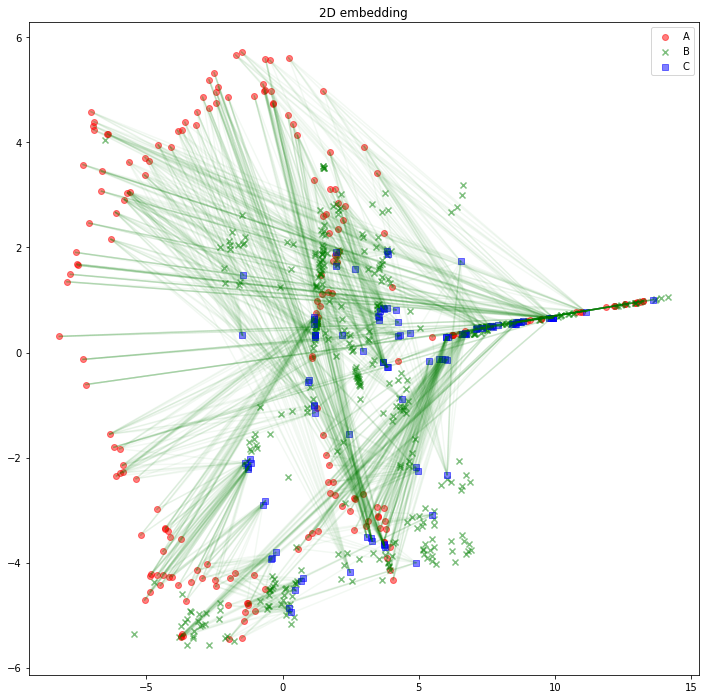
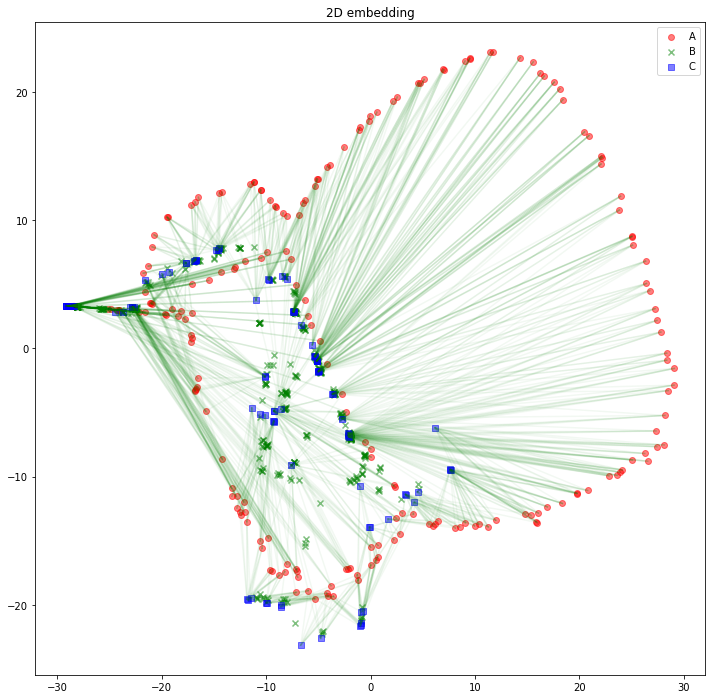
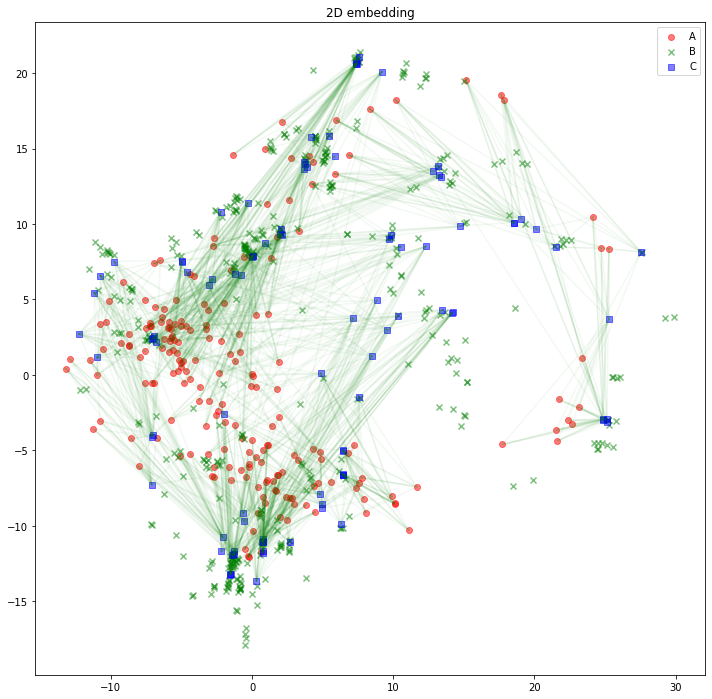
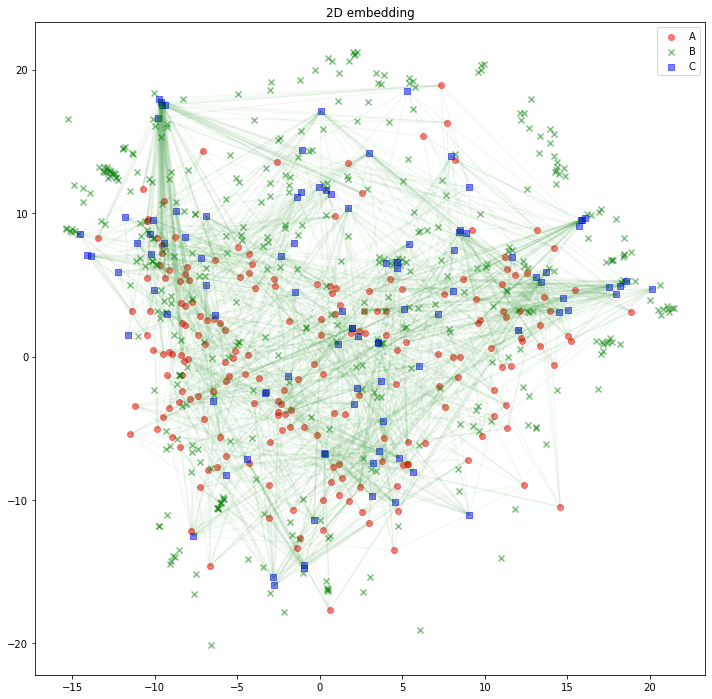
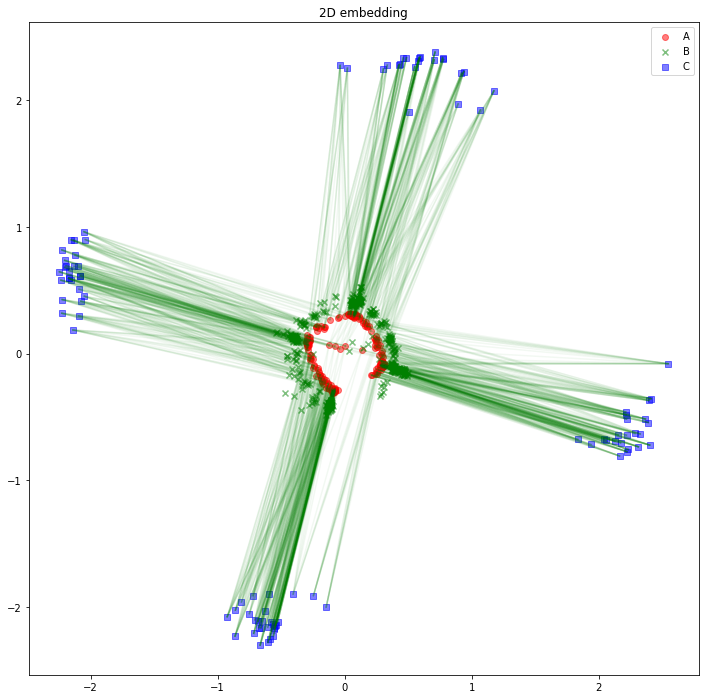
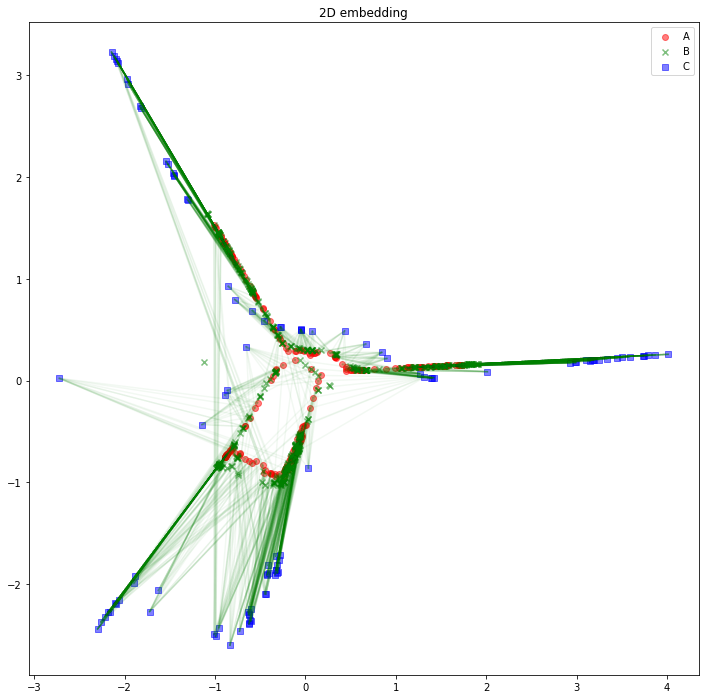
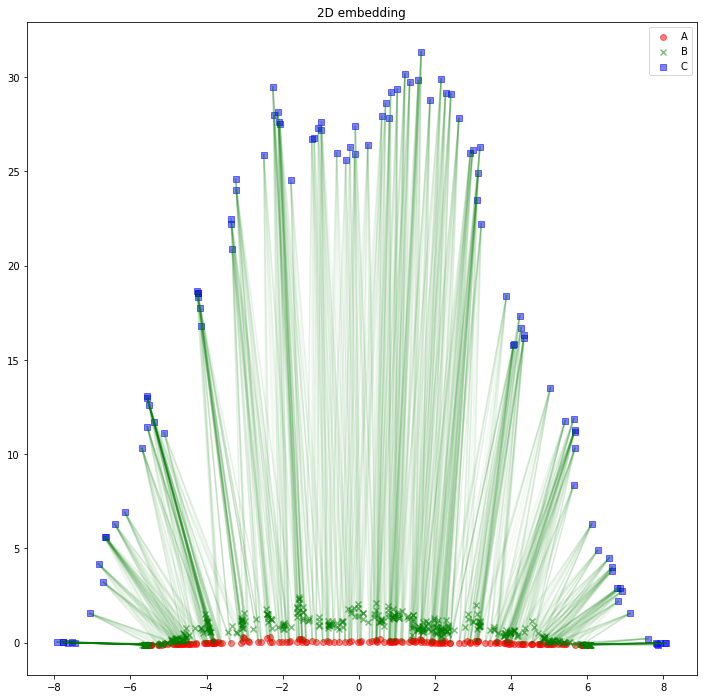
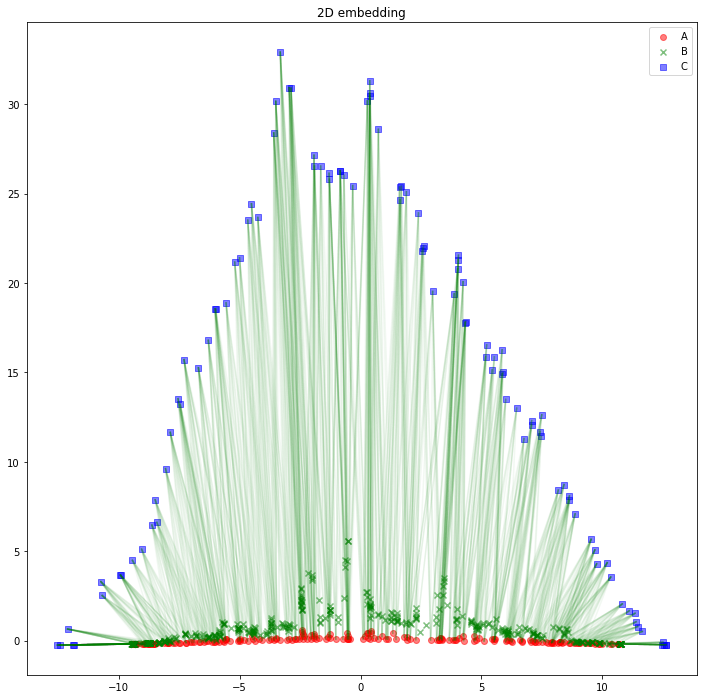
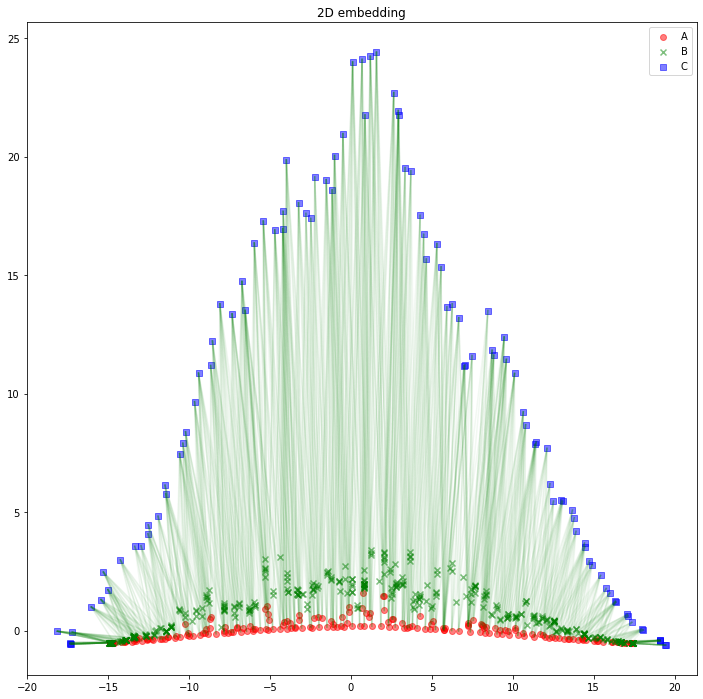
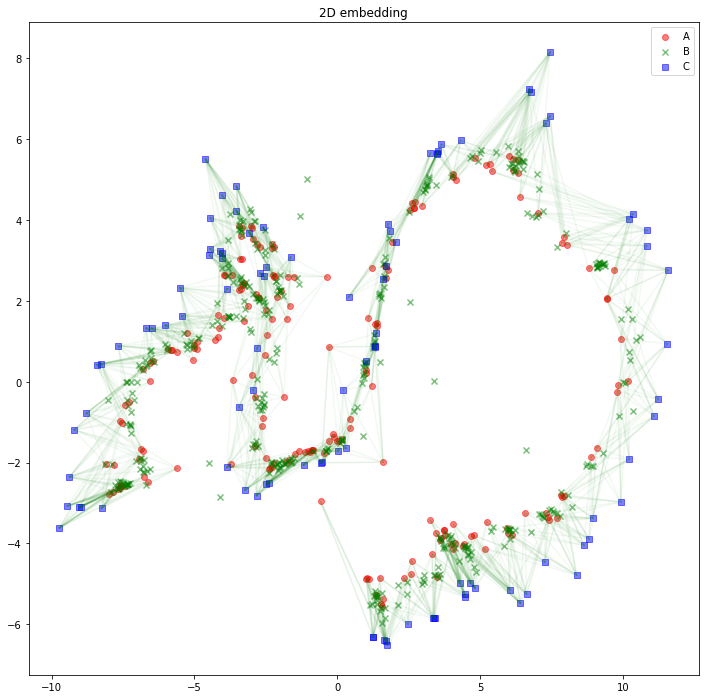
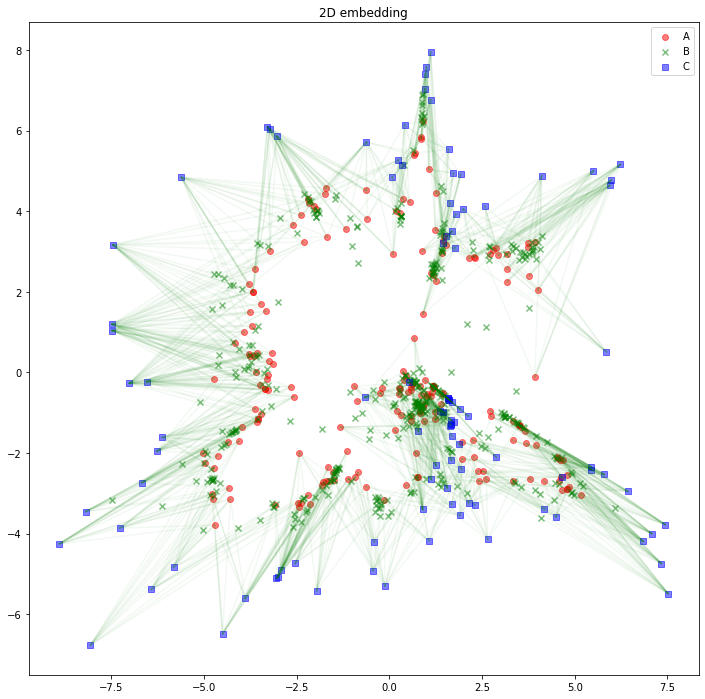
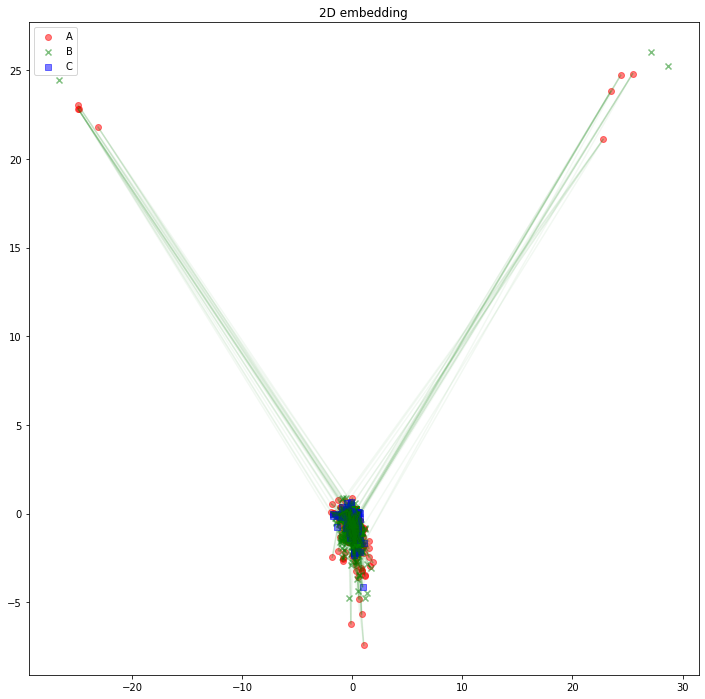
解釈の助けになるかどうかは分かりませんが、AB間、BC間のエッジは消して、AC間のエッジを緑線で表示してみるとこのようになります。
import matplotlib.pyplot as plt
plt.figure(figsize=(12, 12))
plt.scatter(z_A_2d[:,0],z_A_2d[:,1],color="red",alpha=0.5,label="A")
plt.scatter(z_B_2d[:,0],z_B_2d[:,1],color="green",alpha=0.5,label="B", marker="x")
plt.scatter(z_C_2d[:,0],z_C_2d[:,1],color="blue",alpha=0.5,label="C", marker="s")
for edge in edges_AC:
plt.plot(
[z_A_2d[edge[0],0], z_C_2d[edge[1],0]],
[z_A_2d[edge[0],1], z_C_2d[edge[1],1]],
color='green', alpha=0.05)
plt.legend()
plt.title("2D embedding")
plt.show()
MetaPath2Vec (embedding_dim = 4)
過去記事と同様に、embedding_dim を大きくするとどうなるか実験したいと思います。まずは embedding_dim = 4
むむっ、embedding_dim = 2 のときよりも結果がつぶれて悪化したように見えますね...
MetaPath2Vec (embedding_dim = 8)
embedding_dim = 4 のときよりは良いかもしれませんが、embedding_dim = 2 のときのほうがもっと良いな...
MetaPath2Vec (embedding_dim = 16)
なんだろこれ...理解不能
MetaPath2Vec (embedding_dim = 32)
おっ?なんとなく理解「可」能っぽい構造になってきた?
MetaPath2Vec (embedding_dim = 64)
タイプが異なっていても、関係の深いノード間が近くに配置されるようになってきた、かも?
MetaPath2Vec (embedding_dim = 128)
理解不能理解不能理解不能理解不能ッ...!
Node2Vec (embedding_dim = 2)
それでは、次に Node2Vec で計算してみて、 MetaPath2Vec の結果と比較しますよ! 基本的には過去記事と同じなので参照していただくとして、主な変更部分だけお示ししますね。
import numpy as np
from torch_geometric.data import Data, InMemoryDataset
class HeteroDataset(InMemoryDataset):
def __init__(self, transform = None):
super().__init__('.', transform)
embeddings = np.concatenate([A, B, C])
embeddings = torch.tensor([[x] for x in embeddings])
edges = []
for edge in edges_AB:
edges.append([edge[0], edge[1] + len(A)])
for edge in edges_BC:
edges.append([edge[0] + len(A), edge[1] + len(A) + len(B)])
edges = torch.tensor(edges).T
ys = np.concatenate([A, B, C])
ys = np.where(ys > 0.5, 1, 0)
ys = torch.tensor(ys)
data = Data(x=embeddings, edge_index=edges, y=ys)
self.data, self.slices = self.collate([data])
dataset = HeteroDataset()
data = dataset.data
data.x = torch.zeros_like(data.x)
embedding_dim = 2
model = torch_geometric.nn.Node2Vec(
data.edge_index, embedding_dim=embedding_dim, walk_length=20,
context_size=10, walks_per_node=20,
num_negative_samples=10, p=0.5, q=1, sparse=True).to(device)
loader = model.loader(batch_size=64, shuffle=True, num_workers=2)
optimizer = torch.optim.SparseAdam(list(model.parameters()), lr=0.01)
z = best_model(torch.arange(data.num_nodes, device=device)).detach().cpu().numpy()
z.shape
z_A = z[:len(A), :]
z_B = z[len(A):len(A)+len(B), :]
z_C = z[len(A)+len(B):, :]
なんだこれはw
よく分からないが、これはこれで面白いww
Node2Vec (embedding_dim = 4)
うーん、何と言ったら良いやら...w
Node2Vec (embedding_dim = 8)
おわw 面白いw これ、今回自作したデータ構造を非常によく表しているかもしれない!
Node2Vec (embedding_dim = 16)
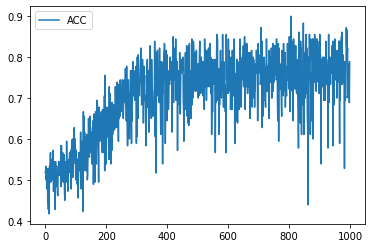
結果はあまり変わらないけど学習曲線は安定している
Node2Vec (embedding_dim = 32)
うーん、安定しているなぁ。ひょっとして MetaPath2Vec より Node2Vec のほうが良いんじゃない?
Node2Vec (embedding_dim = 64)
お? ぐにゃっと曲がった。でもまだデータ構造をうまく表しているかも。
Node2Vec (embedding_dim = 128)
学習曲線も、潜在空間も、乱れ始めたかな...?
Node2Vec (embedding_dim = 256)
なるほど、もうダメっぽいですね。
まとめ
自作ネットワークを作成して、MetaPath2Vec でノードを潜在空間にプロットしたり、 Node2Vec と比較したりしました。結果としては、Node2Vec のほうが良いかも、となりましたが、これは、今回用いたデータや、ハイパーパラメーターに依存するんだろうとは思います。まだ分からないことも多いので、またいろいろ勉強してみます。