はじめに
乳がんは、世界中の女性にとって大きな問題となっており、早期発見が重要な要素です。
データから乳がんの診断を精度高く行うことができれば、診断の主観性の排除やコスト削減等のメリットがあります。
本記事では、乳がんデータセットを用いたデータ分析の一例について説明します。
概要
scikit-learn付属の乳がんのデータセットを使って、機械学習の教師あり(分類)を行います。
分析は、以下の手順で進めます。
- ライブラリの読み込み
- データセットの確認
- 複数のアルゴリズムを用いた精度比較
- パラメータチューニングによる精度向上の検証
ライブラリの読み込み
今回比較に使用するモデルは、いずれもscikit-learnで提供されています。
いずれのパッケージもPyPIで公開されており、簡単にインストールすることが可能です。
import pandas as pd
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split, cross_val_score
from sklearn.preprocessing import StandardScaler
from sklearn.linear_model import LogisticRegression
from sklearn.svm import SVC
from sklearn.ensemble import RandomForestClassifier
import optuna
import matplotlib.pyplot as plt
import seaborn as sns
データセットの確認
scikit-learnが公開しているデータは、sklearn.datasetsのメソッドを使って読み込むことができます。
今回使用する乳がんのデータセットは、ウィスコンシン大学の乳がん診断データセット(Wisconsin Diagnostic Breast Cancer, WDBC)と呼ばれています(参照)。
このデータセットは、乳がんの診断に関連する特徴量を持つ患者のデータを含んでいます。
# データの読み込み
data_breast_cancer = load_breast_cancer()
df_target = pd.DataFrame(data_breast_cancer["target"], columns=["target"])
df_data = pd.DataFrame(data_breast_cancer["data"], columns=data_breast_cancer["feature_names"])
特徴量のデータ型を確認します。
# 各columnのデータ型を表示
print(f'{df_data.dtypes} \n')
本データに含まれる特徴量は、いずれも連続値であることがわかります。
次に、統計量を確認します。
# データにおける、数値データの統計量を表示
display(df_data.describe())
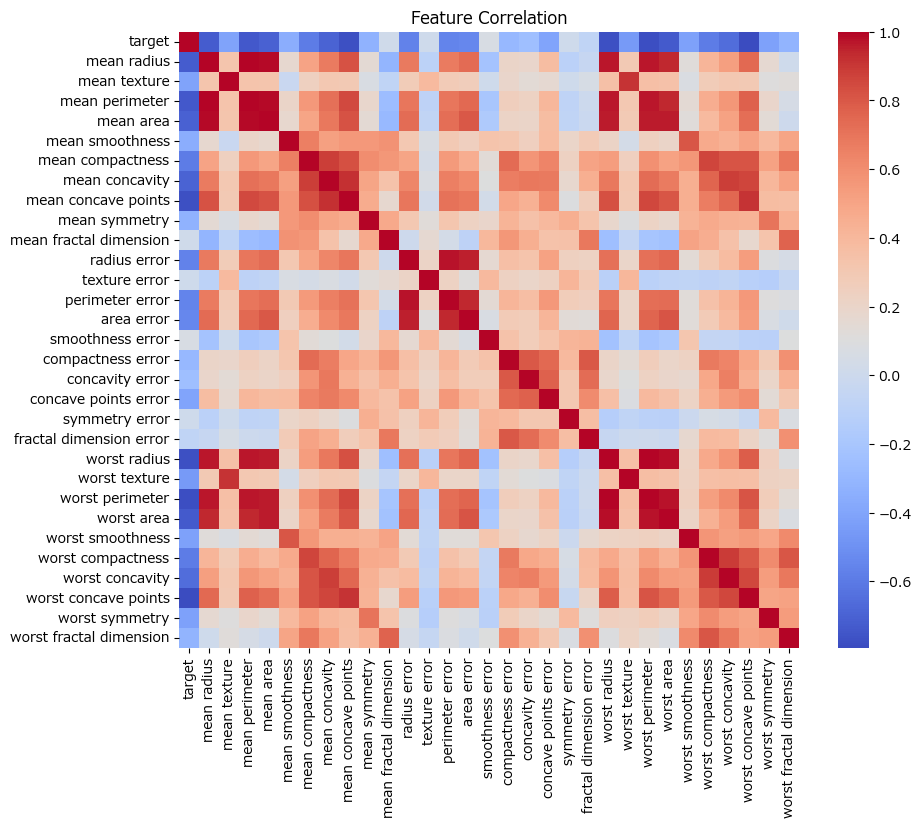
続いて、各特徴量の分布及び相関を確認します。
# 特徴量の名前の取得
feature_names = data_breast_cancer.feature_names
# 特徴量の分布を可視化
plt.figure(figsize=(12, 24))
for i, feature in enumerate(feature_names):
plt.subplot(10, 3, i + 1)
sns.histplot(df_data[feature], kde=True)
plt.title(feature)
plt.tight_layout()
plt.show()
# 特徴量間の相関を可視化
plt.figure(figsize=(10, 8))
sns.heatmap(df_data.corr(), cmap='coolwarm')
plt.title('Feature Correlation')
plt.show()
予測モデルの構築
ロジスティック回帰、サポートベクターマシン(SVM)、ランダムフォレストのモデルの精度を比較します。
X = data_breast_cancer.data
y = data_breast_cancer.target
# データを訓練用とテスト用に分割
X_train, X_test, y_train, y_test = train_test_split(df_data, df_target, test_size=0.3, random_state=1)
# 特徴量の正規化
scaler = StandardScaler()
X_train = scaler.fit_transform(X_train)
X_test = scaler.transform(X_test)
# ロジスティック回帰
log_reg = LogisticRegression(random_state=1)
log_reg.fit(X_train, y_train)
log_reg_acc = cross_val_score(log_reg, X_train, y_train, cv=5).mean()
# サポートベクターマシン
svc = SVC(kernel='linear', C=1, random_state=1)
svc.fit(X_train, y_train)
svc_acc = cross_val_score(svc, X_train, y_train, cv=5).mean()
# ランダムフォレスト
rand_forest = RandomForestClassifier(random_state=1)
rand_forest.fit(X_train, y_train)
rand_forest_acc = cross_val_score(rand_forest, X_train, y_train, cv=5).mean()
print("Logistic Regression Accuracy: {:.2f}%".format(log_reg_acc * 100))
print("SVM Accuracy: {:.2f}%".format(svc_acc * 100))
print("Random Forest Accuracy: {:.2f}%".format(rand_forest_acc * 100))
Optunaによるパラメータチューニング
続いて、Optunaを用いてパラメータ最適化を行います。
Optunaを使うと、ベイズ最適化により、定義したハイパーパラメーターの範囲から最適なパラメータの組み合わせを探索することができます。
SVM
def objective_svm(trial):
C = trial.suggest_loguniform('C', 1e-10, 1e10)
gamma = trial.suggest_loguniform('gamma', 1e-10, 1e10)
kernel = trial.suggest_categorical('kernel', ['linear', 'rbf', 'sigmoid'])
clf = SVC(C=C, gamma=gamma, kernel=kernel)
return cross_val_score(clf, X_train, y_train,
n_jobs=-1, cv=5).mean()
study_svm = optuna.create_study(direction='maximize')
study_svm.optimize(objective_svm, n_trials=100)
best_params_svm = study_svm.best_params
best_accuracy_svm = study_svm.best_value
print("Best Parameters (SVM):", best_params_svm)
print("Best Accuracy (SVM):", best_accuracy_svm)
# Best Parameters (SVM): {'C': 1.2980731009141622, 'gamma': 0.00045010691978746915, 'kernel': 'linear'}
# Best Accuracy (SVM): 0.9824050632911392
Logistic Regression
def objective_logreg(trial):
C = trial.suggest_loguniform('C', 1e-10, 1e10)
max_iter = trial.suggest_int('max_iter', 100, 1000)
clf = LogisticRegression(C=C, max_iter=max_iter)
return cross_val_score(clf, X_train, y_train,
n_jobs=-1, cv=5).mean()
study_logreg = optuna.create_study(direction='maximize')
study_logreg.optimize(objective_logreg, n_trials=100)
best_params_logreg = study_logreg.best_params
best_accuracy_logreg = study_logreg.best_value
print("Best Parameters (Logistic Regression):", best_params_logreg)
print("Best Accuracy (Logistic Regression):", best_accuracy_logreg)
Random Forest
def objective_rf(trial):
n_estimators = trial.suggest_int('n_estimators', 100, 1000)
max_depth = trial.suggest_int('max_depth', 5, 15)
min_samples_split = trial.suggest_int('min_samples_split', 2, 10)
random_state = trial.suggest_int('random_state', 0, 42)
clf = RandomForestClassifier(n_estimators=n_estimators, max_depth=max_depth,
min_samples_split=min_samples_split, random_state=random_state)
return cross_val_score(clf, X_train, y_train,
n_jobs=-1, cv=5).mean()
study_rf = optuna.create_study(direction='maximize')
study_rf.optimize(objective_rf, n_trials=100)
best_params_rf = study_rf.best_params
best_accuracy_rf = study_rf.best_value
print("Best Parameters (Random Forest):", best_params_rf)
print("Best Accuracy (Random Forest):", best_accuracy_rf)
まとめ
精度評価を行った結果を表にまとめます。
| アルゴリズム | Accuracy(パラメータチューニング無し) | Accuracy(パラメータチューニングあり) |
|---|---|---|
| SVM | 0.9799 | 0.9824 |
| Logistic Regression | 0.9699 | 0.9774 |
| Random Forest | 0.9548 | 0.9623 |
アルゴリズムの中では、パラメータチューニングありのSVMが最も高い精度で乳がん患者を予測することができていました。
また、いずれのアルゴリズムにおいても、Optunaによるパラメータチューニングにより、Accuracyが向上することがわかりました。