書籍「ゼロから作るディープラーニング」をもとに
畳み込みニューラルネットワーク(CNN、Convolutional Neural Network)を勉強しているのですが、途中に出てくるim2col関数というものについて理解が難しかったため、
自分なりの噛み砕きの経緯を書いてみました。
もしもどなたかの参考になれば幸いです。
im2col関数について
CNNの畳み込み演算において複雑なループ処理を避けるため、
フィルター適用領域ごとに一列のデータになるよう変換する関数です。
入力データとフィルターにこの関数を適用することで、
行列のドット演算で一気に畳み込み演算が行えます。
オリジナルの実装
「ゼロから作る〜」で紹介されている実装はこのようなものです。
def im2col(input_data, filter_h, filter_w, stride=1, pad=0):
N, C, H, W = input_data.shape
out_h = (H + 2*pad - filter_h)//stride + 1
out_w = (W + 2*pad - filter_w)//stride + 1
img = np.pad(input_data, [(0,0), (0,0), (pad, pad), (pad, pad)], 'constant')
col = np.zeros((N, C, filter_h, filter_w, out_h, out_w))
for y in range(filter_h):
y_max = y + stride*out_h
for x in range(filter_w):
x_max = x + stride*out_w
col[:, :, y, x, :, :] = img[:, :, y:y_max:stride, x:x_max:stride]
col = col.transpose(0, 4, 5, 1, 2, 3).reshape(N*out_h*out_w, -1)
return col
書籍の説明を見ると目指すところは理解できるのですが、
このループ処理部分で行っている事がどうにも理解できませんでした。
引っかかった点
-
y_maxとx_maxが何を意味しているのかよくわからない - imgのスライスで何を取り出しているのかよくわからない
y:y_max:stride,、x:x_max:strideって何? - フィルターサイズについてのループだけで処理できる理由がよくわからない
そこで、まずは自分なりに素朴な発想で実装してみました。
素朴な実装
フィルターを移動させるループ
→フィルタ内の各画素をコピーするループ
の順でx方向・y方向、4重ループ処理すればいけるはず...
という発想でforループ部分のみ変更してみたのが以下のim2col_slowです。
def im2col_slow(input_data, filter_h, filter_w, stride=1, pad=0):
N, C, H, W = input_data.shape
out_h = (H + 2*pad - filter_h)//stride + 1
out_w = (W + 2*pad - filter_w)//stride + 1
img = np.pad(input_data, [(0,0), (0,0), (pad, pad), (pad, pad)], 'constant')
col = np.zeros((N, C, filter_h, filter_w, out_h, out_w))
for move_y in range(out_h):
for move_x in range(out_w):
for y in range(filter_h):
for x in range(filter_w):
col[:, :, y, x, move_y, move_x] = \
img[:, :, y + stride * move_y, x + stride * move_x]
col = col.transpose(0, 4, 5, 1, 2, 3).reshape(N*out_h*out_w, -1)
return col
実行してみます。
(見やすくするためにデータ数1、チャンネル数1に絞ってます)
data = np.random.rand(1, 1, 7, 7) * 100 // 1
print('========== input ==========\n', data)
print('=====================')
filter_h = 3
filter_w = 3
stride = 2
pad = 0
col = im2col(data, filter_h=filter_h, filter_w=filter_w, stride=stride, pad=pad)
col2 = im2col_slow(data, filter_h=filter_h, filter_w=filter_w, stride=stride, pad=pad)
print('========== col ==========\n', col)
print('=====================')
print('========== col2 ==========\n', col2)
print('=====================')
同様の結果を得られました。
========== input ==========
[[[[30. 91. 11. 13. 52. 44. 98.]
[99. 6. 35. 41. 97. 72. 79.]
[ 5. 92. 15. 95. 72. 8. 10.]
[68. 5. 86. 25. 69. 46. 70.]
[95. 32. 98. 49. 51. 19. 46.]
[32. 15. 39. 44. 76. 58. 49.]
[43. 47. 95. 1. 1. 12. 21.]]]]
=====================
========== col ==========
[[30. 91. 11. 99. 6. 35. 5. 92. 15.]
[11. 13. 52. 35. 41. 97. 15. 95. 72.]
[52. 44. 98. 97. 72. 79. 72. 8. 10.]
[ 5. 92. 15. 68. 5. 86. 95. 32. 98.]
[15. 95. 72. 86. 25. 69. 98. 49. 51.]
[72. 8. 10. 69. 46. 70. 51. 19. 46.]
[95. 32. 98. 32. 15. 39. 43. 47. 95.]
[98. 49. 51. 39. 44. 76. 95. 1. 1.]
[51. 19. 46. 76. 58. 49. 1. 12. 21.]]
=====================
========== col2 ==========
[[30. 91. 11. 99. 6. 35. 5. 92. 15.]
[11. 13. 52. 35. 41. 97. 15. 95. 72.]
[52. 44. 98. 97. 72. 79. 72. 8. 10.]
[ 5. 92. 15. 68. 5. 86. 95. 32. 98.]
[15. 95. 72. 86. 25. 69. 98. 49. 51.]
[72. 8. 10. 69. 46. 70. 51. 19. 46.]
[95. 32. 98. 32. 15. 39. 43. 47. 95.]
[98. 49. 51. 39. 44. 76. 95. 1. 1.]
[51. 19. 46. 76. 58. 49. 1. 12. 21.]]
=====================
オリジナルとの比較
自分で実装してみたおかげで、オリジナルの実装は
上記の素朴版で行っているフィルター移動分の2重ループをしなくて済むよう効率化したものだと気づきました。
(ストライド幅刻みでスライスすることで、フィルタを移動させて取得する分を一気に取得・コピーしている)
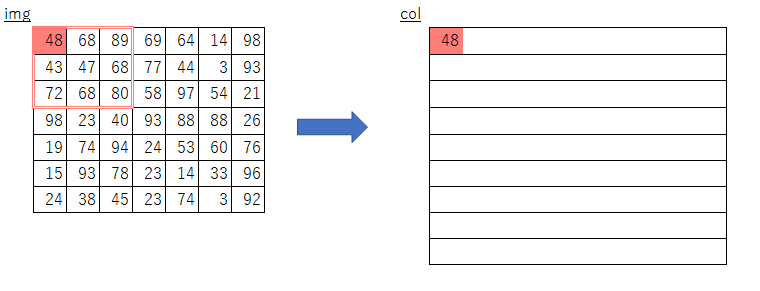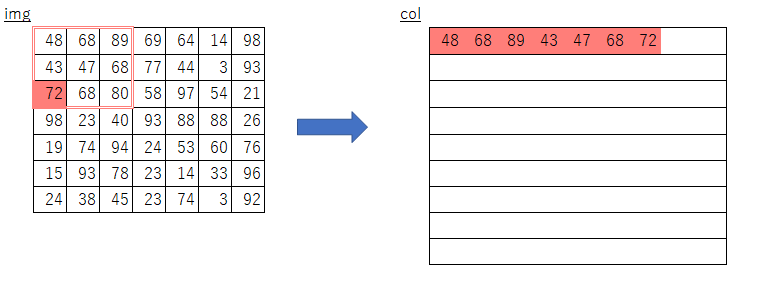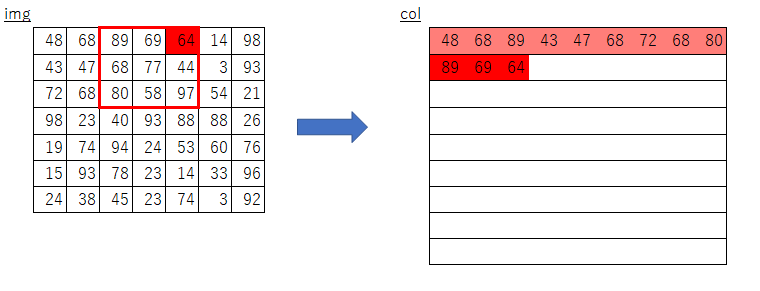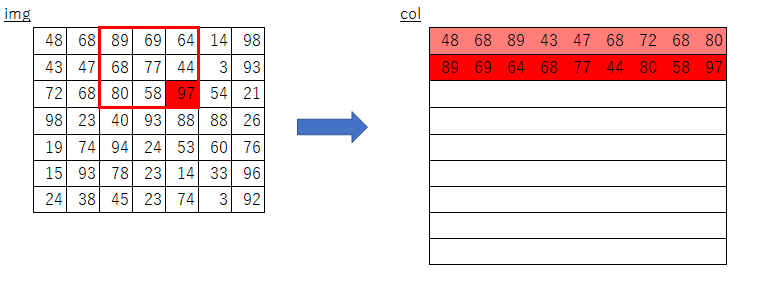
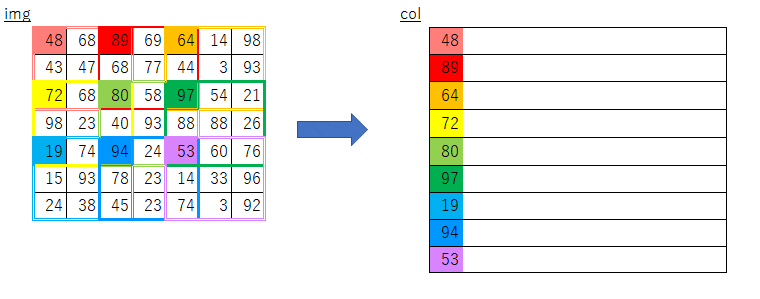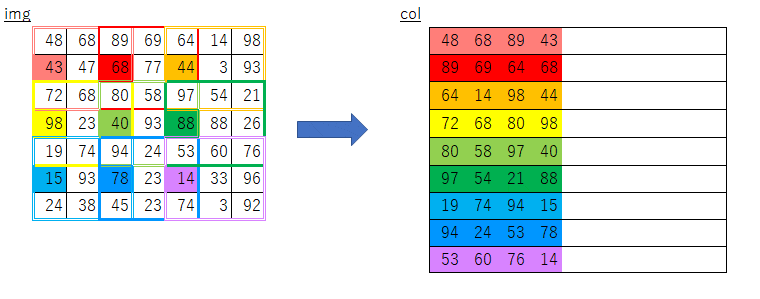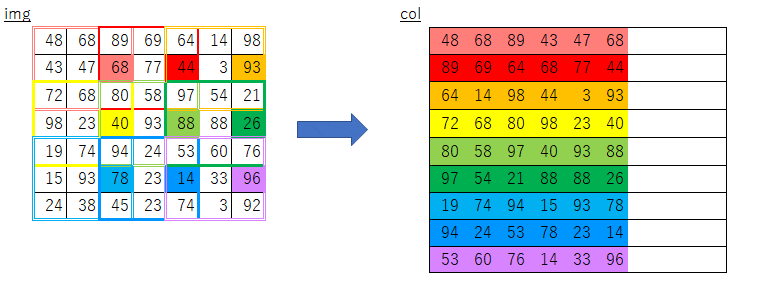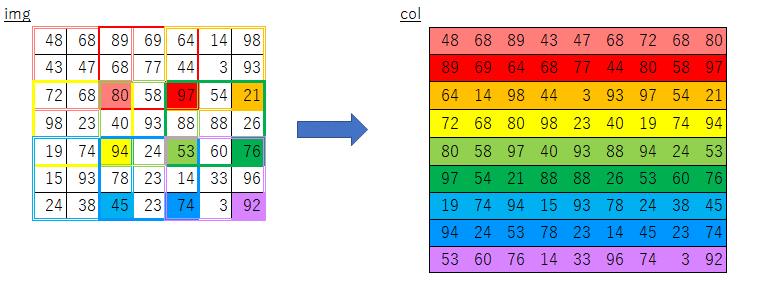
絵にしてみるとこんな感じになるかと思います。
素朴版の実装におけるimgからcolへのコピー
オリジナルim2colにおけるimgからcolへのコピー
ループ処理が効率化された結果、あのような実装になっているのだなぁ...
という感じで自分の理解は落ち着きました。