出力結果には公式ドキュメントからの引用を載せていることが多いです。
たまに自分のサンプルから得られた出力結果も混ざっています。
公式ドキュメント
https://flowkit.readthedocs.io/en/latest/
https://pypi.org/project/FlowKit/
ライブラリのインストール
pip install FlowKit
ライブラリのインポート
import flowkit as fk
import bokeh
from bokeh.plotting import show
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
bokeh.io.output_notebook()
バージョンの確認
fk.__version__
'1.0.1'
FCSファイルを読み込む
flowkit.Sample()(ここではfk.Sample())の引数にFCSファイルのパスを指定。
#FCSファイルの読み込み
fcs_path = './1SI.fcs'
sample = fk.Sample(fcs_path)
#sample情報の出力(FCSが読み込まれているか確認)
print(sample)
FCSファイル名、チャンネル数、イベント数が表示される。
Sample(v3.1, 1SI.fcs, 28 channels, 251390 events)
metadataの出力
辞書型で出力される。
この中にはいろいろなデータが格納されているが、使うのはコンペンセーションの値くらいだと思われる。
#sampleメタデータの出力(辞書型)
print(sample.get_metadata())
出力結果(長いので折り畳み)
{'byteord': '4,3,2,1', 'datatype': 'I', 'nextdata': '0', 'sys': 'Macintosh System Software 9.0.4', 'creator': 'CELLQuestª 3.3', 'tot': '13367', 'mode': 'L', 'par': '8', 'p1n': 'FSC-H', 'p1r': '1024', 'p1b': '16', 'p1e': '0,0', 'p1g': '3.67', 'p2n': 'SSC-H', 'p2r': '1024', 'p2b': '16', 'p2e': '0,0', 'p2g': '8', 'p3n': 'FL1-H', 'p3r': '1024', 'p3b': '16', 'p3e': '4,0', 'p4n': 'FL2-H', 'p4r': '1024', 'p4b': '16', 'p4e': '4,0', 'p5n': 'FL3-H', 'p5r': '1024', 'p5b': '16', 'p5e': '4,0', 'p1s': 'FSC-Height', 'p2s': 'SSC-Height', 'p3s': 'CD4 FITC', 'p4s': 'CD8 B PE', 'p5s': 'CD3 PerCP', 'p6n': 'FL2-A', 'p6r': '1024', 'p6b': '16', 'p6e': '0,0', 'timeticks': '100', 'p7n': 'FL4-H', 'p7r': '1024', 'p7e': '4,0', 'p7b': '16', 'p7s': 'CD8 APC', 'p8n': 'Time', 'p8r': '1024', 'p8e': '0,0', 'p8b': '16', 'p8s': 'Time (102.40 sec.)', 'sample id': 'Default Patient ID', 'src': 'Default', 'case number': 'Default Case Number', 'cyt': 'FACSCalibur', 'cytnum': 'E3820', 'btim': '16:31:33', 'etim': '16:31:52', 'bdacqlibversion': '3.1', 'bdnpar': '7', 'bdp1n': 'FSC-H', 'bdp2n': 'SSC-H', 'bdp3n': 'FL1-H', 'bdp4n': 'FL2-H', 'bdp5n': 'FL3-H', 'bdp6n': 'FL2-A', 'bdp7n': 'FL4-H', 'bdword0': '24', 'bdword1': '394', 'bdword2': '492', 'bdword3': '477', 'bdword4': '566', 'bdword5': '397', 'bdword6': '397', 'bdword7': '397', 'bdword8': '398', 'bdword9': '397', 'bdword10': '300', 'bdword11': '299', 'bdword12': '551', 'bdword13': '4', 'bdword14': '397', 'bdword15': '501', 'bdword16': '481', 'bdword17': '586', 'bdword18': '574', 'bdword19': '100', 'bdword20': '100', 'bdword21': '100', 'bdword22': '100', 'bdword23': '1', 'bdword24': '1', 'bdword25': '0', 'bdword26': '0', 'bdword27': '0', 'bdword28': '136', 'bdword29': '52', 'bdword30': '52', 'bdword31': '52', 'bdword32': '52', 'bdword33': '52', 'bdword34': '12', 'bdword35': '201', 'bdword36': '6', 'bdword37': '138', 'bdword38': '280', 'bdword39': '3', 'bdword40': '3', 'bdword41': '100', 'bdword42': '100', 'bdword43': '0', 'bdword44': '1023', 'bdword45': '1023', 'bdword46': '1023', 'bdword47': '53', 'bdword48': '550', 'bdword49': '56', 'bdword50': '72', 'bdword51': '52', 'bdword52': '0', 'bdword53': '0', 'bdword54': '0', 'bdword55': '0', 'bdword56': '0', 'bdword57': '0', 'bdword58': '0', 'bdword59': '0', 'bdword60': '0', 'bdword61': '0', 'bdword62': '0', 'bdword63': '0', 'bdlasermode': '1', 'calibfile': 'FALSE', 'p7thresvol': '52', 'fil': 'B07', 'date': '23-Aug-02', 'number well info keywords': '3', '&1sample': '200', '&2number of washes': '1', '&3mixing vol': '100', '&4number of mixes': '2', '&5data file prefix part #1\\\\&6data file prefix part #2\\\\&7data file prefix part #3\\\\&8acquisition doc.': 'LYMPH SUBSET ACQ', '&9instr. sett. file': 'E#7 Settings #1', '&10patient id': ' FJ#192659', '&11day': '35d', '&12sample id': 'T-cells', '&13analysis doc.': ''}
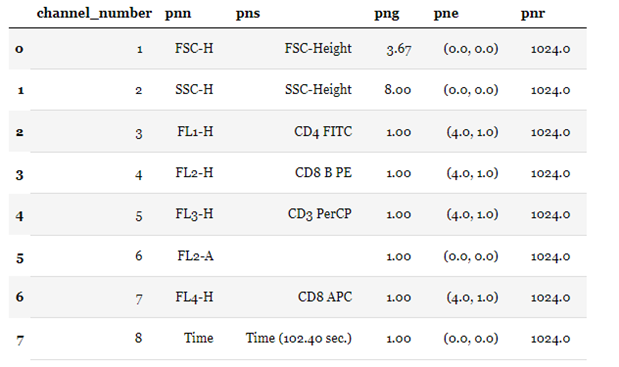
チャンネル情報の出力
print(sample.channels)
DataFrameで出力される。
コンペンセーション値の出力
metadataの中からコンペンセーション値を取り出す。
(この文字列を加工してコンペンセーションとして適用するのは別の記事で。)
print(sample.get_metadata()['spillover'])
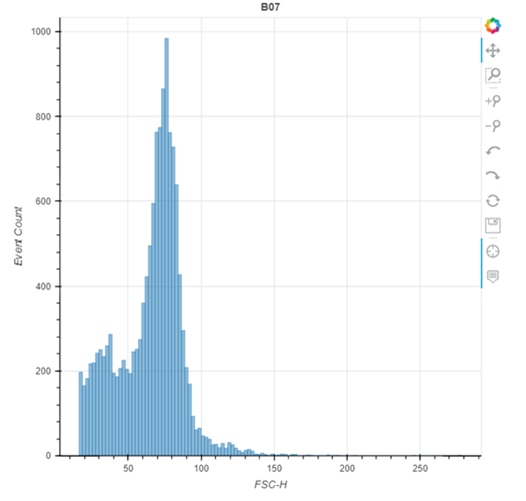
ヒストグラムの出力
引数にチャンネル名を入れる。例としてFSC-Hの場合を示す。
p = sample.plot_histogram('FSC-H', source='raw', bins=256)
show(p)
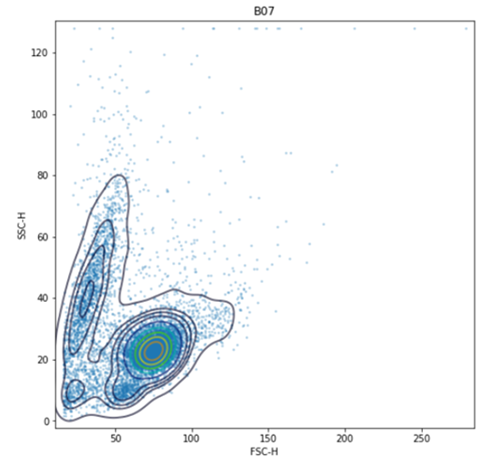
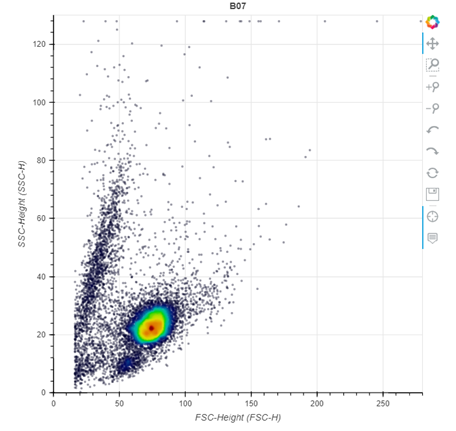
プロットの出力
他にも異なる種類の表示方法で出力できるが割愛。公式ドキュメント参照。
ここでは以下の例として2種類を紹介。
#by default, plot_contour uses sub-sampled events for performance
f = sample.plot_contour('FSC-H', 'SSC-H', source='raw', plot_events=True)
plt.show()
p = sample.plot_scatter(
'FSC-H', 'SSC-H',
source='raw', y_min=0., y_max=130, x_min=0., x_max=280, color_density=True
)
対数軸にしてプロットする
まず、サンプルに対数を適用する。(実行しても何も出力されない)
xform = fk.transforms.LogicleTransform('my_logicle', param_t=1024, param_w=0.5, param_m=4.5, param_a=0)
sample.apply_transform(xform)
プロットの際に引数sourceにxformを指定する。
FSCとSSCの表示には必要ないが、蛍光の測定結果をプロットする際には必要。
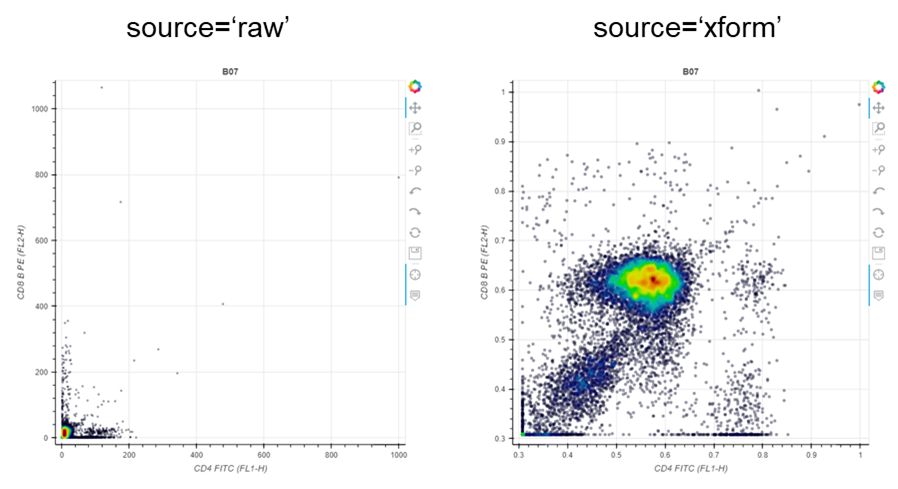
#source='raw' だと見にくい(左図)
p = sample.plot_scatter('FL1-H', 'FL2-H', source='raw')
show(p)
#source='xform' にすることで対数軸になる(右図)
p = sample.plot_scatter('FL1-H', 'FL2-H', source='xform')
show(p)
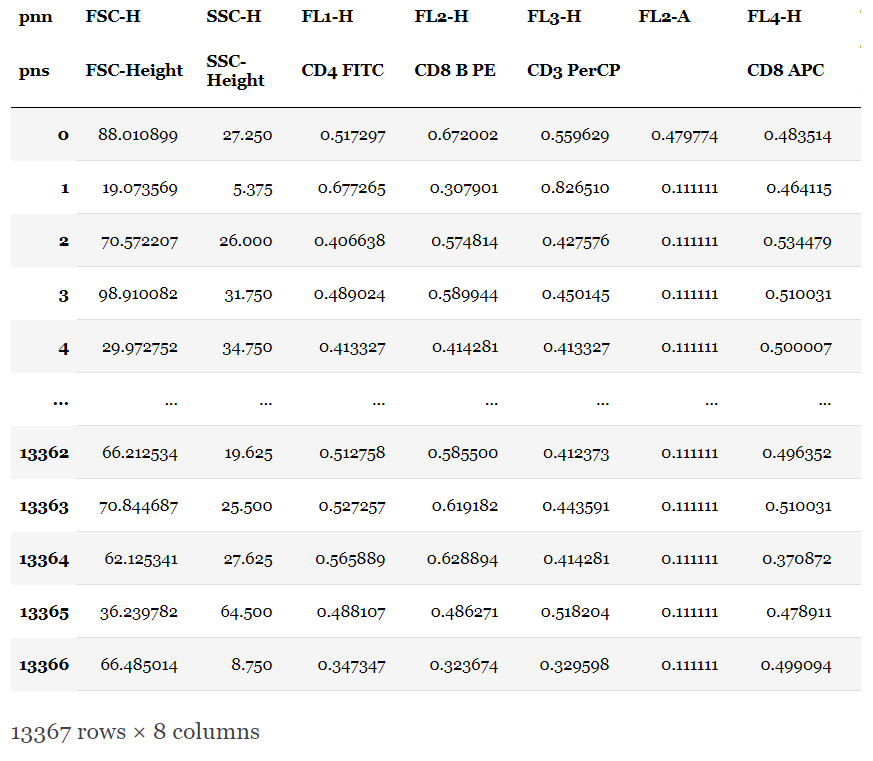
eventsの取り出し
ここに1つ1つのeventの測定値が格納されている。
#eventを取り出してDataFrameに
df_events = sample.as_dataframe(source='raw')
#eventを取り出してndarrayに
array_events = sample.get_events(source='raw')
次回予告
次の記事では、コンペンセーションの適用方法について記します。