機械学習のためのDocker/AWS 入門/SageMakerの統一的な本を書いたので紹介していきたいと思います。
Dockerとは?機械学習との関わりとは?
Dockerとは、「コンテナ仮想化を用いてアプリケーションを開発・配置・実行するためのオープンプラットフォーム」(Wikipediaより)です。
Dockerの基本的な説明は多数解説記事があるので割愛しますが、Dockerの大きな特徴として「使い捨てできる環境」が紹介されることがあります。機械学習での使い捨て環境といえば「Google Colaboratory」があり、「Colabで十分だからDockerいらないのでは」と思っている方も一定数いるでしょう。
Colabは非常に有用で、「とりあえずテスト感覚でコードを動かしてみる使い捨て環境」としては便利です。「使い捨てではない」本番を見据えた開発や、「ハイパーパラメーターやビッグデータの分散処理」など実践的なデータ分析ではColabでは不十分になってくることも多いです。このとき、機械学習でもDockerの有用性が出てきます。
機械学習でDockerを使うと何が嬉しいのか
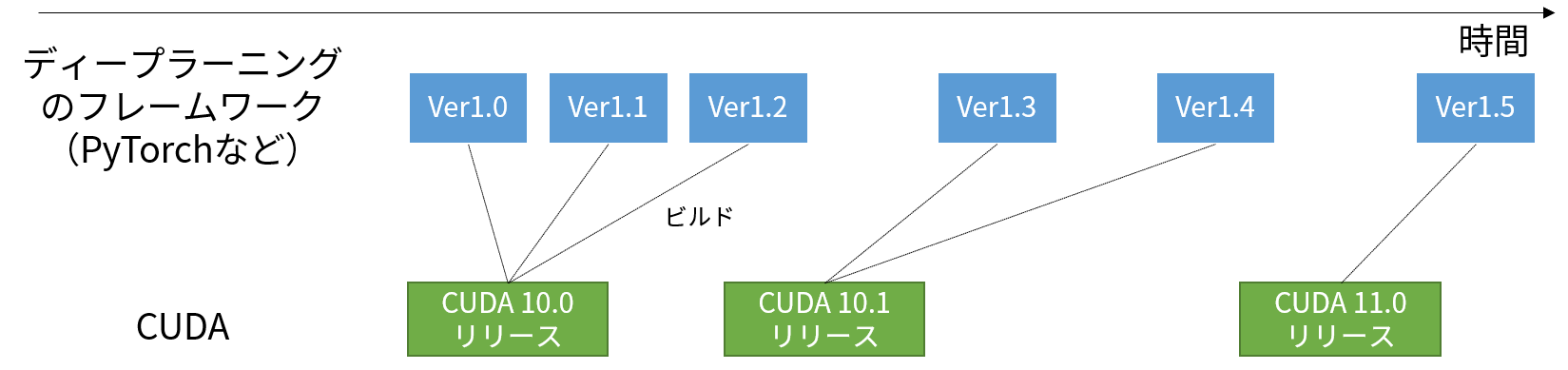
CUDAのバージョンを切り替えができる
PyTorchやTensorFlowといったディープラーニングのフレームワークでGPUを使う場合、CUDAのバージョンに依存します。
「TensorFlowのバージョンをあげようとしたら、CUDAのバージョンが対応してなくて、CUDAのインストールからやらないといけないのが面倒」という経験をした方もいるのではないでしょうか。
最新バージョンにアップデートするだけならこれでも良いのですが、例えば「ある論文Aのコードを使うために、一時的にフレームワークのバージョンを下げないといけない場合」は、使い終わったあと、また新しいバージョンの再インストールをしないといけなく、正直やってられませんよね。
Dockerならベースのイメージを変えればCUDAのバージョンを自在に切り替えられます。例えば、CUDA10.2+Ubuntu18.04なら、
FROM nvidia/cuda:10.2-cudnn8-devel-ubuntu18.04
CUDA11.3+Ubuntu20.04なら、ベースイメージのFROM~を変更するだけでOKです。
FROM nvidia/cuda:11.2.0-cudnn8-devel-ubuntu20.04
自前でDockerをビルドしなくても、NVIDIA-dockerのPyTorchのイメージを使い、最新のPyTorch環境を簡単に利用できます。
docker pull nvcr.io/nvidia/pytorch:22.07-py3
CUDAの環境を自在に変更できる、これがDockerの大きなメリットといえるでしょう。
ColabがDocker使えなくサイレントでライブラリのバージョンが変わる
これはDockerのメリットというより、Colabのデメリットですが、Colabではインストールされているライブラリのバージョンが予告なく変更され、アップデート履歴をトレースできません。
Dockerイメージのビルドで使うDockerfileでは、ライブラリのバージョンを指定できます。
# Sklearnの0.2.2を使いたい場合
RUN pip --no-cache-dir install sklearn==0.2.2
バージョンに由来するバグは稀によくあるので、バージョンの再現性を保証しやすいというのもメリットでしょう。
Windowsの場合WSL上で実行すると高速になることがある
特にこれはWindows上でPyTorchを動かすケースですが、Windows上で訓練を動かすよりも、Windows11付属のWSLにDocker(Ubuntu)を立ち上げ、Docker上で訓練したほうが訓練が速いことが確認できました。これは私が検証した例ですが、CIFAR-10をResNet-50で訓練したとき、WSL上のほうが、倍ぐらいの速度が出ているのが確認できます(単位:病)。
Windows+コマンドラインの場合
| 手法 | 1回目 | 2回目 | 3回目 | 平均 |
|---|---|---|---|---|
| PyTorch | 920.5 | 921.7 | 912 | 918.1 |
| Lightning | 1077.8 | 1081.3 | 1073.9 | 1077.7 |
| Lightning(Mixed) | 1014.9 | 1012.7 | 1013.2 | 1013.6 |
WSL2+NVIDIA-docker
| 手法 | 1回目 | 2回目 | 3回目 | 平均 |
|---|---|---|---|---|
| PyTorch | 463.3 | 471.8 | 466.8 | 467.3 |
| Lightning | 498.6 | 503.2 | 522.1 | 508 |
| Lightning(Mixed) | 446.6 | 430.9 | 446.1 | 441.2 |
詳細:Windows11でWSL2+nvidia-dockerでPyTorchを動かすのがすごすぎた
おそらくLinux上のほうがCUDAの最適化がかかりやすいのでしょうが、WindowsでもWSLを使って、Docker内で動かしたほうが単純に速いというメリットがあります。
Dockerを使った機械学習の例
例えば、次のようなオートエンコーダの例を考えましょう。
import torch
import torch.nn as nn
import torch.nn.functional as F
from torch.utils.data import DataLoader
import torchvision
import pytorch_lightning as pl
import argparse
from PIL import Image
import numpy as np
import os
class AutoEncoder(pl.LightningModule):
def __init__(self, opt):
super().__init__()
self.opt = opt
self.conv1 = nn.Conv2d(1, 16, 3, stride=2, padding=1)
self.conv1_bn = nn.BatchNorm2d(16)
self.conv2 = nn.Conv2d(16, 32, 3, stride=2, padding=1)
self.conv2_bn = nn.BatchNorm2d(32)
self.deconv1 = nn.ConvTranspose2d(32, 16, 3, stride=2, padding=1, output_padding=1)
self.deconv1_bn = nn.BatchNorm2d(16)
self.deconv2 = nn.ConvTranspose2d(16, 1, 3, stride=2, padding=1, output_padding=1)
self.cache_validation_batch = None
def forward(self, inputs):
x = F.relu(self.conv1_bn(self.conv1(inputs)))
x = F.relu(self.conv2_bn(self.conv2(x)))
x = F.relu(self.deconv1_bn(self.deconv1(x)))
x = torch.sigmoid(self.deconv2(x))
return x
def configure_optimizers(self):
optimizer = torch.optim.Adam(self.parameters(), lr=1e-3)
return optimizer
def training_step(self, train_batch, batch_idx):
x, _ = train_batch
x_pred = self.forward(x)
loss = F.l1_loss(x_pred, x)
self.log("train_loss", loss, prog_bar=False, logger=True)
return loss
def write_outputs(self, batch):
grid = torchvision.utils.make_grid(
batch[:64], nrow=8, value_range=(0.0, 1.0)
) # (C, H, W)
grid = grid.cpu().numpy().transpose([1, 2, 0]) # (H, W, C)
grid = (grid * 255.0).astype(np.uint8)
os.makedirs(self.opt.output_dir, exist_ok=True)
with Image.fromarray(grid) as img:
img.save(f"{self.opt.output_dir}/epoch_{self.current_epoch:03}.png")
def validation_step(self, val_batch, batch_idx):
x, _ = val_batch
x_pred = self.forward(x)
if batch_idx == 0:
# ここで直接吐くとDocker環境で「AssertionError: can only test a child process」という警告文が出る(処理は続く)のでキャッシュさせる
# ローカル環境でエラーは出ないので、Docker→ローカルのI/Oボトルネックと、マルチプロセスが噛み合っていないのかも?
#self.write_outputs(x_pred)
self.cache_validation_batch = x_pred
loss = F.l1_loss(x_pred, x)
self.log("val_loss", loss, prog_bar=True, logger=True)
def validation_epoch_end(self, outputs):
if self.cache_validation_batch is not None:
self.write_outputs(self.cache_validation_batch)
self.cache_validation_batch = None
class KMNISTModule(pl.LightningDataModule):
def __init__(self, opt):
super().__init__()
self.opt = opt
def prepare_data(self):
self.train_dataset = torchvision.datasets.KMNIST(
self.opt.data_dir, train=True, download=True,
transform=torchvision.transforms.ToTensor())
self.val_dataset = torchvision.datasets.KMNIST(
self.opt.data_dir, train=False, download=True,
transform=torchvision.transforms.ToTensor())
def train_dataloader(self):
return DataLoader(self.train_dataset, batch_size=256, num_workers=4, shuffle=True)
def val_dataloader(self):
return DataLoader(self.val_dataset, batch_size=256, num_workers=4, shuffle=False)
def main(opt):
model = AutoEncoder(opt)
cifar = KMNISTModule(opt)
if opt.gpus == 0:
train_flag = {"accelerator":"cpu"}
elif opt.gpus > 0:
train_flag = {"accelerator":"gpu", "devices":opt.gpus}
trainer = pl.Trainer(max_epochs=10, **train_flag)
trainer.fit(model, cifar)
if __name__ == "__main__":
parser = argparse.ArgumentParser(description="MNIST Training")
parser.add_argument("--data_dir", type=str, default="./data")
parser.add_argument("--ckpt_dir", type=str, default="./ckpt")
parser.add_argument("--output_dir", type=str, default="./output")
parser.add_argument("--gpus", type=int, default=0) # 0-> CPU, 1 -> use 1 gpu
opt = parser.parse_args()
main(opt)
これをDockerのボリュームマウントを使って動かすと
docker run -it --rm --gpus all -v $(pwd):/mnist some_images
root@118805f6912b:/# cd mnist
root@118805f6912b:/mnist# python kmnist_autoencoder.py
カレントディレクトリがDocker内の「/mnist」としてマウントされます。つまり、Docker内で訓練プログラムを動かしつつ、ログや途中生成結果、訓練済みモデルをWindows上へリアルタイムで吐き出すことが可能です。
SageMaker
本書の最後では、AWSのサービスの1つであるSageMaker、特にSageMaker Training Job(便利ではあるが使い方に癖のある)を独自にビルドしたコンテナを使って、自在に扱うまでの手順を紹介しています。本書の最後では、Training Jobを使って、YouTube上の動画をダウンロードしYOLOXを推論し、Bounding Boxをレンダリングした後の動画をOpenH264でエンコードする方法を例示しています。
元動画:https://www.youtube.com/watch?v=-SQhoG6PsKg
SageMakerに興味がある、使っている方には楽しめる内容になっていると思います。
目次
本書の目次は以下の通りです。
第1章:Docker入門
- なぜDockerなのか?
- CUDAのバージョン問題
- 論文の公式実装の環境依存問題
- Colabが本番運用に向かない問題
- WSL2導入
- WSL2上でのDocker-CEのインストール
- Dockerイメージとコンテナの概念の理解
- Dockerは複数の環境を切り替えられる
- インスタンスとしてのコンテナ
- 演習問題(1)
- Dockerイメージのビルド(docker build)
- WSL2におけるWindowsのファイルの扱い
- Dockerイメージを削除する(docker rmi)
- 事例:Pythonのイメージを作る
- コンテナへのファイルコピー(docker cp)
- 独自Dockerfileの作り方・考え方
- docker runのボリュームのマウントによるお手軽デバッグ
- 演習問題(2)
第2章:AWS S3/ECR
- AWSアカウントの登録
- MFAの設定
- 独自のアカウント名をつける
- IAMユーザー
- Amazon S3
- ブラウザ上からのS3操作
- AWS CLIインストール
- aws configure
- AWS CLIによるS3のオブジェクト操作
- アップロード・ダウンロード(s3 cp)
- ファイル移動や名前変更(s3 mv)
- ファイルの削除(s3 rm)
- ディレクトリ同期でコピー削除を同時にこなす(s3 sync)
- 演習問題(1)
- Boto3の導入と認証
- Boto3によるS3オブジェクトの操作
- バケットのバージョニング
- バージョニング環境下でのファイル操作
- バケットのライフサイクルルール
- Amazon ECR
- 演習問題(2)
第3章:SageMaker Notebook
- SageMaker ノートブックとは
- SageMakerノートブックとColabの違い
- SageMakerノートブックの料金計算
- ノートブックインスタンスの初期化
- ノートブックインスタンスの動作確認
- ノートブックインスタンスとS3の連携
- ノートブックインスタンスとECRの連携
- まとめ
第4章:SageMaker Training Job
- SageMaker Training job
- ケース1:MNIST
- ケース2:シェルファイルの実行
- ケース3:独自DockerイメージでTraining Jobを動作させる
- ケース4:実行ファイルが複数ある場合
- ケース5:ハイパーパラメータと環境変数
- ケース6:Training Jobの並列実行
- ケース7:訓練データをS3から入力する
- ケース8:スポットインスタンスによる訓練
- ケース9:Training Jobのローカルモード
- ケース10:Training Jobを使用したOpenH264による動画エンコード
- ケース11:Stable Diffusion
- 演習問題
模範解答
試し読み・通販
試し読みはこちらから
https://koshian2.booth.pm/items/4150087
通販はこちらからどうぞ