はじめに
前回の記事では画像の前処理方法であるバンドパスフィルターを解説しました。
実験系研究者のための画像処理技術~バンドパスフィルターを用いた画像処理をPythonで実装する~
ただ、フィルター処理の方法を解説しただけで、実際に形状分布をどのように測定するかは説明しませんでした。
なので、今回は、OpenCV-Pythonを用いた粒子の形状分布測定を解説します。
解析対象として、Wikimedia Commonsから持ってきた以下の画像を用いました。

作者 Zephyris [CC BY-SA 3.0 (https://creativecommons.org/licenses/by-sa/3.0) または GFDL (http://www.gnu.org/copyleft/fdl.html)], ウィキメディア・コモンズより(リンク)
ガラスコロイド粒子の光学顕微鏡像です。これを色々と分析していこうと思います。
解析にはJupyter-Notebook(Python=3.6.5, opencv-python=3.4.1.15, scipy=1.1.0)を用いています。
使用するパッケージ
import cv2
import numpy as np
import scipy.ndimage as ndimage
from scipy.optimize import curve_fit
import matplotlib.pyplot as plt
cv2がOpenCV-pythonのパッケージです。
OpenCV-pythonは以下のようにすれば一発でインストールできます。
$ pip install opencv-python
また、画像のFIll_Holes処理(後述)と分布関数のフィッティングにscipyを用います。
scipyも同様に以下のようにすればインストールできます。
$ pip install scipy
画像の読み込み
画像は以下のようにすれば読み込めます。
img_raw = cv2.imread('800px-ColloidCrystal_10xBrightField_GlassInWater.jpg', 1)
img = cv2.cvtColor(img_raw, cv2.COLOR_BGR2GRAY)
パスの後ろの数字は、1の時は3チャンネル(BGR)、0の時は1チャンネル(グレースケール)として読み込むことを示しています。
読み込んだ画像は、img_rawの中に[高さ, 幅, (blue, green, red)]のnumpy_arrayとして読み込まれています。
色空間の変換にはcv2.cvtColorを用います。詳細はこちら。
画像の2値化
2値化前にガウシアンブラーでぼかしてノイズ除去を行っています。
また、2値化後の粒子サイズが元画像と比較して少し小さくなってしまったので、モルフォロジー変換で少し膨張させています。
(ここはThresholdの値で調整した方がいいです)
また、粒子のサイズがかなり小さいので、画像を前もって3倍に拡大しています。
(今回は画像がもともときれいなのでフィルターはほぼ使いません)
# 画像の高さ, 幅を取得
h, w = img.shape
# 画像の前処理(拡大)
mag = 3
img = cv2.resize(img, (w*mag, h*mag))
# 画像の前処理(ぼかし)
img_blur = cv2.GaussianBlur(img,(5,5),0)
# 2値画像を取得
ret,th = cv2.threshold(img_blur,0,255,cv2.THRESH_BINARY+cv2.THRESH_OTSU)
# モルフォロジー変換(膨張)
kernel = np.ones((3,3),np.uint8)
th = cv2.dilate(th,kernel,iterations = 1)
# 画像を保存
cv2.imwrite('thresholds.png', th)
Fill Holes処理
以上の2値化はかなりうまくいっているように見えますが、一部で粒子がドーナツ状になってしまっています。
これを塗りつぶすために、Fill Holes処理を行います。
Fill Holes処理はOpenCVには実装されていない(たぶん)ので、scipyのndimageというモジュールに実装されているのを使います。使い方は簡単です。
# Fill Holes処理
th_fill = ndimage.binary_fill_holes(th).astype(int) * 255
cv2.imwrite('thresholds_fill.png', th_fill)
Fill Holes処理後は、下のようにドーナツ状の穴が消えます。
境界検出と描画
これで2値化が終わったので、OpenCVを用いて境界検出を行います。
境界検出の詳細はチュートリアルを参照。
# 境界検出と描画
__, cnt, __ = cv2.findContours(th_fill.astype(np.uint8), cv2.RETR_TREE, cv2.CHAIN_APPROX_SIMPLE)
img_raw = cv2.resize(img_raw, (w*mag, h*mag))
img_cnt = cv2.drawContours(img_raw, cnt, -1, (0,255,255), 1)
cv2.imwrite('cnt.png', img_cnt)
また、cv2.drawContoursで、元画像の上に検出された境界を描画します。
検出結果は以下のような感じです。

検出した粒子の境界はcntの中にリスト形式で保存されています。
ちなみに総検出数は24908個でした。この数を手動で解析するのは不可能ですね。
なので、これからすべての粒子の性質を自動で解析していきます。
粒子の形状を測定
今回は、以下の3つの性質を測定します。
- 面積(Area)
- 円形度(Circularity)
- 円相当径(Equivalent Diameter)
円形度は0~1までの間を取る値で、1であれば完全に円であることを示します。
# 面積、円形度、等価直径を求める。
Areas = []
Circularities = []
Eq_diameters = []
for i in cnt:
#面積(px*px)
area = cv2.contourArea(i)
Areas.append(area)
#円形度
arc = cv2.arcLength(i, True)
circularity = 4 * np.pi * area / (arc * arc)
Circularities.append(circularity)
#等価直径(px)
eq_diameter = np.sqrt(4*area/np.pi)
Eq_diameters.append(eq_diameter)
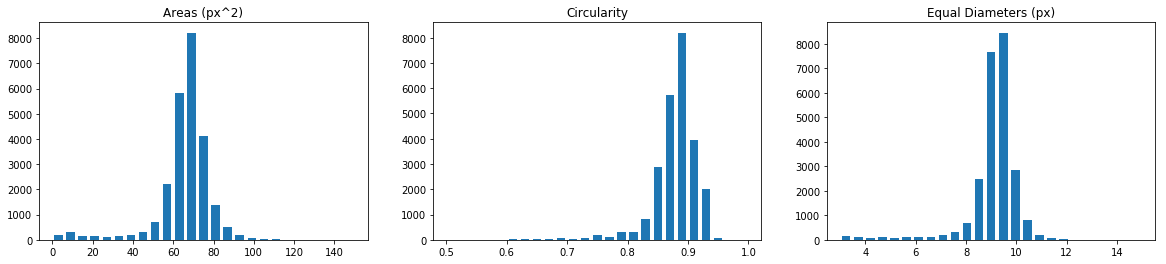
これでそれぞれの粒子の性質がわかったので、matplotlibを用いて以下のようにヒストグラムを作成します。
fig = plt.figure(figsize=(8,6))
plt.subplot(2,2,1)
plt.title("Areas (px^2)")
plt.hist(Areas, bins=25, range=(0,150), rwidth=0.7)
plt.subplot(2,2,2)
plt.title("Circularity")
plt.hist(Circularities, bins=25, range=(0.5,1), rwidth=0.7)
plt.subplot(2,2,3)
plt.title("Equal Diameters (px)")
plt.hist(Eq_diameters, bins=25, range=(3.0, 15.0), rwidth=0.7)
最終的に得られるヒストグラムは以下のようになります。
粒度分布の解析
上の3つの測定値のうち、粒径に相当する円相当径を詳しく解析します。
といっても、ヒストグラムをガウシアンでフィッティングするだけです。
scipy.optimizeに入っているcurve_fitをつかいます。
from scipy.optimize import curve_fit
# numpyでヒストグラムを作成
hist = np.histogram(Eq_diameters, bins=30, range=(3.0, 15.0))
# ヒストグラムの中央値の配列を作成
diff = np.diff(hist[1])
hist_center = hist[1][:-1] + diff
# フィッティング用のガウス関数を作成
def Gaussian(x, *params):
amp = params[0]
wid = params[1]
ctr = params[2]
y = amp * np.exp( -((x - ctr)/wid)**2)
return y
# ガウシアンでフィッティング
guess = [5000, 4, 9]
params, cov = curve_fit(Gaussian, hist_center, hist[0], p0=guess)
# 強度, 半値幅, 平均値を表示
fmhm = 2 * np.sqrt(2*np.log(2)) * params[1]
print("Amplitude = "+str(params[0]))
print("FMHM = "+str(fmhm))
print("Mean = "+str(params[2]))
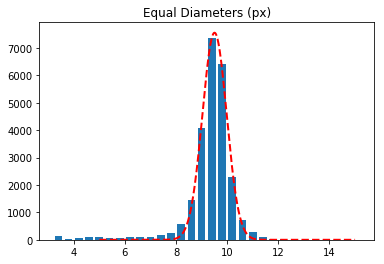
# ヒストグラムと分布関数(ガウシアン)を重ねて表示
plt.title("Equal Diameters (px)")
plt.bar(hist_center, hist[0], width=0.3)
x = np.linspace(5,15, 1000)
plt.plot(x, Gaussian(x, *params), c="red",lw=2, ls="--")
また、フィッティングによって得られたパラメーターは以下の通りです
Amplitude(強度) = 7561.472139684951
FMHM(半値幅) = 1.5764610490222037
Mean(平均値) = 9.505377793721298
半値幅と平均値はpixel単位になっているので、スケールを何らかの方法で求めて、実際の単位に変換する必要があります。
まとめ
以上のように、OpenCVとScipyを用いてコロイド粒子の基本的な粒度分布測定を行いました。
OpenCVを用いた解析は非常に幅が広く、ここで示した以外にも様々な解析を行うことができます。
今後は、応用的な画像解析も紹介していけたらなと思います。
また、こんな解析ができないかという提案があったら教えていただけると嬉しいです。
それでは!