はじめに
バンドパスフィルターは、特に実験的に得られた顕微鏡画像(光顕, SEM, AFMなど……)から物体を認識するための前処理として役立ちます。
今までImageJのFFT Filterを使っていたのですが、画像処理をすべて自動化したかったのでPythonで書いてみました。
参考にしたページは以下の通りです。
フーリエ変換 — OpenCV-Python Tutorials 1 documentation
画像処理におけるフーリエ変換④〜pythonによるフィルタ設計〜
5.周波数領域における画像処理
実装はPythonで行いましたが、画像処理ソフトのImageJも説明に使います。
とても役立つソフトで、しかもオープンソース(!)なので、画像処理をやりたいなと思っている方はぜひダウンロードしてください。
ついでなので、いつも自分が行っている画像処理の流れをImageJで簡単に説明してから、Pythonでの実装に関してお話しします。
一般的な画像処理の手法
ImageJのSample ImagesにあるCell_colonyを例として使います。
こんな画像です。

この中のコロニーの数を数えたり、形状を測定したりしたいですね。
ふつうは、スムージングでノイズを処理して、適当な方法で二値化することを考えます。
では、やってみましょう。
この画像をImageJで開いて、Process=>Smoothでスムージングをしてから、Image=>Adjust=>Thresholdsから大津の二値化(Otsu)を行った結果がこちらになります。

案外うまくいってますね。ここで満足してしまう人も多いのではないでしょうか。
ただ、よ~くみてみると、左上では小さなコロニーの数が少ないのに対して、右下のコロニーは数多く検出されています。どういうことでしょう?
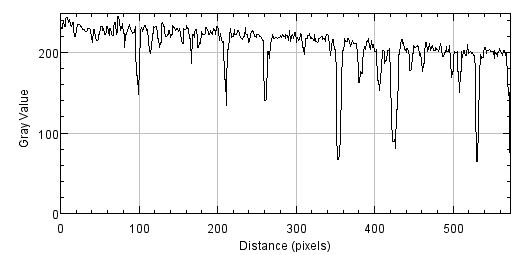
では、画像の左上から右下へ直線をひいて、直線上の輝度変化をプロットしてみます。
原点が画像の左上、x=573が右下を表しています。
わずかながらですが、輝度の値が減少していっている様子がわかります。
これは、画像全体に輝度の分布(バックグラウンド)が生じていることを示しています。
こういった、なだらかで連続的な変化はスムージングで取り除くことができません。
ImageJではこのようなバックグラウンド変化を取り除くSubtract Background...という処理がありますが、バンドパスフィルターを使っても取り除くことができます。
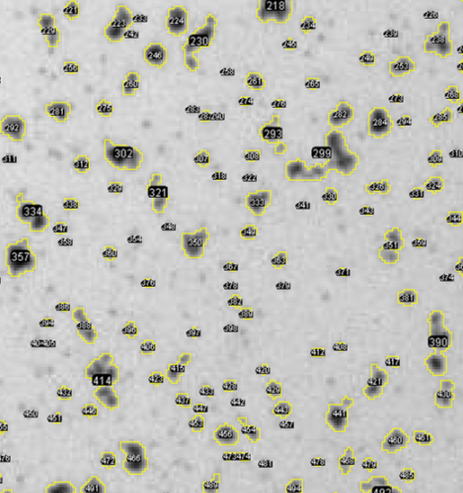
また、Thresholdsで二値化した画像をAnalyze=>Analyze Particles...で境界検出してみます。
検出された境界を元画像上にオーバーレイで表示させたものが以下になります。
近い別々のコロニーが、1つのコロニーとして認識されてしまっています。
このような連結を分離する方法としては分水嶺処理、Watershedが有名です。しかし、バンドパスフィルターもけっこういい働きをしてくれます。
バンドパスフィルターの効果
バンドパスフィルターとは、いわゆるハイパスフィルターとローパスフィルターを組み合わせたものです。
フーリエ変換によって得られた画像の周波数成分のうち、ある閾値(Low)より大きく、ある閾値(High)より小さな領域以外をマスクした後、逆フーリエ変換を行って画像を復元します。
例えば先ほどのCell_colonyを、ImageJを用いてProcess=>FFT=>FFTでフーリエ変換すると以下のようになります。
この画像のうち、原点に近い部分が画像の低周波成分を、遠い部分が高周波成分の情報をふくんでいます。
ここで、以下のようなフィルターを用意します。
この画像の黒は0、白は1を表します。この画像をFFTの結果と掛け合わせることで、周波数成分の中のある領域だけを取り出します。
この処理はImageJではProcess=>FFT=>Bandpass filter...で行えます。
最終的に、このバンドパスフィルターで処理した結果が以下のようになります。
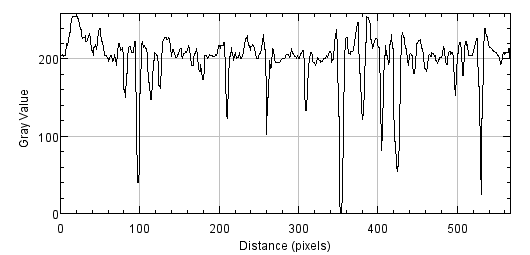
さて、では先ほどと同様に左上から右下への輝度変化を見てみましょう。
左上から右下へのなだらかな輝度勾配はなくなっていますね。
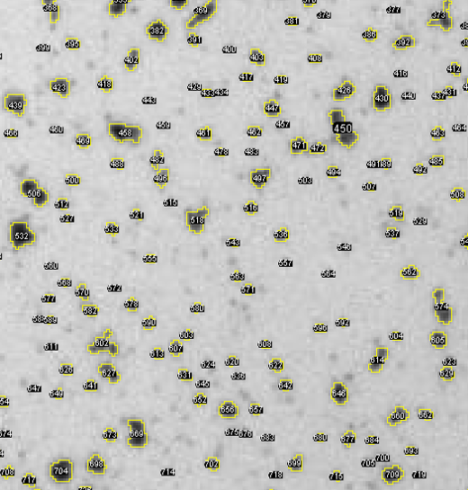
では、最後にこの画像を二値化して、領域検出をしてみた結果が以下になります。
バンドパスフィルター適用前と比べると、コロニーが良く分離されていることが分かります。
以上のように、画像処理、特に粒子状の物質の自動検出に関してバンドパスフィルターは大きな威力を発揮しますが、画像処理ライブラリとしていつも使っているOpenCVには実装されていません。
なので、自分で作ることにしました。
Pythonでの実装
ImageJのバンドパスフィルターは内部でちょっと変わったことをやっているらしく、いまいちよくわからなかったので自己流で実装しました。
こんな感じになります。
def MakeFilter(img, high, low, isShowFilter):
#バンドパスフィルターの作成
h,w = img.shape
filter_low = np.zeros((h,w))
filter_high = np.zeros((h,w))
center = np.array([h/2, w/2], dtype=np.uint16)
R = high/2
r = low/2
for i in range(0,w):
for j in range(0,h):
dist_R = np.sqrt(R * R) - np.sqrt(((i-center[1])*(i-center[1]) + (j-center[0])*(j-center[0])))
dist_r = -np.sqrt(r * r) + np.sqrt(((i-center[1])*(i-center[1]) + (j-center[0])*(j-center[0])))
filter_low[j][i] = (1+math.erf(dist_r/(r/2)))/2
filter_high[j][i] = (1+math.erf(dist_R/(R/2)))/2
filter_matrix = filter_low + filter_high
filter_matrix = filter_matrix - np.min(filter_matrix)
filter_matrix = filter_matrix/np.max(filter_matrix)
#isShowFilterがTrueの時はフィルターを表示する。
if(isShowFilter):
fig = plt.figure()
plt.imshow(filter_matrix)
plt.title("BandPassFilter, high="+str(high)+", low="+str(low))
plt.colorbar()
fig = plt.figure()
plt.plot(np.linspace(0, w-1, w), filter_low[int(h/2),:])
plt.plot(np.linspace(0, w-1, w), filter_high[int(h/2),:])
plt.plot(np.linspace(0, w-1, w), filter_matrix[int(h/2),:])
plt.title("2D-plot at y=0, high="+str(high)+", low="+str(low))
return filter_matrix
def BandPassFilter(img, filter_matrix, isShowImage):
#フーリエ変換
ifimg = np.fft.fft2(img)
ifimg = np.fft.fftshift(ifimg)
#逆フーリエ変換
ifimg = ifimg*filter_matrix
ifimg = np.fft.fftshift(ifimg)
ifimg = np.fft.ifft2(ifimg)
ifimg = ifimg.real
ifimg = ifimg - np.min(ifimg)
ifimg = (ifimg/np.max(ifimg)) * 255
if(isShowImage):
fig = plt.figure()
plt.title("Result")
plt.imshow(ifimg.astype(np.uint8))
plt.colorbar()
return ifimg.astype(np.uint8)
MakeFilterがフィルターを作る関数、BandPassFilterがフィルターを画像に適用する関数です。
以下のように実行してみます。
(最後の引数にTrueを渡すとそれぞれフィルターと処理後画像を出力します)
import numpy as np
import cv2
import matplotlib.pyplot as plt
import math
img = cv2.imread('Cell_colony.png', 0)
bpfilter = MakeFilter(img, 200, 50, True)
bpf_img = BandPassFilter(img, bpfilter, True)
ImageJの再現、とまではいきませんが、そこそこうまくできたんじゃないでしょうか。
フィルターの設計も自分の好きなようにやれるのがいいですね、今回は誤差関数(math.erf())を使っていますが、他の分布を使ってみても面白そうです。
まとめ
ImageJでのバンドパスフィルターを使った顕微鏡画像の解析のやり方と、Pythonを用いたバンドパスフィルターの実装例を紹介しました。
大津の2値化や領域検出はOpenCVにすでに実装されているので、これでImageJで行った画像解析はすべてPythonで行えるようになりました。
(OpenCV-pythonでの2値化と領域検出のやり方はチュートリアルを参考にしてください)
顕微鏡画像の自動解析は時間の節約になるだけでなく、目視での判別につきものの実験者によるバイアス(偏見)を取り除くことにもつながります。
ぜひぜひ活用していきましょう!