この記事はbioinformatics2019アドベントカレンダー用の記事でした
今年はじめてbioinfoアドベントカレンダーに登録したのですが、全く間に合いませんでした。
すっかり穴を開けてしまいましたので、できたところまで公開します。最後の方におまけもあります。
なお、この記事は必ずしも学術的に正確なものではなく、プログラミングの楽しさやグラフの美しさを主題に焦点を当てたものですのでご容赦ください。詳しい人のツッコミは歓迎します
がんゲノムのデータがなかなか手に入らないと思ったら
がんのゲノムがどうなってるか知りたい人はたくさんいると思います。でも実際には、なかなかがんのゲノムを観察するわけにはいきません。それにはいくつかの理由があります。まず、がんは人間の体の一部です。患者さんが、がんの遺伝子のデータを提供したいと思わなければなりません。次に、調べるべきがんの組織があったとして、シークエンサーが可動していて、バイオインフォできる人がいて、予算もあるという施設はそんなに多くはないでしょう。またそういった条件がすべてクリアされていたとしても、解析できる検体の数は非常に限られるのが現状でしょう。
TCGAのデータちょっとつらい
そこで、みんなTCGAにデータを取りにいくことを考えると思います。TCGAとは、The Cancer Genome Atlas (TCGA) というグループで、がん組織のシークエンスデータを公共に公開しています。でも、これが意外と大変ですよね。シークエンスの生データって1ファイル何GBも容量あります。さらにダウンロードには申請と許可が必要です。早い話、個人がお家でダウンロードするのは難しいと思います。
 (画像はTCGAのサイトより)
(画像はTCGAのサイトより)
しかも、仮に生データをダウンロードできたとしても、マッピングを自分で行わなければなりません。今はSTARやhisat2など素晴らしいソフトや、GalaxyやCWLなどワークフローがあるから、メモリ128GB積んだRyzenの自作PCとか用意して、マッピングをすること自体は可能かもしれませんが、その解析結果が正しいかどうかなんてちょっと素人にはわかりません……。だから、そういうのは、できれば避けたいところです。もちろん融合遺伝子を探したいとか、目的がはっきりしている人には大きな意味があると思います。
そこでcBioPortalですよ
なので、素直に考えたら、cBioPortalのデータを見に行くしかないと思うんですよね。ひょっとしたら中国あたりに、cBioPortalぐらいの規模のデータベースが、私が知らないだけで隠されているのかも知れませんが、今のところ私はそういうものを知らないので、cBioPortalが地球最大のデータベースということになるかと思います。
cBioPortalの素晴らしいところはたくさんあります。フロントエンドも素晴らしくて、Webアプリとして使うこともできます。
 (悪性黒色腫についてcBioPortalでOncoPlotを表示してみたところ)
(悪性黒色腫についてcBioPortalでOncoPlotを表示してみたところ)
 (変異の意義もOncoKBなどのデータベースと照らし合わせることができる)
(変異の意義もOncoKBなどのデータベースと照らし合わせることができる)
 (肝臓がんの全体のデータを表示したところ。この画面から任意の層別化を行い、生存曲線を描くことも可能である)
(肝臓がんの全体のデータを表示したところ。この画面から任意の層別化を行い、生存曲線を描くことも可能である)
cBioPortalはAPIも提供しており、APIにアクセスすることで効率的にデータを検索することができます。dockerを使えば比較的簡単にcBioPortalサーバーを起動することができますので、そうやって使えば大量アクセスを気にする必要もありません。
 (素晴らしいAPIが提供されている)
(素晴らしいAPIが提供されている)
 (Dockerを使えばcBioPortalを自分のマシンで動かすことができる。)
(Dockerを使えばcBioPortalを自分のマシンで動かすことができる。)
でも、cBioPortalの一番すごいところは、タブ区切りテキスト形式のデータをGithubのdatahubというリポジトリで公開しているところです。タブ区切りテキストならば、エクセルでも開くことができるので、誰でもがんゲノムのデータを見ることができるわけです。
結論 → Datahubのデータを見ればよい。
(※cBioPortalは、プロモーター領域の変異などは登録されていないという弱点もあります)
cBioPortalからPublicのデータを全部ダウンロードする

こちらのリポジトリの内容をダウンロードします。READMEを見るとやり方が書いてありますが、[Git LFS](Git Large File Storage (LFS) ) というコマンドラインツールが必要です。Git LFSをインストールして、READMEの通りにします。
git lfsの設定
git lfs install --skip-repo --skip-smudge
リポジトリのクローン
git clone https://github.com/cBioPortal/datahub.git
cd datahub
git lfs install --local --skip-smudge
public以下のファイルを全て取得する
git lfs pull -I public
ディレクトリのサイズは全部で 90GB もあるのでダウンロードには時間がかかります。しかしsraファイルを直接ダウンロードして自分で処理する手間や時間を考えたらほぼ 0GBに等しいと言って差し支えないでしょう(強弁)。
がんの変異、RNA発現、コピーナンバー、メチル化のデータをプロットする
さて、このように大量にデータを入手してみたら、なんとなく全体像を可視化してみたいと思わないでしょうか?
突然ですが私はRubyが大好きです。Rubyでグラフを描くためのツールGR.rbを自作しています。(正確にはffiを用いたGR FrameworkのRubyバインディングです。) そこで自作のツールを使用して、がんゲノムのデータを大雑把に可視化してみることにしました。

https://qiita.com/kojix2/items/456259f40d5563005cd1
また、データフレームの代用として、ApachArrowのRubyバインディングであるRedArrowを使用しています。クラスタリングにはRumaleとnumo-linalgを使用しました。いずれもとてもマイナーなツールですので、たぶんこの記事を読んでもよほどRubyについて詳しい方以外は環境を整える時点で大変で、なかなか再現できないだろうなと思います。Rubyでやるメリットはあまりなくて、私の場合は単にRubyが好きだからそうしています。Ruby以外では、JuliaとGR.jl、Pythonとpython-grなどを使うと、比較的簡単に同じような結果が得られると思います。
でもねえ、やっぱり人間は自分で作ったツールを使いたくなる生き物だから、たとえ不出来でも自分でツールを作った人だけに見えてくる世界もあると思うんだよね。私はそう信じている…
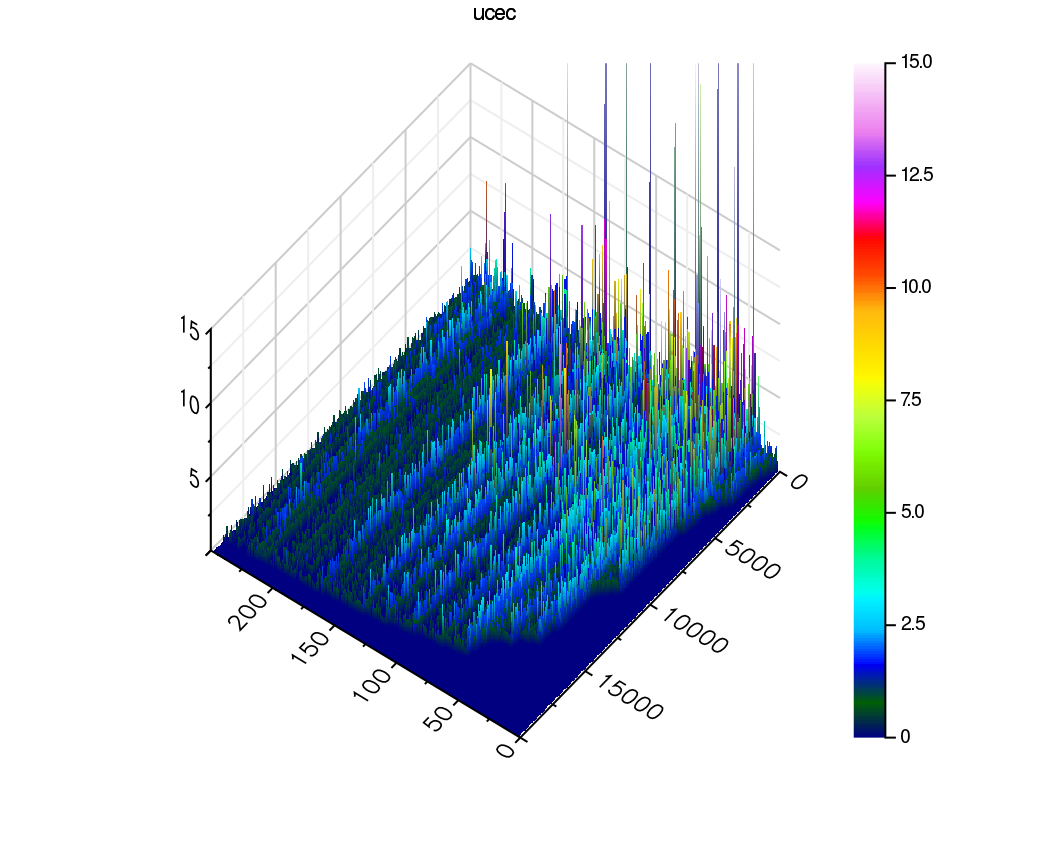
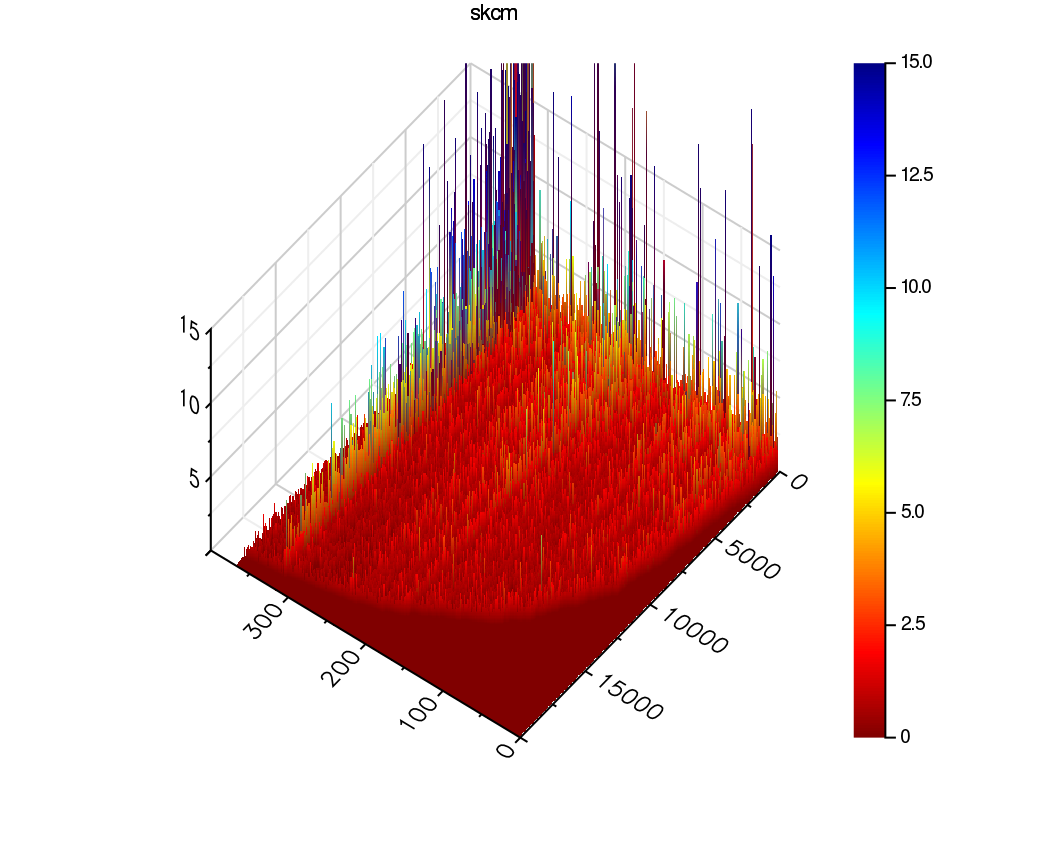
Mutation
- X軸 サンプル
- Y軸 遺伝子(ここでは20000にしました)
- Z軸 1遺伝子のなかのmutationの数
# frozen_string_literal: true
require 'fileutils'
require 'gr/plot'
require 'arrow'
require 'progress_bar/core_ext/enumerable_with_progress'
FileUtils.mkdir_p 'mutation'
def read_tsv(path)
Arrow::Table.load(path,
format: 'csv',
delimiter: "\t".ord)
end
CANCER_TYPES = %w[paad stad kirp laml thym meso uvm hnsc lihc kich chol pcpg blca
coadread ov sarc prad lgg lusc thca esca ucs cesc gbm skcm kirc
tgct brca ucec dlbc acc luad].freeze
CANCER_TYPES.each_with_progress do |cancer_type|
path = '../datahub/public/' + cancer_type + '_tcga/data_mutations_extended.txt'
df = read_tsv(path)
p cancer_type: cancer_type, n_rows: df.n_rows
next if cancer_type == 'brca'
barcodes = df.select_columns('Tumor_Sample_Barcode')[0].uniq
sampleids = df.select_columns('Hugo_Symbol')[0].uniq
result = Array.new(20_000) { Array.new(barcodes.size, 0) }
df.select_columns('Hugo_Symbol', 'Tumor_Sample_Barcode', 'Consequence').each_record do |record|
id, sb, co = record.to_a
unless co == 'synonymous_variant'
result[sampleids.find_index(id)][barcodes.find_index(sb)] += 1
end
end
opts = { title: cancer_type, size: [200, 160], xflip: true, yflip: true }
GR.heatmap(result, opts.merge(colormap: 6))
GR.savefig('mutation/' + cancer_type + '_0.png')
GR.contour(result, opts)
GR.savefig('mutation/' + cancer_type + '_1.png')
GR.surface(result, opts.merge(zlim: [0, 15], colormap: 4))
GR.savefig('mutation/' + cancer_type + '_2.png')
GR.surface(result, opts.merge(zlim: [0, 15], colormap: 43))
GR.savefig('mutation/' + cancer_type + '_3.png')
GR.surface(result, opts.merge(zlim: [0, 15], colormap: 29))
GR.savefig('mutation/' + cancer_type + '_4.png')
end
ACC (Adrenocortical carcinoma) 副腎皮質がん
BLCA (Bladder Urothelial Carcinoma) 膀胱がん・尿路上皮がん
BRCA (Breast invasive carcinoma) 乳がん
※データ量が多すぎるのか、バーチャルメモリエラーと表示されて出力できず。
CESC (Cervical squamous cell carcinoma and endocervical adenocarcinoma) 子宮頸がん
※なぜかこのスタディのデータだけ発現変異の多い順にまとめられていないようです
CHOL (Cholangiocarcinoma) 胆管がん
COAD READ (Colon adenocarcinoma) (Rectum adenocarcinoma) 大腸がん・直腸がん
※cBioPortalでは、COADとREADをセットにしてCOADREADとして扱っています。
DLBC (Lymphoid Neoplasm Diffuse Large B-cell Lymphoma) リンパ腫・びまん性大細胞型B細胞性リンパ腫
ESCA (Esophageal carcinoma) 食道がん
GBM (Glioblastoma multiforme) 膠芽腫
HNSC (Head and Neck squamous cell carcinoma) 頭頚部がん
KICH (Kidney Chromophobe)
KIRC (Kidney renal clear cell carcinoma)
KIRP (Kidney renal papillary cell carcinoma)
LAML (Acute Myeloid Leukemia) 急性骨髄性白血病
LGG (Lower Grade Glioma) 神経膠腫
LIHC (Liver hepatocellular carcinoma) 肝臓がん
LUAD (Lung adenocarcinoma) 肺腺がん
LUSC (Lung squamous cell carcinoma) 肺扁平上皮がん
MESO (Mesothelioma) 中皮腫
OV (Ovarian serous cystadenocarcinoma) 卵巣漿液性のう胞腺腫
PAAD (Pancreatic adenocarcinoma) 膵臓がん
※ 1つだけ飛び抜けて変異(mutation)が多い症例があります
PCPG (Pheochromocytoma and Paraganglioma) 褐色細胞腫 傍神経節腫
PRAD (Prostate adenocarcinoma) 前立腺がん
SARC (Sarcoma) 肉腫
SKCM (Skin Cutaneous Melanoma) 悪性黒色腫
STAD (Stomach adenocarcinoma) 胃がん
TGCT (Testicular Germ Cell Tumors) 精巣がん
THCA (Thyroid carcinoma) 甲状腺がん
THYM (Thymoma) 胸腺腫
UCEC (Uterine Corpus Endometrial Carcinoma) 子宮体がん
UCS (Uterine Carcinosarcoma) 子宮肉腫
UVM (Uveal Melanoma) ぶどう膜黒色腫
RNA expression
# frozen_string_literal: true
require 'fileutils'
require 'gr/plot'
require 'arrow'
require 'progress_bar/core_ext/enumerable_with_progress'
FileUtils.mkdir_p 'rna'
def read_tsv(path)
Arrow::Table.load(path,
format: 'csv',
delimiter: "\t".ord)
end
CANCER_TYPES = %w[paad stad kirp laml thym meso uvm hnsc lihc kich chol pcpg blca
coadread ov sarc prad lgg lusc thca esca ucs cesc gbm skcm kirc
tgct brca ucec dlbc acc luad].freeze
CANCER_TYPES.each_with_progress do |cancer_type|
path = '../datahub/public/' + cancer_type + '_tcga/data_RNA_Seq_v2_expression_median.txt'
df = read_tsv(path)
z = df.select_columns(2..-1).raw_records
GR.contour(z, size: [120, 100], title: cancer_type, colormap: 4, zlim: [0, 600_000])
GR.savefig('rna/' + cancer_type + '_contour.png')
GR.surface(z, size: [120, 100], title: cancer_type, colormap: 4, zlim: [0, 600_000])
GR.savefig('rna/' + cancer_type + '_surface.png')
end
ACC (Adrenocortical carcinoma) 副腎皮質がん
BLCA (Bladder Urothelial Carcinoma) 膀胱がん・尿路上皮がん
BRCA (Breast invasive carcinoma) 乳がん
※メモリ不足表示
CESC (Cervical squamous cell carcinoma and endocervical adenocarcinoma) 子宮頸がん
CHOL (Cholangiocarcinoma) 胆管がん
COAD READ (Colon adenocarcinoma) (Rectum adenocarcinoma) 大腸がん・直腸がん
DLBC (Lymphoid Neoplasm Diffuse Large B-cell Lymphoma) リンパ腫・びまん性大細胞型B細胞性リンパ腫
ESCA (Esophageal carcinoma) 食道がん
GBM (Glioblastoma multiforme) 膠芽腫
HNSC (Head and Neck squamous cell carcinoma) 頭頚部がん
KICH (Kidney Chromophobe)
KIRC (Kidney renal clear cell carcinoma)
KIRP (Kidney renal papillary cell carcinoma)
LAML (Acute Myeloid Leukemia) 急性骨髄性白血病
LGG (Lower Grade Glioma) 神経膠腫
LIHC (Liver hepatocellular carcinoma) 肝臓がん
LUAD (Lung adenocarcinoma) 肺腺がん
LUSC (Lung squamous cell carcinoma) 肺扁平上皮がん
MESO (Mesothelioma) 中皮腫
OV (Ovarian serous cystadenocarcinoma) 卵巣漿液性のう胞腺腫
PAAD (Pancreatic adenocarcinoma) 膵臓がん
PCPG (Pheochromocytoma and Paraganglioma) 褐色細胞腫 傍神経節腫
PRAD (Prostate adenocarcinoma) 前立腺がん
SARC (Sarcoma) 肉腫
SKCM (Skin Cutaneous Melanoma) 悪性黒色腫
STAD (Stomach adenocarcinoma) 胃がん
TGCT (Testicular Germ Cell Tumors) 精巣がん
THCA (Thyroid carcinoma) 甲状腺がん
THYM (Thymoma) 胸腺腫
UCEC (Uterine Corpus Endometrial Carcinoma) 子宮体がん
UCS (Uterine Carcinosarcoma) 子宮肉腫
UVM (Uveal Melanoma) ぶどう膜黒色腫
コピーナンバー
今回、一番驚いたのはコピーナンバーでした。想像以上に癌腫ごとに特徴が現れていることがわかります。
色違いで2種類の画像を作ったので掲載します。
# frozen_string_literal: true
require 'fileutils'
require 'gr/plot'
require 'arrow'
require 'progress_bar/core_ext/enumerable_with_progress'
FileUtils.mkdir_p 'copy_number'
def read_tsv(path)
Arrow::Table.load(path,
format: 'csv',
delimiter: "\t".ord)
end
require 'numo/linalg'
require 'rumale'
def cluster(z, a, b)
samples = Numo::DFloat.cast(z)
normalizer = Rumale::Preprocessing::StandardScaler.new
samples_std = normalizer.fit_transform(samples.transpose)
samples_std = samples_std.transpose
if a == 1
xindex = true
else
gmm = Rumale::Clustering::KMeans.new(n_clusters: a)
pca = Rumale::Decomposition::PCA.new(n_components: 50)
samples_std_pca = pca.fit_transform(samples_std)
cluster_ids = gmm.fit_predict(samples_std_pca)
xindex = cluster_ids.sort_index
end
if b == 1
yindex = true
else
gmm2 = Rumale::Clustering::KMeans.new(n_clusters: b)
pca2 = Rumale::Decomposition::PCA.new(n_components: 50)
samples_std_pca2 = pca2.fit_transform(samples_std.transpose)
cluster_ids2 = gmm2.fit_predict(samples_std_pca2)
yindex = cluster_ids2.sort_index
end
z3 = Numo::DFloat.cast(z)
z3 = z3[xindex, yindex]
z3
end
CANCER_TYPES = %w[paad stad kirp laml thym meso uvm hnsc lihc kich chol pcpg blca
coadread ov sarc prad lgg lusc thca esca ucs cesc gbm skcm kirc
tgct brca ucec dlbc acc luad].freeze
CANCER_TYPES.each_with_progress do |cancer_type|
path = '../datahub/public/' + cancer_type + '_tcga/data_CNA.txt'
df = read_tsv(path)
p cancer_type: cancer_type, n_rows: df.n_rows, time: Time.now
next if cancer_type == "brca"
z = df.select_columns(2..-1).raw_records
z = cluster(z, 1, 10)
GR.histogram(z.flatten, size: [120, 100])
GR.heatmap(z, size: [120, 100])
GR.savefig('copy_number/' + cancer_type + '_heatmap0.png')
GR.heatmap(z, size: [120, 100], colormap: 11)
GR.savefig('copy_number/' + cancer_type + '_heatmap1.png')
GR.surface(z, size: [120, 100], title: cancer_type)
GR.savefig('copy_number/' + cancer_type + '_surface0.png')
GR.surface(z, size: [120, 100], title: cancer_type, colormap: 4)
GR.savefig('copy_number/' + cancer_type + '_surface1.png')
GR.surface(z, size: [120, 100], title: cancer_type, colormap: 11)
GR.savefig('copy_number/' + cancer_type + '_surface2.png')
end
ACC (Adrenocortical carcinoma) 副腎皮質がん
BLCA (Bladder Urothelial Carcinoma) 膀胱がん・尿路上皮がん
BRCA (Breast invasive carcinoma) 乳がん
※なし
CESC (Cervical squamous cell carcinoma and endocervical adenocarcinoma) 子宮頸がん
CHOL (Cholangiocarcinoma) 胆管がん
COAD READ (Colon adenocarcinoma) (Rectum adenocarcinoma) 大腸がん・直腸がん
DLBC (Lymphoid Neoplasm Diffuse Large B-cell Lymphoma) リンパ腫・びまん性大細胞型B細胞性リンパ腫
ESCA (Esophageal carcinoma) 食道がん
GBM (Glioblastoma multiforme) 膠芽腫
HNSC (Head and Neck squamous cell carcinoma) 頭頚部がん
KICH (Kidney Chromophobe)
KIRC (Kidney renal clear cell carcinoma)
KIRP (Kidney renal papillary cell carcinoma)
LAML (Acute Myeloid Leukemia) 急性骨髄性白血病
LGG (Lower Grade Glioma) 神経膠腫
LIHC (Liver hepatocellular carcinoma) 肝臓がん
LUAD (Lung adenocarcinoma) 肺腺がん
LUSC (Lung squamous cell carcinoma) 肺扁平上皮がん
MESO (Mesothelioma) 中皮腫
OV (Ovarian serous cystadenocarcinoma) 卵巣漿液性のう胞腺腫
PAAD (Pancreatic adenocarcinoma) 膵臓がん
PCPG (Pheochromocytoma and Paraganglioma) 褐色細胞腫 傍神経節腫
PRAD (Prostate adenocarcinoma) 前立腺がん
SARC (Sarcoma) 肉腫
SKCM (Skin Cutaneous Melanoma) 悪性黒色腫
STAD (Stomach adenocarcinoma) 胃がん
TGCT (Testicular Germ Cell Tumors) 精巣がん
THCA (Thyroid carcinoma) 甲状腺がん
THYM (Thymoma) 胸腺腫
UCEC (Uterine Corpus Endometrial Carcinoma) 子宮体がん
UCS (Uterine Carcinosarcoma) 子宮肉腫
UVM (Uveal Melanoma) ぶどう膜黒色腫
メチル化
これは画像を拡大する意味があまりないかなと思うので、小さい画像でご覧ください。単に綺麗であるという理由で色違い3種類を用意しています。そこまで意味はないので、タペストリー的に見てください。上はクラスタリングなし、下はごく適当にクラスタリングをかけています。中には精巣がんのようにくっきりと層別化されているように見えるものもあります。厳密にはあまり意味のない画像かもしれませんが、それぞれのstudyがどれぐらい層別化できそうかの雰囲気ぐらいはつかめると思います。
# frozen_string_literal: true
require 'fileutils'
require 'gr/plot'
require 'arrow'
require 'progress_bar/core_ext/enumerable_with_progress'
FileUtils.mkdir_p 'methylation'
def read_tsv(path)
Arrow::Table.load(path,
format: 'csv',
delimiter: "\t".ord)
end
require 'numo/linalg'
require 'rumale'
def cluster(z, a, b)
samples = Numo::DFloat.cast(z)
normalizer = Rumale::Preprocessing::StandardScaler.new
samples_std = normalizer.fit_transform(samples.transpose)
samples_std = samples_std.transpose
if a == 1
xindex = true
else
gmm = Rumale::Clustering::KMeans.new(n_clusters: a)
pca = Rumale::Decomposition::PCA.new(n_components: 50)
samples_std_pca = pca.fit_transform(samples_std)
cluster_ids = gmm.fit_predict(samples_std_pca)
xindex = cluster_ids.sort_index
end
if b == 1
yindex = true
else
gmm2 = Rumale::Clustering::KMeans.new(n_clusters: b)
pca2 = Rumale::Decomposition::PCA.new(n_components: 50)
samples_std_pca2 = pca2.fit_transform(samples_std.transpose)
cluster_ids2 = gmm2.fit_predict(samples_std_pca2)
yindex = cluster_ids2.sort_index
end
z3 = Numo::DFloat.cast(z)
z3 = z3[xindex, yindex]
z3
end
CANCER_TYPES = %w[paad stad kirp laml thym meso uvm hnsc lihc kich chol pcpg blca
coadread ov sarc prad lgg lusc thca esca ucs cesc gbm skcm kirc
tgct brca ucec dlbc acc luad].freeze
CANCER_TYPES.each_with_progress do |cancer_type|
path = '../datahub/public/' + cancer_type + '_tcga/data_methylation_hm450.txt'
next unless File.exist? path
df = read_tsv(path)
p cancer_type: cancer_type, n_rows: df.n_rows, time: Time.now
next if cancer_type == 'brca'
z_ = df.select_columns(2..-1).raw_records
z0 = z_.map { |i| i.map { |j| j == 'NA' ? 0.0 : j.to_f } }
z1 = cluster(z0, 1, 6)
z2 = cluster(z0, 6, 6)
[z0, z1, z2].each_with_index do |z, i|
GR.heatmap(z, size: [120, 100], title: cancer_type)
GR.savefig('methylation/' + cancer_type + "_#{i}" + '_heatmap0.png')
GR.heatmap(z, size: [120, 100], title: cancer_type, colormap: 4)
GR.savefig('methylation/' + cancer_type + "_#{i}" + '_heatmap1.png')
GR.heatmap(z, size: [120, 100], title: cancer_type, colormap: 6)
GR.savefig('methylation/' + cancer_type + "_#{i}" + '_heatmap2.png')
end
end
ACC (Adrenocortical carcinoma) 副腎皮質がん
BLCA (Bladder Urothelial Carcinoma) 膀胱がん・尿路上皮がん
BRCA (Breast invasive carcinoma) 乳がん
※ なし(メモリ不足表示)
CESC (Cervical squamous cell carcinoma and endocervical adenocarcinoma) 子宮頸がん
CHOL (Cholangiocarcinoma) 胆管がん
COAD READ (Colon adenocarcinoma) (Rectum adenocarcinoma) 大腸がん・直腸がん
DLBC (Lymphoid Neoplasm Diffuse Large B-cell Lymphoma) リンパ腫・びまん性大細胞型B細胞性リンパ腫
ESCA (Esophageal carcinoma) 食道がん
GBM (Glioblastoma multiforme) 膠芽腫
HNSC (Head and Neck squamous cell carcinoma) 頭頚部がん
KICH (Kidney Chromophobe)
KIRC (Kidney renal clear cell carcinoma)
KIRP (Kidney renal papillary cell carcinoma)
LAML (Acute Myeloid Leukemia) 急性骨髄性白血病
LGG (Lower Grade Glioma) 神経膠腫
LIHC (Liver hepatocellular carcinoma) 肝臓がん
LUAD (Lung adenocarcinoma) 肺腺がん
LUSC (Lung squamous cell carcinoma) 肺扁平上皮がん
MESO (Mesothelioma) 中皮腫
OV (Ovarian serous cystadenocarcinoma) 卵巣漿液性のう胞腺腫
PAAD (Pancreatic adenocarcinoma) 膵臓がん
PCPG (Pheochromocytoma and Paraganglioma) 褐色細胞腫 傍神経節腫
PRAD (Prostate adenocarcinoma) 前立腺がん
SARC (Sarcoma) 肉腫
SKCM (Skin Cutaneous Melanoma) 悪性黒色腫
STAD (Stomach adenocarcinoma) 胃がん
TGCT (Testicular Germ Cell Tumors) 精巣がん
THCA (Thyroid carcinoma) 甲状腺がん
THYM (Thymoma) 胸腺腫
UCEC (Uterine Corpus Endometrial Carcinoma) 子宮体がん
UCS (Uterine Carcinosarcoma) 子宮肉腫
UVM (Uveal Melanoma) ぶどう膜黒色腫
おまけ1 機械学習について
みたいなことをみんな考えているんじゃないでしょうか。知らんけど。
新NP問題つよい!
おまけ2 cBioPortalのGoogleSummerOfCode
cBioPortalでは学生さんがコードを書くGoogleSummerOfCodeをやっています。パスウェイのどこが亢進しているのかみたり、SparkやParquetとの連携をしようとしているようです。
画像の出典:https://twitter.com/cbioportal/status/1167531397134897153
おまけ3 cBioPortalはインスタンスの調査をしているようです
下記のツイッター画像にあるように、cBioPortalでは、世界のどこでローカルインスタンスを使っているか、アンケート調査を行っています。日本でcBioPortalのインスタンスを立ち上げたことがある人は、ぜひアンケートに協力してあげてください。7月17日の時点では日本が空白になっています。
この記事は以上です。ふう。
ここまでスクロールしてくれた人がいたらありがとうございました。