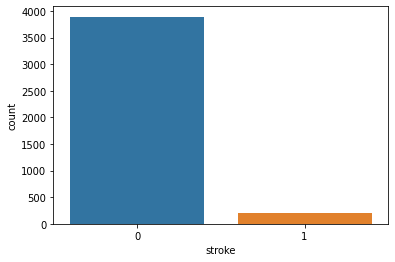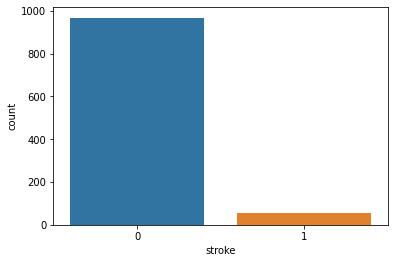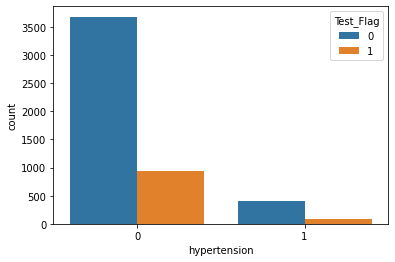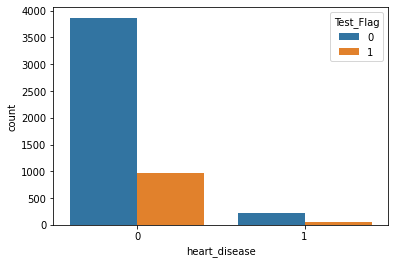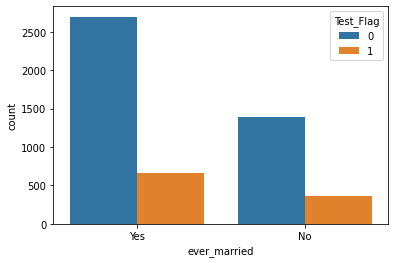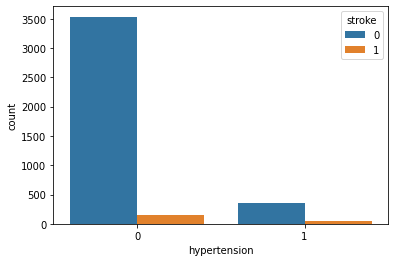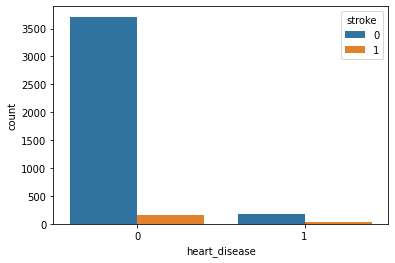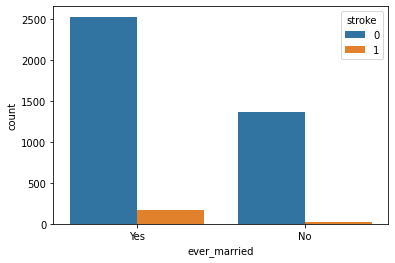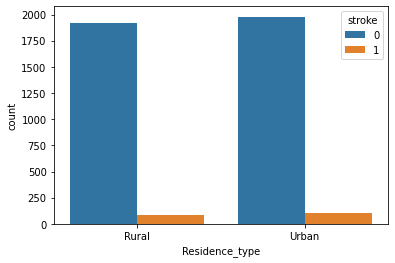目次
- はじめに
- 実行環境
- 利用データ詳細
- 分析
- 結果の解釈
- さいごに
内容
1. はじめに
2022年10月より学習を始めたオンライン学習サービス「Aidemy」の「データ分析講座」における学習の集大成として、自身がこれまでのキャリアで長く関わってきた「ヘルスケア」の分野に焦点を当て、一連のモデル構築、学習、評価の流れを実践した。
2. 実行環境
PC:MacBook Air(M1)
環境:google colaboratory
Python ver : 3.7.0
3. 利用データ詳細
世界保健機関(WHO)によると、脳卒中は世界第2位の死因であり、総死亡者数の約11%を占めていると明らかになっている。
上記データセットの提供は、性別、年齢、高血圧などの合併疾患、喫煙状況などの特徴量に基づいて、患者が脳卒中になる可能性があるかどうかを予測することを、目的とされている。
4. 分析
分析方針の決定
データセットの提供背景を踏まえ、本分析の方針は以下のとおりとした。
- 目的変数を「stroke(脳梗塞)の発症結果」とする。
- 目的変数に相関があると考えられる特徴量を選定する。
- データセットを訓練データとテストデータに分割、訓練データから作成したモデルを元に、テストデータでの脳梗塞の発症を予測する。
- 正解率、再現率を元に精度の高いモデルを構築するための最適なモデル、特徴量を探し出す。
データ読み込みと統計量確認
- BMIのみ欠損値が含まれることを確認。
- 0 or 1の「stroke」が0.0487238なので、目的変数の分布は大きく偏りがある。
- 「age」「avg_glucose_level」「bmi」の最大値が、平均値と比べて大きいので、外れ値が含まれている可能性がある。
# 各ライブラリのインポート
import pandas as pd
import numpy as np
from numpy import nan as NA
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.linear_model import LogisticRegression
from sklearn.svm import LinearSVC
from sklearn.svm import SVC
from sklearn.tree import DecisionTreeClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.neighbors import KNeighborsClassifier
from sklearn.model_selection import KFold
import lightgbm as lgb
from sklearn.utils.class_weight import compute_sample_weight
from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score
from sklearn.model_selection import train_test_split
from sklearn.pipeline import make_pipeline
from sklearn.model_selection import learning_curve
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import confusion_matrix
# データの読み込み
st_df = pd.read_csv("/content/drive/MyDrive/Colab_Notebooks/portfolio/healthcare-dataset-stroke-data.csv")
# データの確認
display(st_df)
# 各カラムのデータ型の確認
print(f'{st_df.dtypes} \n')
# 統計量の確認
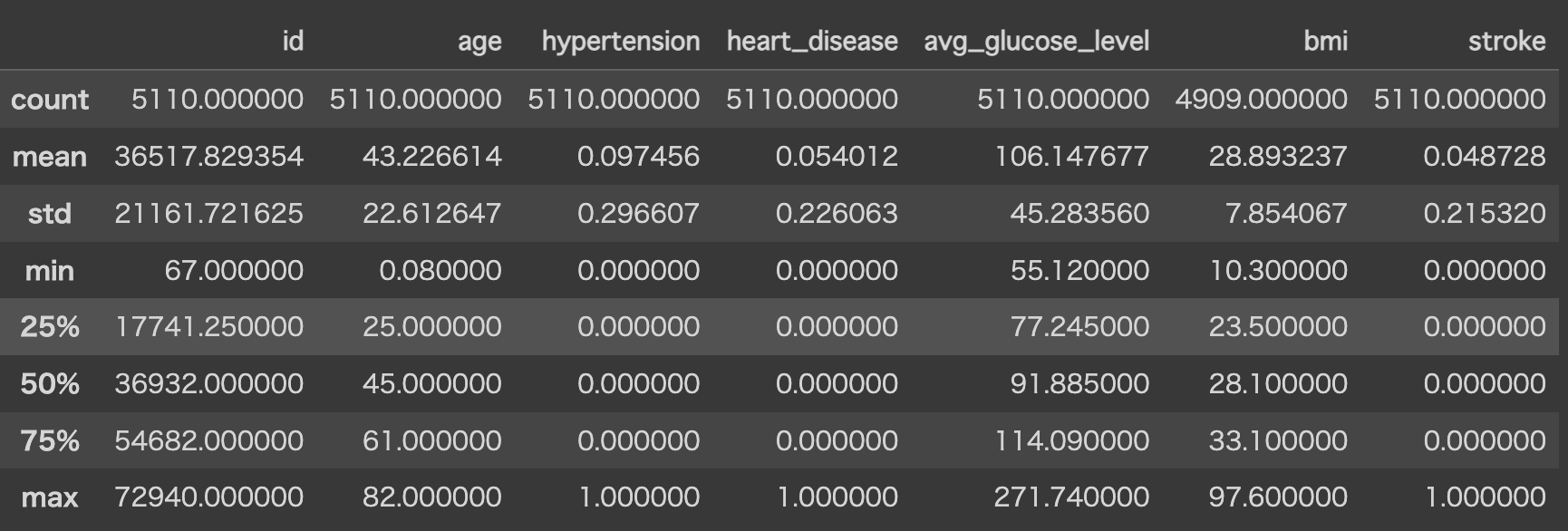
# 数値データ
display(st_df.describe())
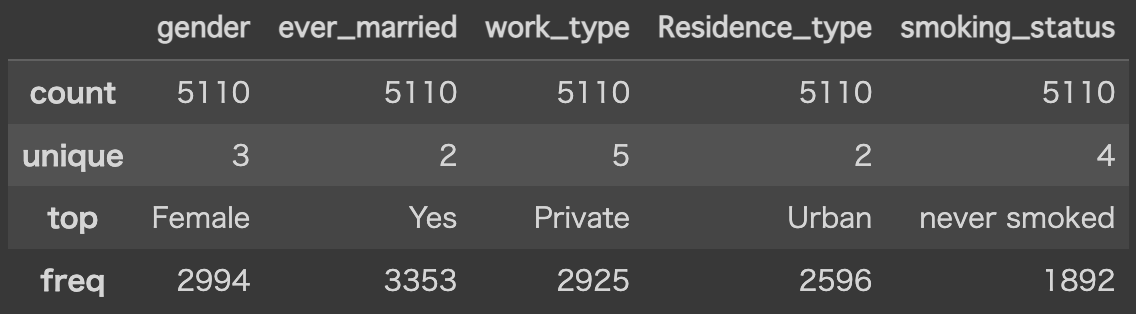
# カテゴリカルデータ
display(st_df.describe(exclude = "number"))
データ分布の確認
- 最初に、読み込んだデータを訓練データとテストデータに分割。
# 訓練データとテストデータの準備
st_df_x = st_df.drop("stroke", axis = 1)
st_df_y = st_df["stroke"]
train_X, test_X, train_y, test_y = train_test_split(
st_df_x, st_df_y, test_size = 0.2, random_state = 0)
st_df_train = pd.concat([train_X, train_y], axis = 1)
st_df_test = pd.concat([test_X, test_y], axis = 1)
- 目的変数(stroke)の分布について、訓練データとテストデータでそれぞれ確認する。
# 訓練データ
sns.countplot(x = "stroke", data = st_df_train)
# テストデータ
sns.countplot(x = "stroke", data = st_df_test)
# 訓練データとテストデータを再度結合し、Test_Flagで区別する
st_df_all = pd.concat([st_df_train, st_df_test], axis = 0).reset_index(drop = True)
st_df_all["Test_Flag"] = 0
st_df_all.loc[st_df_train.shape[0]:, "Test_Flag"] = 1
■訓練データ・テストデータの各特徴量の分布を確認
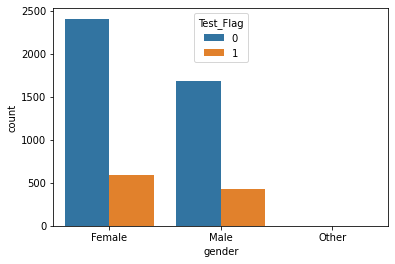
- gender
- 分布に大きな違いはなし
sns.countplot(x = "gender", hue = "Test_Flag", data = st_df_all)
- hypertension
- 分布に大きな違いはなし。
sns.countplot(x = "hypertension", hue = "Test_Flag", data = st_df_all)
- heart_disease
- 分布に大きな違いはなし。
ns.countplot(x = "heart_disease", hue = "Test_Flag", data = st_df_all)
- ever_married
- 分布に大きな違いはなし。
sns.countplot(x = "ever_married", hue = "Test_Flag", data = st_df_all)
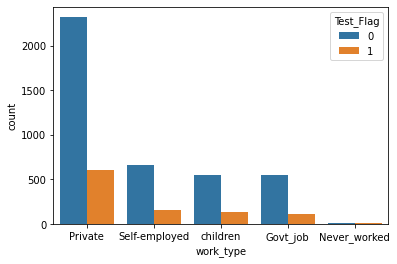
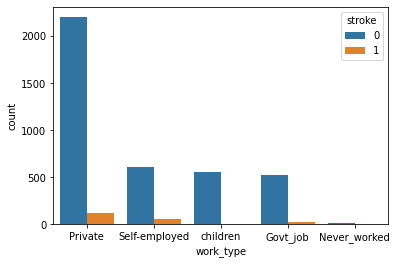
- work_type
- 分布に大きな違いはなし。
sns.countplot(x = "work_type", hue = "Test_Flag", data = st_df_all)
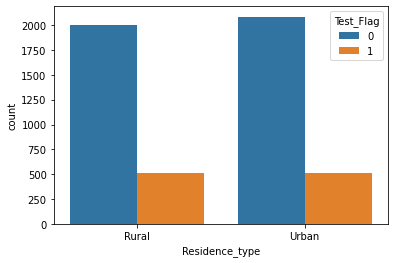
- Residence_type
- 分布に大きな違いはなし。
sns.countplot(x = "Residence_type", hue = "Test_Flag", data = st_df_all)
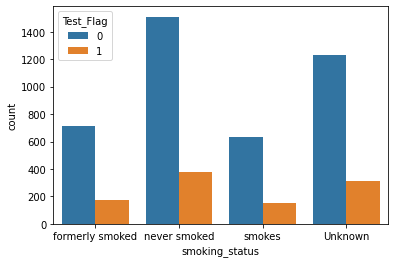
- smoking_status
- 分布に大きな違いはなし。
sns.countplot(x = "smoking_status", hue = "Test_Flag", data = st_df_all)
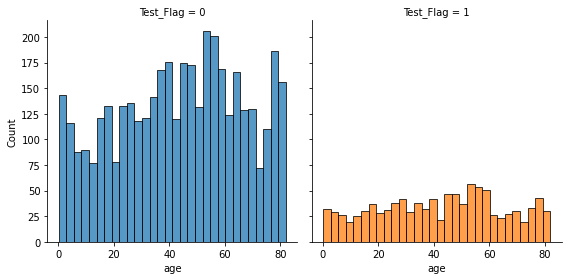
- age
- N数の違いもあり、訓練データとテストデータで分布の形状に差異がある。
fig = sns.FacetGrid(st_df_all, col = "Test_Flag", hue = "Test_Flag", height = 4)
fig.map(sns.histplot, "age", bins = 30, kde = False)
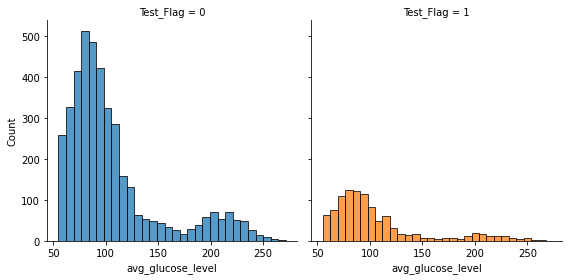
- avg_glucose_level
- N数の違いもあり、訓練データとテストデータで分布の形状に差異がある。
fig = sns.FacetGrid(st_df_all, col = "Test_Flag", hue = "Test_Flag", height = 4)
fig.map(sns.histplot, "avg_glucose_level", bins = 30, kde = False)
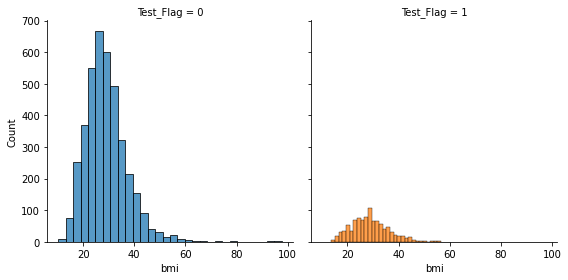
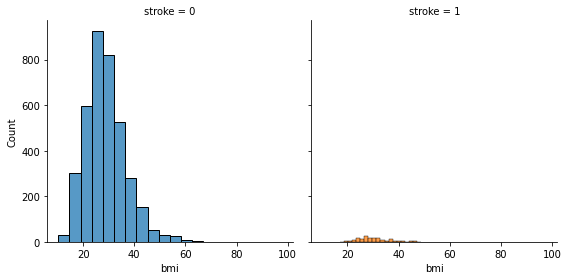
- bmi
- N数の違いもあり、訓練データとテストデータで分布の形状に差異がある
fig = sns.FacetGrid(st_df_all, col = "Test_Flag", hue = "Test_Flag", height = 4)
fig.map(sns.histplot, "bmi", bins = 30, kde = False)
- 分布の特徴まとめ
- 目的変数(stroke)は訓練データ・テストデータと共に、未発症の数が少ない。
- avg_glucose_lecel、bmiには外れ値が存在する。
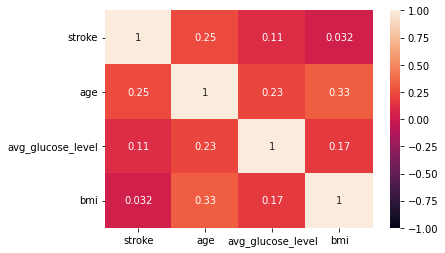
データ相関の確認
- 相関の特徴まとめ
- ageとstroke、 ageとbmiに正の弱い相関が確認された。
# 目的変数、数値データの各特徴量の相関確認
sns.heatmap(st_df_train[['stroke', 'age', 'avg_glucose_level', 'bmi']].corr(),
vmax = 1, vmin = -1, annot = True)
■目的変数と各カテゴリカルデータの相関
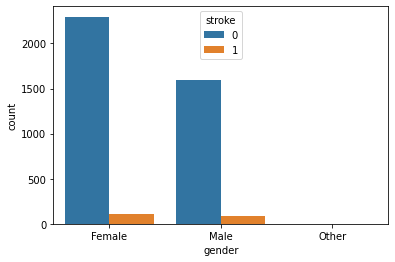
- gender
- stroke:1(発症者)とstroke:0(未発症者)の合計に占める、stroke:1は「male」の方が割合は高い。
sns.countplot(x = "gender", hue = "stroke", data = st_df_train)
- hypertension
- stroke:1(発症者)とstroke:0(未発症者)の合計に占める、stroke:1は「hypertension:1(罹患あり)」の方が割合は高い。
sns.countplot(x = "hypertension", hue = "stroke", data = st_df_train)
- heart_disease
- stroke:1(発症者)とstroke:0(未発症者)の合計に占める、stroke:1は、「heart_disease:1(罹患あり)」の方が割合は高い。
sns.countplot(x = "heart_disease", hue = "stroke", data = st_df_train)
- ever_married
- 各グループにて相関に大きな差異はない。
sns.countplot(x = "ever_married", hue = "stroke", data = st_df_train)
- work_type
- 各グループにて相関に大きな差異はない。
sns.countplot(x = "work_type", hue = "stroke", data = st_df_train)
- Residence_type
- 各グループにて相関に大きな差異はない。
sns.countplot(x = "Residence_type", hue = "stroke", data = st_df_train)
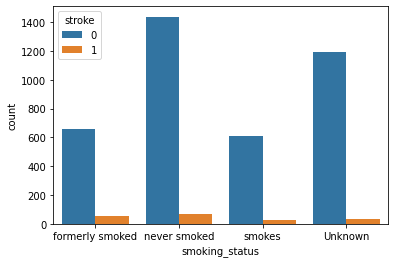
- smoking_status
- stroke:1(発症者)とstroke:0(未発症者)の合計に占める、stroke:1は「never smoked」と比べると「formarly smoked」「smokes」の方が割合は高い。
sns.countplot(x = "smoking_status", hue = "stroke", data = st_df_train)
- カテゴリカルデータの相関の特徴まとめ
- gender,hypertension,heart_disease,smoking_statusにおいては、グループ間での差異が認められた。
■目的変数と各数値データの相関
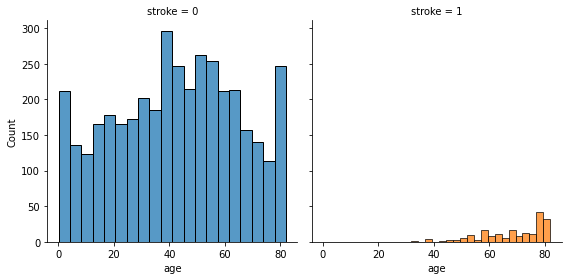
- age
- ageが高くなるにつれて、strokeの発症数が増加する傾向が確認された。
fig = sns.FacetGrid(st_df_train, col = "stroke", hue = "stroke", height = 4)
fig.map(sns.histplot, "age", bins = 20, kde = False)
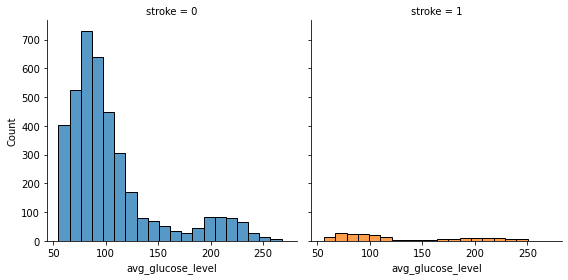
- avg_glucose_level
- 特徴的な傾向は確認されなかった。
fig = sns.FacetGrid(st_df_train, col = "stroke", hue = "stroke", height = 4)
fig.map(sns.histplot, "avg_glucose_level", bins = 20, kde = False)
- bmi
- 特徴的な傾向は確認されなかった。
fig = sns.FacetGrid(st_df_train, col = "stroke", hue = "stroke", height = 4)
fig.map(sns.histplot, "bmi", bins = 20, kde = False)
- 数値データの相関の特徴まとめ
- ageが高くなるにつれて、strokeの発症数が増加する傾向が確認された。
- それ以外の項目については、特徴的な傾向を確認されなかった。
データクレンジング
# bmiに欠損がある行の削除
st_df_all = st_df_all.dropna()
# インデックスを振り直す
st_df_all = st_df_all.reset_index(drop = True)
# 外れ値の対策として、4つの区間に分類したカテゴリ変数に変換
# avg_glucose_level
st_df_all["avg_glucose_level_band"] = pd.qcut(st_df_all["avg_glucose_level"], 4)
# bmi
st_df_all["bmi_band"] = pd.qcut(st_df_all["bmi"], 4)
# カテゴリカル変数の変換
st_df_all = pd.get_dummies(st_df_all, columns = ['gender', 'hypertension', 'heart_disease',
'smoking_status', 'avg_glucose_level_band', 'bmi_band'])
# ageをfloat型からint型に変更
st_df_all = st_df_all.astype({"age": "int64"})
display(st_df_all)
print(st_df_all.columns)
- クレンジングの結果として、以下のindexリストに記載のカラムを持つ、データフレームを作成した。
Index(['id', 'age', 'ever_married', 'work_type', 'Residence_type',
'avg_glucose_level', 'bmi', 'stroke', 'Test_Flag', 'gender_Female',
'gender_Male', 'gender_Other', 'hypertension_0', 'hypertension_1',
'heart_disease_0', 'heart_disease_1', 'smoking_status_Unknown',
'smoking_status_formerly smoked', 'smoking_status_never smoked',
'smoking_status_smokes', 'avg_glucose_level_band_(55.119, 77.07]',
'avg_glucose_level_band_(77.07, 91.68]',
'avg_glucose_level_band_(91.68, 113.57]',
'avg_glucose_level_band_(113.57, 271.74]',
'bmi_band_(10.299000000000001, 23.5]', 'bmi_band_(23.5, 28.1]',
'bmi_band_(28.1, 33.1]', 'bmi_band_(33.1, 97.6]'],
dtype='object')
学習用データの作成
-
利用した特徴量の選定理由
- age:特徴量の増加とともに、strokeの発症数が増加する傾向が確認されたため。
- gender:グループの違いに、相関が認められたため。
- hypertension:グループの違いに、相関が認められたため。
- heart_disease:グループの違いに、相関が認められたため。
- avg_glucose_level_band:学習の結果、特徴量に含んでいるほうが精度が高くなったため。※比較結果は省略
- bmi_band:学習の結果、特徴量に含んでいるほうが精度が高くなったため。※比較結果は省略
-
利用しなかった特徴量の不選定理由
- id:特徴量ではないため。
- bmi:カテゴリカル化し、ダミー変数に変換したため。
- stroke:目的変数であるため。
- avg_glucose_level:カテゴリカル化し、ダミー変数に変換したため。
- Test_Flag:特徴量ではないため。
- ever_married: 相関より傾向の違いが確認できなかったため。
- Residence_type:相関より傾向の違いが確認できなかったため。
- smoking_status:喫煙率は不明のグループにも一定の発症者がおり、不確かな情報が含まれているため。
# 訓練データの一部を検証データとし、予測性能を評価する
# st_df_allを訓練データとテストデータに分割
train = st_df_all[st_df_all["Test_Flag"] == 0]
test = st_df_all[st_df_all["Test_Flag"] == 1].reset_index(drop = True)
# 訓練データのSurvivedをtargetにする
target = train["stroke"]
# 学習に用いないカラムを削除
drop_col = [
"id", "stroke", "avg_glucose_level", "bmi", "Test_Flag",
'ever_married',
'work_type',
'Residence_type',
'smoking_status_Unknown',
'smoking_status_formerly smoked',
'smoking_status_never smoked',
'smoking_status_smokes'
]
train = train.drop(drop_col, axis = 1)
test = test.drop(drop_col, axis = 1)
# 訓練データの一部を分割し検証データを作成
X_train, X_val, y_train, y_val = train_test_split(
train, target, test_size = 0.2, shuffle = True, random_state = 0)
モデル構築と学習実施
-
モデルの採用としては、教師あり学習の分類において選定されるものは一通り採用し、それぞれモデル構築と学習を進めた。
-
結果として、以下の流れとなった。
①ロジスティック回帰、線形SVM、非線形SVM、決定木、ランダムフォレストを、最低限のハイパーパラメータで実施
→ランダムフォレストが最も精度(f値)が高そうと判断
②ランダムフォレストにおける、訓練データと検証データでの精度の乖離を抑制するため、ハイパーパラメータ(max_depth)を調整
③ランダムフォレストの結果を踏まえ、LinghtGBMでより高い精度が出るかを追加検証
- 今回のモデル作成においては、以下2つの問題が発生したため、それぞれハイパーパラメータを調整し、解消を試みた。
●なまけ学習:学習結果として、目的変数がすべてネガティブ(stroke=0)と予測された。
- 目的変数のポジティブ(stroke = 1)が全体のうちの4%という、不均衡データであった。
- それぞれのモデルのハイパーパラメータに`class_weight='balanced'`を採用することで、解消された。
●過学習:訓練データと評価データの精度が大きく乖離した。
- ランダムフォレストにおいて、`max_depth`が設定されてなかった。
- ハイパーパラメータに組み込むことで、過学習が解消された。
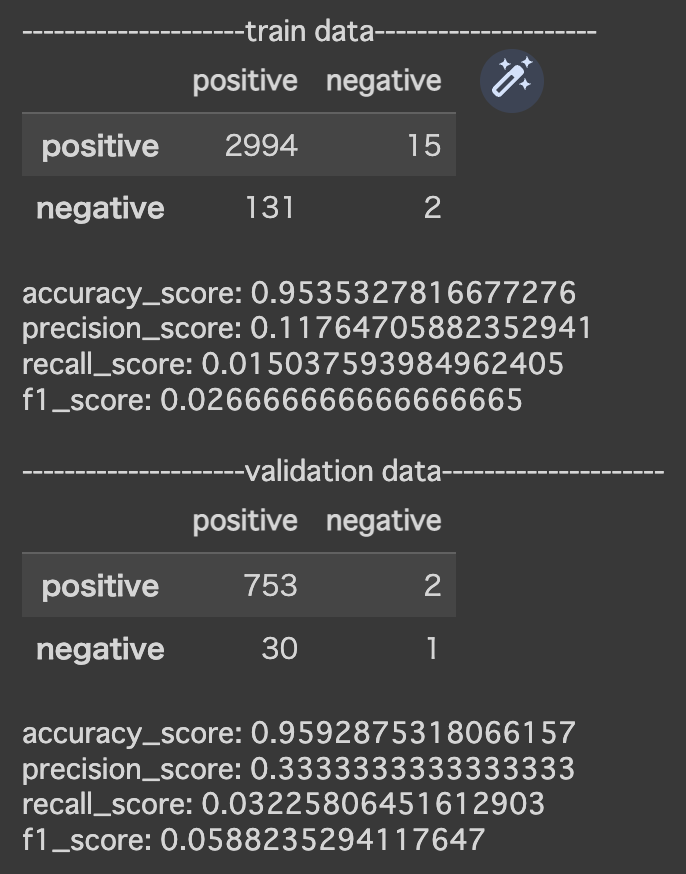
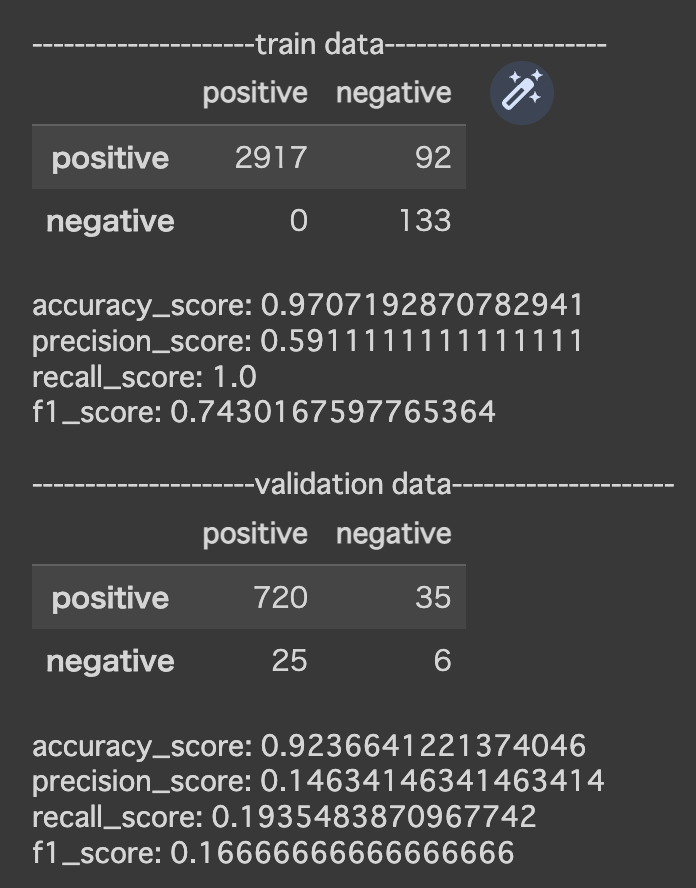
■ロジスティック回帰
# モデルを定義し、学習
model = LogisticRegression(penalty='l2', class_weight='balanced', random_state=0, max_iter=10000)
model.fit(X_train, y_train)
# 訓練データに対しての予測を行う
y_pred = model.predict(X_train)
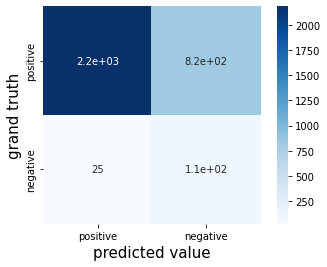
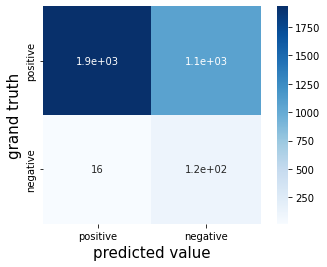
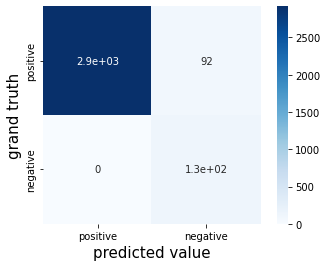
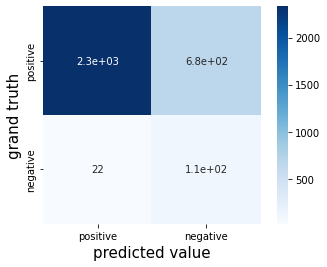
# 混同行列を作成する
cm = confusion_matrix(y_train, y_pred)
names = ["positive", "negative"]
cm = pd.DataFrame(data=cm, index=names, columns=names)
sns.heatmap(cm, square=True, cbar=True, annot=True, cmap='Blues')
plt.xlabel('predicted value', fontsize=15)
plt.ylabel('grand truth', fontsize=15)
plt.show()
print()
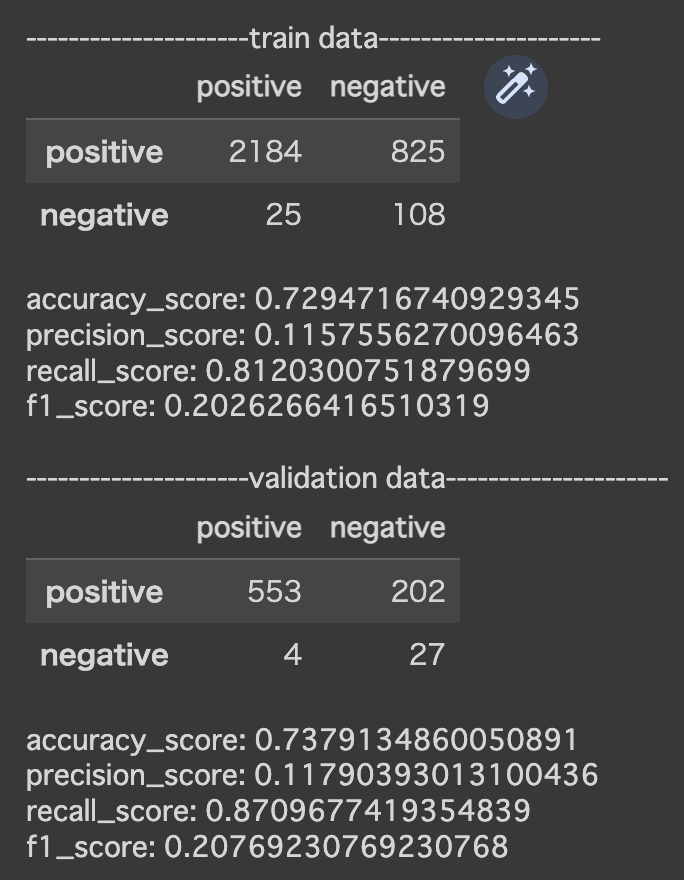
print("---------------------train data---------------------")
display(cm)
print()
# 正答率、適合率、再現率を算出
print("accuracy_score: {}".format(accuracy_score(y_train, y_pred)))
print("precision_score: {}".format(precision_score(y_train, y_pred)))
print("recall_score: {}".format(recall_score(y_train, y_pred)))
print("f1_score: {}".format(f1_score(y_train, y_pred)))
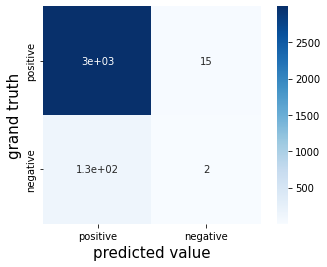
# 評価データに対しての予測を行う
y_pred_val = model.predict(X_val)
# 混同行列を作成する
cm = confusion_matrix(y_val, y_pred_val)
cm = pd.DataFrame(data=cm, index=names, columns=names)
print()
print("---------------------validation data---------------------")
display(cm)
print()
# 評価データの正答率、適合率、再現率を算出
print("accuracy_score: {}".format(accuracy_score(y_val, y_pred_val)))
print("precision_score: {}".format(precision_score(y_val, y_pred_val)))
print("recall_score: {}".format(recall_score(y_val, y_pred_val)))
print("f1_score: {}".format(f1_score(y_val, y_pred_val)))
※以降のモデルでは、モデル構築以降のコードは「ロジステック回帰」でのコードと同様であるため省略
■線形SVM
# モデルを定義し、学習
model = LinearSVC(penalty='l2', class_weight='balanced', random_state=0, max_iter=10000)
model.fit(X_train, y_train)
■非線形SVM
# モデルを定義し、学習
model = SVC(class_weight='balanced', random_state=0, max_iter=10000)
model.fit(X_train, y_train)
■決定木
# モデルを定義し、学習
model = DecisionTreeClassifier(class_weight='balanced', random_state=0)
model.fit(X_train, y_train)
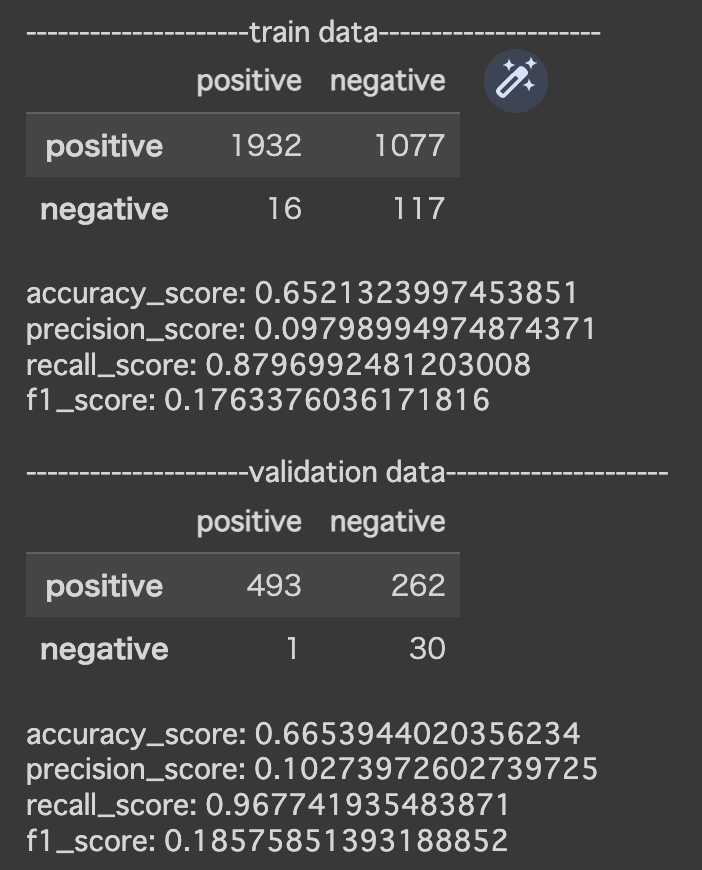
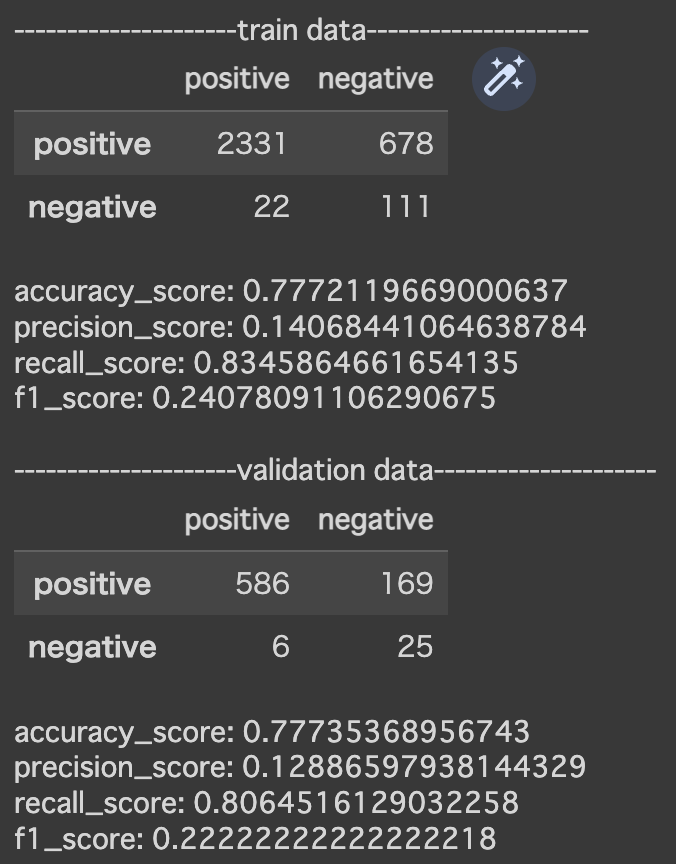
■ランダムフォレスト
# モデルを定義し、学習
model = RandomForestClassifier(class_weight='balanced', random_state=0, max_depth = 5)
model.fit(X_train, y_train)
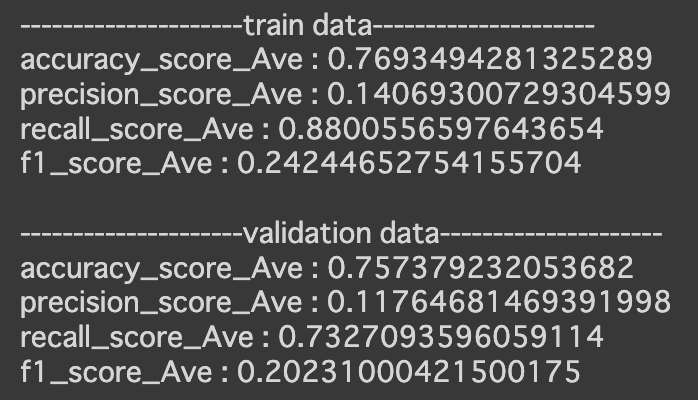
■Light GBM
# モデルを定義し、学習
# KFoldの条件を設定
cv = KFold(n_splits = 3, random_state = 0, shuffle = True)
# 学習結果を格納するリストの作成
train_acc_list = []
train_pre_list = []
train_rec_list = []
train_f1_list = []
val_acc_list = []
val_pre_list = []
val_rec_list = []
val_f1_list = []
models = []
# ハイパーパラメータを定義
lgb_params = {
"objective":"binary",
"metric": "binary_error",
"force_row_wise" : True,
"seed" : 0,
'learning_rate': 0.1,
'min_data_in_leaf': 5,
'num_leaves': 8
}
# LightGBMがカラム名に[]や{}を含んでいるとエラーが出るので、変更する
rename_dict = {
'avg_glucose_level_band_(55.119, 77.07]': "avg_glucose_level_band_1",
'avg_glucose_level_band_(77.07, 91.68]': "avg_glucose_level_band_2",
'avg_glucose_level_band_(91.68, 113.57]': "avg_glucose_level_band_3",
'avg_glucose_level_band_(113.57, 271.74]': "avg_glucose_level_band_4",
'bmi_band_(10.299000000000001, 23.5]': "avg_glucose_level_band_1",
'bmi_band_(23.5, 28.1]': "avg_glucose_level_band_2",
'bmi_band_(28.1, 33.1]': "avg_glucose_level_band_3",
'bmi_band_(33.1, 97.6]': "avg_glucose_level_band_4",
}
# KFoldでFoldごとに学習させる
for i, (trn_index, val_index) in enumerate(cv.split(train, target)):
print(f'Fold : {i}')
X_train, X_val = train.loc[trn_index].rename(columns = rename_dict), train.loc[val_index].rename(columns = rename_dict)
y_train, y_val = target[trn_index], target[val_index]
# LigthGBM用のデータセットを定義、不均衡データのため重み付け
# https://qiita.com/Dixhom/items/fd78f461d038798a7e2e
train_weight = compute_sample_weight(class_weight='balanced', y=y_train).astype('float32')
lgb_train = lgb.Dataset(X_train, y_train, weight = train_weight)
lgb_valid = lgb.Dataset(X_val, y_val)
model = lgb.train(
params = lgb_params,
train_set = lgb_train,
valid_sets = [lgb_train, lgb_valid],
verbose_eval = 100 ,
early_stopping_rounds=10,
)
# 訓練データでモデルを学習
y_pred = model.predict(X_train)
# 訓練データの正解率を算出
train_acc = accuracy_score(
y_train, np.where(y_pred>=0.5, 1, 0)
)
print(train_acc)
train_acc_list.append(train_acc)
# 訓練データの適合率を算出
train_pre = precision_score(
y_train, np.where(y_pred>=0.5, 1, 0)
)
print(train_pre)
train_pre_list.append(train_pre)
# 訓練データの再現率を算出
train_rec = recall_score(
y_train, np.where(y_pred>=0.5, 1, 0)
)
print(train_rec)
train_rec_list.append(train_rec)
# 訓練データのf値を算出
train_f1 = f1_score(
y_train, np.where(y_pred>=0.5, 1, 0)
)
print(train_f1)
print()
train_f1_list.append(train_f1)
# 評価データでモデルを学習
y_pred_val = model.predict(X_val)
# 評価データの正解率を算出
val_acc = accuracy_score(
y_val, np.where(y_pred_val>=0.5, 1, 0)
)
print(val_acc)
val_acc_list.append(val_acc)
# 評価データの適合率を算出
val_pre = precision_score(
y_val, np.where(y_pred_val>=0.5, 1, 0)
)
print(val_pre)
val_pre_list.append(val_pre)
# 評価データの再現率を算出
val_rec = recall_score(
y_val, np.where(y_pred_val>=0.5, 1, 0)
)
print(val_rec)
val_rec_list.append(val_rec)
# 訓練データのf値を算出
val_f1 = f1_score(
y_val, np.where(y_pred_val>=0.5, 1, 0)
)
print(val_f1)
val_f1_list.append(val_f1)
models.append(model)
print()
print("---------------------train data---------------------")
print(f'accuracy_score_Ave : {np.mean(train_acc_list)}')
print(f'precision_score_Ave : {np.mean(train_pre_list)}')
print(f'recall_score_Ave : {np.mean(train_rec_list)}')
print(f'f1_score_Ave : {np.mean(train_f1_list)}')
print()
print("---------------------validation data---------------------")
print(f'accuracy_score_Ave : {np.mean(val_acc_list)}')
print(f'precision_score_Ave : {np.mean(val_pre_list)}')
print(f'recall_score_Ave : {np.mean(val_rec_list)}')
print(f'f1_score_Ave : {np.mean(val_f1_list)}')
5. 結果の解釈
今回は、最終的に「ランダムフォレスト」と「Light GBM」の検証結果の比較を行った。
■訓練データ
| 利用モデル | ランダムフォレスト | LightGBM |
|---|---|---|
| accuraccy | 0.78 | 0.77 |
| precision | 0.14 | 0.14 |
| recall | 0.83 | 0.88 |
| f1 | 0.24 | 0.24 |
■検証データ
| 利用モデル | ランダムフォレスト | LightGBM |
|---|---|---|
| accuraccy | 0.78 | 0.76 |
| precision | 0.13 | 0.12 |
| recall | 0.81 | 0.73 |
| f1 | 0.22 | 0.20 |
今回の目的である、「脳梗塞の発症予測」においては、「取りこぼし」をなるべく少なくすることが重要であり、「正答率」と共に「再現率」も重要であると考えられる。
その観点より、2つのモデルの結果を比較すると、以下の比較結果となった。
- 正答率:訓練データ、検証データともに同等
- 再現率:訓練データでは「ランダムフォレスト」のほうが精度は高く、検証データではLightGBMの方が精度が高い
以上の結果より、今回作成した2つのモデルの精度は、概ね同等であると判断した。
6. さいごに
機械学習において最適なモデルを構築するには、①特徴量の選定②モデルの選定の2点が必要であることを学ぶことが出来た。
「こういうときにはこれを使おう」「これがだめなら、次はこうしよう」という判断がスムーズにできるよう、自分の中にある引き出しを増やしていくことの大切さを実感した。
今回の学びをスタートに、自身としてのデータ人材としての価値を向上できるよう、さらなる自己研鑽に励みたい。