はじめに
RDKitで反応パターンをSMARTSで定義し反応物を生成するでは、炭素原子の水酸化反応を例にSMARTSによる反応をRDKitでやってみた。しかし、全ての炭素原子を水酸化させたいケースは現実的ではない。そこで今回はより細かい条件をSAMRTSで指定してみた。具体的には、鎖の末端のメチル基を指定し、そこだけ水酸化してみた。
方法
前回、以下のようなSMARTSを書いた。
[C:1]>>[C:1][OH]
しかしこれでは任意の炭素原子に反応してしまう。今回は鎖の末端のメチル基のみ酸化させたい。末端のメチル基は、水素原子が3つついているはずだから、以下のようにルールを変えてみる。
'[CH3:1]>>[CH2:1]-[OH]'
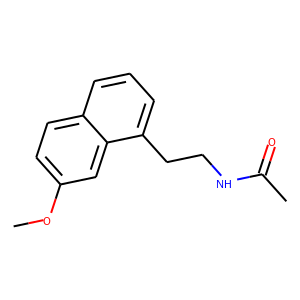
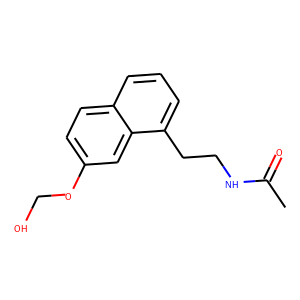
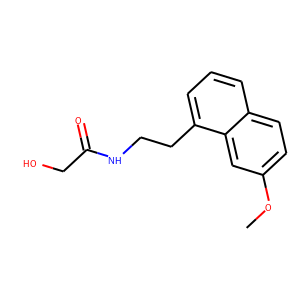
やってみよう。今回テストするのは前回とおなじ化合物である。前回は4つ反応物が生成されたが、今回は2つある末端のメチル基のみの反応物が生成されるはずだ。
やってみると生成された反応物は2つ。見てみよう。
2つ目。もう一つの末端のメチル基が反応している。予想通りである。
ちなみに、鎖の中間にある水素原子が2つ結合してる2つの炭素原子のみを反応させるSMARTSは以下の通りとなる。
'[CH2:1]>>[CH1:1]-[OH]'
ソース
最後に画像生成も含むソースの全体を掲載する。
from rdkit.Chem import AllChem
from rdkit.Chem.Draw import rdMolDraw2D
from io import BytesIO
from cairosvg import svg2png
from IPython.display import SVG
from rdkit import Chem
# 参考(rdkit-smarts)https://magattaca.hatenablog.com/entry/2019/02/10/194853
# 参考(smarts) https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5515729/table/Tab1/?report=objectonly
# https://sourceforge.net/p/rdkit/mailman/message/36294482/
def generate_image(mol, size, path, name):
image_data = BytesIO()
view = rdMolDraw2D.MolDraw2DSVG(size[0], size[1])
tm = rdMolDraw2D.PrepareMolForDrawing(mol)
view.DrawMolecule(tm)
view.FinishDrawing()
svg = view.GetDrawingText()
SVG(svg.replace('svg:', ''))
print(path + "/" + name)
try:
svg2png(bytestring=svg, write_to=path + "/" + name + ".png")
except:
print("errro")
pass
def main():
reactant_1 = Chem.MolFromSmiles('COc2ccc1cccc(CCNC(C)=O)c1c2')
generate_image(reactant_1, (300, 300), "./tmp", "reactant")
#reaction_pattern = '[CH3:1]>>[CH2:1]-[OH]'
reaction_pattern = '[CH3:1]>>[CH2:1]-[OH]'
rxn = AllChem.ReactionFromSmarts(reaction_pattern)
x = rxn.RunReactants([reactant_1])
for i, mol in enumerate(x):
print(mol[0])
try:
Chem.SanitizeMol(mol[0])
generate_image(mol[0], (300, 300), "./tmp", "metablite_{0}".format(i))
except:
print("Error")
print(Chem.MolToMolBlock(mol[0]))
if __name__ == "__main__":
main()
おわりに
- 厳密には電荷等も考慮しないといけないかもしれない。化学的にはシロートなのでよくわからず。
- 今後の課題として反応部位が分かっている時に、その部位のみ反応させてみたい。また、SMARTSとは関係ないかもしれないが、反応のされやすさを評価してみたい。
- 応用として、SMARTSで反応テンプレート群を定義し、そこから生成された反応物を使って、https://www.frontiersin.org/articles/10.3389/fphar.2019.01586/full
のようなDeepLearningによる反応予測してみても面白そうではある。